Selenoproteïnes en Eptesicus fuscus
Cebrián M.C, Franco M, Gallardo P, Gallego E.
| ABSTRACT | INTRODUCCIÓ | MATERIALS I MÈTODES | RESULTATS | DISCUSSIÓ | CONCLUSIONS | REFERÈNCIES | AGRAÏMENTS | CONTACTA'NS |
INTRODUCCIÓ
Eptesicus fuscus és un ratpenat de l’orde dels quiròters i de la familia Vespertilionidae. La família més propera és la Molossidae, seguida de Natalidae, que totes tres formen la superfamilia Vespertillonoidea.
Segons un estudi fet per Mario Mariotti et al. sobre la composició i evolució de selenoprotemes de vertebrats i mamífers, el genoma d'E.fuscus hauria de tenir aproximadament 25 selenoproteïnes, ja que tant el Microbat (com per exemple Myotis lucifugus) com el Macrobat (com per exemple Pteropus vampyrus), que són ratpenats on s'han estudiat les selenoproteïnes, presentaven aquesta quantitat. Aquestes selenoproteïnes són les següents: Sel15, GPx1, GPx2, GPx3, GPx4, GPx6, DI1, DI2, DI3, SelR, SPS2b, SelH, SelI, SelK, SelM, SelN, SelO, SelP, SelS, SelT1a, SelV, SelW, TR1, TR2, TR3.
A continuació explicarem una per una totes les selenoproteïnes trobades, homòlogues de cisteïna i les proteïnes de la maquinària de síntesi.
SELENOPROTEÏNES I HOMÒLEGS DE CISTEÏNA
Família DI
Existeixen tres enzims DI (Iodothyronine deiodinase) en mamífers i tots ells contenen l’enzim selenocisteïna: Di1, Di2 i Di3. Es van obtenir diferents hits i utilitzant els valors d’E-value i la informació dels bits del BLAST vam poder assignar un hit a cada una de les proteïnes. Vam veure que es solapaven els scaffolds entre les diferents proteïnes de la família, ja que hi ha homologia intrafamiliar.
Vam fer la filogènia (veure arxiu) d’aquesta família per saber quins eren els hits que havíem d’escollir per a fer el posterior anàlisi.

No vam haver de fer cap T-Coffee per a saber quina query havíem d’escollir dins de SelenoDb, ja que només tenim una en la base de dades.
Sel DI1
DI1 (Iodothyronine deiodinase 1) activa l’hormona tiroidea a partir de la conversió de la pro-hormona tiroxina (T4) per deiodinatzició de l'anell (PRD) de 3,3-prime,5-triiodotironina (T3). DI degrada tant l’hormona tiroidea com la pro-hormona per deiodinització de l'anell (IRD).
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits. Aquí ens trobem amb la proteïna DI1 que es troba al scaffold JH977620.1, a la regió que va de 1022960 a 1003546 (BLAST) i està codificada en el sentit invers (reverse).
La predicció d’Exonerate té 19415 nucleòtids, que inclouen quatre exons i tres introns i un raw score de 1232. Mentre que la predicció de Genewise té 19414 nucleòtids, que inclouen quatre exons i tres introns i un score de 518.13 bits. En aquest cas les prediccions són quasi idèntiques a excepció que l'Exonerate dóna una predicció més llarga per només un nucleòtid de més. Es pot veure que els dos mètodes emprats ens donen uns bons alineaments del T-Coffee corresponent (score de 100).
La proteïna de Myotis lucifugus té una selenocisteïna i hem observat que aquesta es troba conservada tant a l’Exonerate com en el Genewise. Es troba conservada en el segon exó de les proteïnes predites per l’Exonerate i Genewise. El programa SeciSearch ha trobat tres possibles elements Seci a l’extrem 3’ de la cadena complementària del gen.
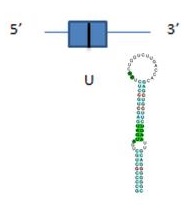
En quant el programa Seblastian, ens prediu un SECIS, amb un grau A i el qual es troba a una distància de 1281 nucleòtids de la selenocisteïna i a 921 del final del gen. Això és possible, ja que el Seblastian és més restrictiu a l'hora de predir els Secis. A més, ens va predir la selenoproteïna de la imatge. Després d’haver analitzat tota la informació anterior, es podria predir una estructura de la proteïna DI1:
EXONERATE I GENEWISE:
Sel DI2
DI1 (Iodothyronine deiodinase 2) activa l’hormona tiroidea a partir de la conversió de la pro-hormona tiroxina (T4) per deiodinatzició de l'anell (PRD) de 3,3-prime,5-triiodotironina (T3). DI degrada tant l’hormona tiroidea com la pro-hormona per deiodinització de l'anell (IRD). Està altament expressada a la tiroide i pot contribuir a l'augment de la producció de T3 en pacients amb la malaltia de Graves i adenomes de la tiroides.
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
Aquí ens trobem amb la proteïna DI2 que es troba al scaffold JH977657.1, que va de la regió 3049587 fins a 3040434 i està codificada en el sentit invers (reverse). La predicció d’Exonerate té 9154 nucleòtids, que inclouen dos exons i un intró i un raw score de 1327 mentre que la predicció de Genewise té 9153 nucleòtids, que inclouen dos exons i un intró amb un score de 595.43 bits. En aquest cas, les prediccions són quasi idèntiques a excepció que l'Exonerate dóna una predicció més llarga per només un nucleòtid de més. Es pot veure que els dos mètodes emprats ens donen uns bons alineaments del T-Coffee corresponent (score de 100).
La proteïna de Myotis lucifugus té una selenocisteïna i hem observat que aquesta es troba conservada tant en el programa Genewise com en Exonerate. La selenocisteïna es troba conservada en el segon exó de la proteïna predita pel Genewise i Exonerate. Es va veure que en humans existeix un segon codó UGA, però aquest no està conservat en Myotis lucifugus. El programa SeciSearch ha trobat dos possibles elements Secis a la cadena complementària del gen. En canvi, el programa Seblastian no ha trobat cap selenoproteïna, però sí que ens ha predit Secis amb SeciSearch3. Podria ser degut a problemes interns del programa o que aquesta selenoproteïna pogués no trobar-se en aquest scaffold, ja que a l’haver fet l’arbre filogenètic s’observa una baixa significança entre el hit obtingut (encara que és el millor segons l’arbre) i la selenoproteïna.
Després d’haver analitzat tota la informació anterior, es podria predir una estructura de la proteïna DI2:
EXONERATE I GENEWISE:
Sel DI3
En humans, DI3 (Iodothyronine deiodinase 3) inactiva l’hormona tiroidea per deiodinització de l’anell intern de la pro-hormona tiroxina (T4) i la hormona bioactiva T3 per inactivar els metàbolis RT3 i T2, respectivament. Aquest enzim s’expressa en l’úter d’embarassades, placenta i teixits fetals i neonatals, de manera que juga un paper essencial en la regulació de la inactivació de l’hormona tiroidea durant el desenvolupament embriològic.
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
Aquí ens trobem amb la proteïna DI3 que es troba al scaffold JH977717.1, que va de la regió 489392 fins a 488595 (BLAST) i està codificada en el sentit invers (reverse).
La predicció d’Exonerate té 834 nucleòtids, que inclou un exó amb un raw score de 1362 mentre que la predicció de Genewise té 833 nucleòtids, que inclou un exó amb un score de 624.40 bits. En aquest cas, les prediccions són quasi idèntiques a excepció que l'Exonerate dóna una predicció més llarga per només un nucleòtid de més. Es pot veure que els dos mètodes emprats, doncs, ens donen uns bons alineaments del T-Coffee corresponent (score de 100).
La proteïna DI3 a Homo sapiens té una selenocisteïna i hem observat que aquesta es troba conservada en Myotis lucifugus tant per Exonerate com per Genewise, la qual es troba a l’únic exó de la proteïna predita. Inicialment, vam pensar que era un pseudogen per retrotransposició, però després vam veure a la literatura que tots els gens Di3 trobats fins a l’actualitat no presenten introns, igual que DI3 en Eptesicus fuscus. El programa SeciSearch ha trobat un possible element Secis a la cadena complementària del gen i el programa Seblastian prediu un element SECI a 578 nucleòtids de la selenoproteïna i a una distància de 980 nucleòtids del element Sec-UGA. També es prediu una selenoproteïna, tal i com es veu a la imatge.
Després d’haver analitzat tota la informació anterior, es podria predir una estructura de la proteïna DI3:
EXONERATE I GENEWISE:
FAMÍLIA GPx
La família de les selenoproteïnes GPx és la més gran en vertebrats. Els mamífers tenen 8 homòlegs, 5 dels quals són selenoproteïnes: GPx1, GPx2, GPx3, GPx4, GPx6. Degut a què és una família de selenoproteïnes amb varis membres, amb un grau d'homologia molt elevat, va resultar complicat decidir quina era la posició exacta en el genoma de cadascuna per separat. Totes les GPx eren en la seva majoria selenoproteïnes però per evolució algunes delles han perdut la selenocisteïna i han passat a ser cisteïnes, convertint-se en homòlegs de cisteïna.
El Blast de cada una d’elles contra Eptesicus fuscus ens ha donat diversos hits significatius (E-value major o igual a 10-5), molts dels quals eren comuns en les diferents proteïnes. Això pot ser degut a una gran similitud entre aquestes proteïnes. Hem escollit el millor hit per a cada proteïna mitjançant l’anàlisi de la identitat, E-value i els resultats dels T-Coffees obtinguts. Tot i això, no hem pogut associar cap hit a la proteïna GPx6, ja que tenia valors molt baixos. Hem pensat que això podria ser degut a què GPx6 no es troba a Myotis lucifugus segons SelenoDB i per això l’hem descartat.
Per escollir les querys hem comparat les seqüències de Myotis lucifugus i Homo sapiens mitjançant alineaments amb T-Coffee. En el cas de les GPx hem vist que la majoria de proteïnes de Myotis no començaven per metionina i en alguns casos les querys eren iguals a les humanes, així que hem escollit les querys humanes perquè estan millor anotades. A les humanes, en alguns casos hi havia diverses seqüències per una proteïna, així que de nou hem fet un T-Coffee per escollir-ne la millor i fer-la servir com a query.
GPx1
La GPx1 (Glutathione peroxidase 1) actua en la detoxificació del peròxid d’hidrogen, i és un dels enzims antioxidants més importants en els humans. S’expressa en el citosol.
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
Segons la nostra predicció el millor hit d’aquesta proteïna es troba a l’scaffold JH977614.1 entre les posicions 39397691- 39398529 a la cadena forward. A SelenoDB, observem que la GPx1 humana anotada que hem fet servir com a query és una selenoproteïna, té 2 exons, amb la selenocisteïna al segon exó. A Myotis lucifugus és un homòleg amb cisteïna.
Analitzant els T-Coffees tant d’Exonerate com de Genewise, observem com el d’Exonerate dóna molt bones correspondències. L’Exonerate d’aquest scaffold dóna 860 nucleòtids, amb 2 exons i 1 intró i un raw score de 920. Per altra banda, el seu Genewise dóna 886 nucleòtids, amb també 2 exons i 1 intró i un score de 396 bits. Per tant, tots dos mètodes prediuen la mateixa proteïna, amb una diferència de 26 nucleòtids. A més, veiem com la selenocisteïna, a diferència de les dades disponibles a SelenoDB, es troba al primer exó de l’scaffold que hem triat.
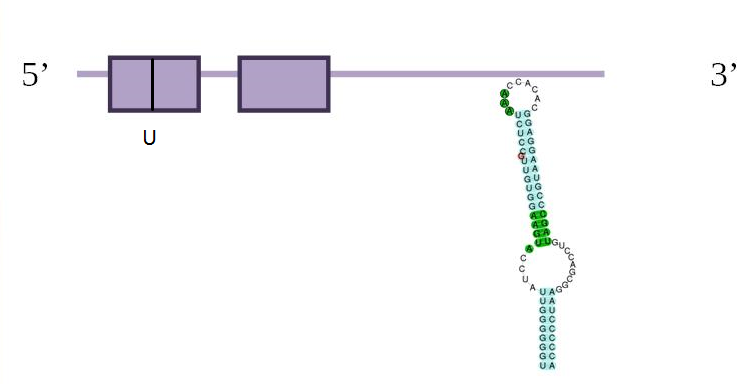
Pel que fa a l’anàlisi dels Secis i el Seblastian veiem com sí que presenta un element Secis, de grau A, amb un bon valor d’energia lliure i situat convenientment a la regió 3’ de la proteïna predita. Per tant, podem predir que aquesta selenoproteïna GPx1 sí es troba al genoma d’Eptesicus fuscus.
GPx2
La GPx2 (Glutathione peroxidase 2) és una glutatió peroxidasa depenent de seleni, i és un dels dos isoenzims responsables per la majoria de l’activitat reductora de peròxid d’hidrogen depenent de glutatió a l’epiteli del tracte gastrointestinal.
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
El millor hit d’aquesta proteïna es troba a l’scaffold JH977619.1 a les posicions 11226640- 11229797 a la cadena forward. A SelenoDB, observem que la GPx2 humana anotada que hem fet servir com a query és una selenoproteïna, té 2 exons, amb la selenocisteïna al segon exó. També és una selenoproteïna a Myotis.
Analitzant els T-Coffees observem com el d’Exonerate dóna molt bones correspondències. L’Exonerate d’aquest scaffold dóna 3158 nucleòtids, amb 2 exons i 1 intró i un raw score de 996. Per altra banda, el seu Genewise dóna 3152 nucleòtids, amb també 2 exons i 1 intró i un score de 436bits. Per tant, tots dos mètodes prediuen la mateixa proteïna, amb una diferència de 6 nucleòtids. A més, veiem com la selenocisteïna, a diferència de les dades disponibles a SelenoDB, es troba al primer exó de l’scaffold que hem triat.
Pel que fa a l’anàlisi dels Secis i el Seblastian veiem com sí que presenta un element Secis, de grau A, amb un bon valor d’energia lliure i situat convenientment a la regió 3’ de la proteïna predita. Per tant, podem predir que aquesta selenoproteïna sí es troba al genoma d’Eptesicus fuscus.
GPx3
La GPx3 (Glutathione peroxidase 3) és una peroxidasa que actua a la detoxificació del peròxid d’hidrogen, sent un dels enzims antioxidants més improtants en els humans. Se secreta en el plasma.
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
El millor hit d’aquesta proteïna es troba a l’scaffold JH977686.1 a les posicions 2081391- 2083978 a la cadena forward. A SelenoDB, observem que la GPx3 humana anotada que hem fet servir com a query té 5 exons, amb la selenocisteïna al segon exó. És una selenoproteïna a humans i a myotis també.
Analitzant els T-Coffees tant d’Exonerate com de Genewise, observem com el d’Exonerate dona molt bones correspondències. L’Exonerate d’aquest scaffold dóna 6924 nucleòtids, amb 5 exons i 4 intró i un raw score de 1037. Per altra banda, el seu Genewise dóna 6923 nucleòtids, amb també 5 exons i 4 intró i un score de 421bits. Per tant, tots dos mètodes prediuen la mateixa proteïna, amb una diferència d’un nucleòtid. A més, veiem com la selenocisteïna, igual que a les dades disponibles SelenoDB, es troba al segon exó de l’scaffold que hem triat.
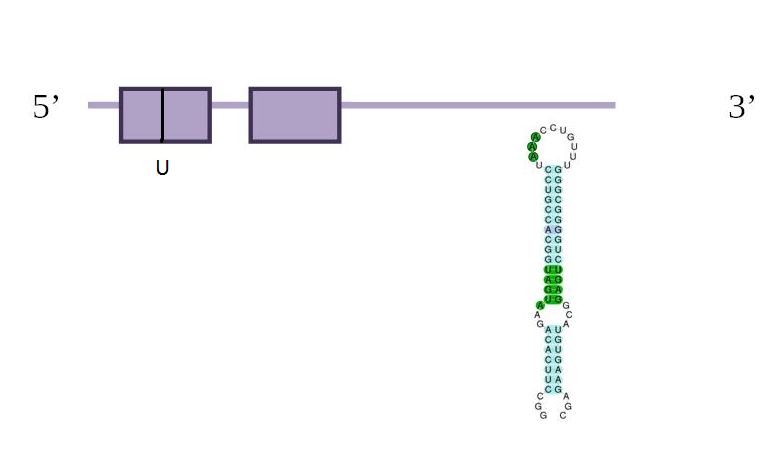
Pel que fa a l’anàlisi dels Secis i el Seblastian veiem com sí que presenta un element Secis, de grau A, amb un bon valor d’energia lliure i situat convenientment a la regió 3’ de la proteïna predita. Per tant podem predir que aquesta selenoproteïna sí es troba al genoma d’Eptesicus fuscus.

La GPx4 (Glutathione peroxidase 4) catalitza la reducció del peròxid d’hirogen, hidroperòxid orgànic i peròxids lípids per la reducció del glutatió, i funciona per la protecció de les cèl·lules front el dany oxidatiu. Està altament expressat en l’esperma.
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
El millor hit d’aquesta proteïna es troba a l’scaffold JH977673.1 entre les posicions 4369148- 4368554 a la cadena reverse. A SelenoDB, observem que la GPx4 humana anotada que hem fet servir com a query té 7 exons, amb la selenocisteïna al tercer exó. És una selenoproteïna a humans i Myotis lucifugus.
Analitzant els T-Coffees tant d’Exonerate com de Genewise, observem com el d’Exonerate dóna molt bones correspondències. L’Exonerate d’aquest scaffold dóna 2160 nucleòtids, amb 7 exons i 6 introns i un raw score de 959. Per altra banda, el seu Genewise dóna 2061 nucleòtids, amb 6 exons i 5 introns i un score de 341 bits. Per tant, tots dos mètodes prediuen la mateixa proteïna, amb una diferència de 99 nucleòtids. A més, veiem com la selenocisteïna, igual que a les dades disponibles SelenoDB, es troba al tercer exó de l’scaffold que hem triat.
Pel que fa a l’anàlisi dels Secis i el Seblastian veiem com sí que presenta un element Secis, de grau A, amb un bon valor d’energia lliure i situat convenientment a la regió 3’ de la proteïna predita. Per tant podem predir que aquesta selenoproteïna sí es troba al genoma d’Eptesicus fuscus.
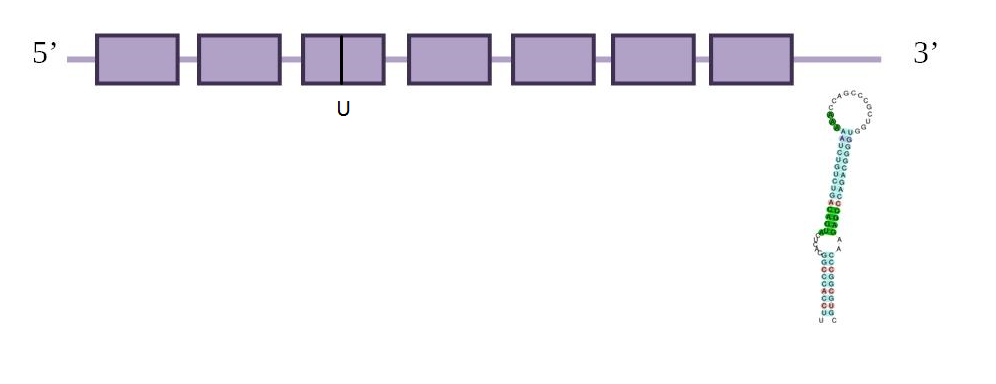
GPx5
GPx5 (Glutathione peroxidase 5) s’expressa de manera específica en l’epidídim del tracte reproductiu masculí dels mamífers, i és regulat pels andrògens. A diferència d’altres mRNAs caracteritzats per altres glutations peroxidases, aquest mRNA no conté cap codó de selenocisteïna (UGA). A més, GPx5 és independent de seleni, i s’ha proposat que té un paper en la protecció de les membranes dels espermatozous front els efectes danyins de la peroxidació dels lípids i/o en la prevenció de la reacció prematura de l’acrosoma.
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
Segons la nostra predicció el millor hit d’aquesta proteïna es troba a l’scaffold JH977676.1 entre les posicions 149093- 136941 a la cadena forward. A SelenoDB, observem que la GPx5 humana anotada que hem fet servir com a query no és una selenoproteïna, és un homòleg de cisteïna. Té 5 exons i 4 introns. A Myotis lucifugus també és un homòleg amb cisteïna. El T-Coffee d’Exonerate no dóna bones correspondències, el T-Coffee Genewise tampoc. L’Exonerate d’aquest scaffold dóna 507 nucleòtids, amb 1 exó i un raw score de 287. Per altra banda, el seu Genewise dóna 467 nucleòtids, amb també 1 exó i un score de 109 bits. Per tant, sembla que tots dos mètodes prediuen la mateixa proteïna, amb una diferència de 40 nucleòtids.
Pel que fa a l’anàlisi del Seblastian veiem com no ens pot predir cap selenoproteïna però sí que presenta un element Secis, de grau A. Els nostres resultats, doncs, demostren la conservació de l'aminoàcid cisteïna i un element SECI. La nostra hipòtesi és que GPx5 inicialment era una selenoproteïna, però ha evolucionat canviant la Selenocisteïna per la cisteïna i encara no s'ha perdut la maquinària pròpia de la selenoproteïna. Després d’analitzar l’arbre filogenètic creiem que no hem seleccionat correctament el hit per aquesta proteïna. Per tant, no podem afirmar que aquesta proteïna es trobi al genoma d’Eptesicus fuscus.
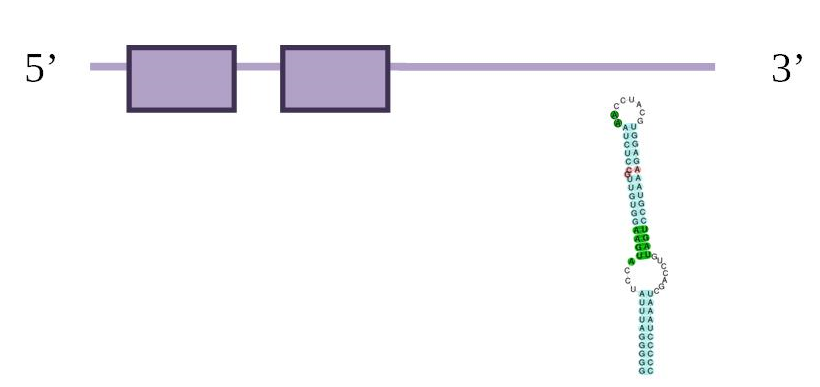
GPx7
La GPx7 (Glutathione peroxidase 7) actua en la detoxificació del peròxid d’hidrogen, i és un dels enzims antioxidants més importants en humans. A diferència d’altres mRNAs de les glutations peroxidases, aquest mRNA no conté el codó selenocisteïna (UGA). A més, la GPx7 és independent del seleni.
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
El millor hit d’aquesta proteïna es troba a l’scaffold JH977620.1 entre les posicions 2096046-2089027 i a la cadena reverse. A SelenoDB, observem que la GPx7 humana anotada que hem fet servir com a query no és una selenoproteïna, té 3 exons. Tampoc és una selenoproteïna a Myotis lucifugus.
Analitzant els T-Coffees tant d’Exonerate com de Genewise, observem com el d’Exonerate dóna bones correspondències. L’Exonerate d’aquest scaffold dóna 7077 nucleòtids, amb 3 exons i 2 intró i un raw score de 897. Per altra banda, el seu Genewise dóna 7076 nucleòtids, amb 3 exons i 2 intró i un score de 399 bits. Per tant, tots dos mètodes prediuen la mateixa proteïna, amb una diferència de 1 nucleòtids.
Pel que fa a l’anàlisi dels Secis i el Seblastian no s’han trobat elements SECIs. Com que no és una selenoproteïna, no detectem cap selenocisteïna, ja que conté cisteïna enlloc de la selenocisteïna. Per tant, amb les dades que tenim no podem predir la selenoproteïna GPx7 en el genoma d'Eptesicus fuscus.
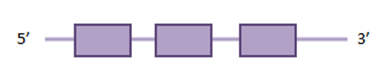
GPx8
El GPx8 (Glutathione peroxidase 8) també actua en la detoxificació del peròxid d’hidrogen, i és un dels enzims antioxidants més importants en humans. A diferència d’altres mRNAs de les glutations peroxidases, aquest mRNA no conté el codó selenocisteïna (UGA). A més, la GPx7 és independent del seleni.
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
El millor hit d’aquesta proteïna es troba a l’scaffold JH977884.1 entre les posicions 452441-455593 a la cadena forward. A SelenoDB, observem que la GPx8 humana anotada que hem fet servir com a query no és una selenoproteïna i té 3 exons. No es troba a Myotis lucifugus.
Analitzant els T-Coffees tant d’Exonerate com de Genewise, observem com el d’Exonerate dóna més o menys bona correspondència L’Exonerate d’aquest scaffold dóna 3153 nucleòtids, amb 3 exons i 2 introns i un raw score de 818. Per altra banda, el seu Genewise dóna 3152 nucleòtids, amb 3 exons i 2 introns i un score de 353 bits. Per tant, tots dos mètodes prediuen la mateixa proteïna, amb una diferència d’1 nucleòtid.
Pel que fa a l’anàlisi dels Secis i el Seblastian no s’han trobat elements SECIs. Com que no és una selenoproteïna sinó un homòleg de cisteïna, no detectem cap selenocisteïna, ja que conté cisteïna enlloc de la selenocisteïna.
Com aquesta proteïna no es troba a Myotis lucifugus i no hem obtingut uns resultats massa clars comparant-la amb humans hem fet un Blastp (veure arxiu).
El resultat del Blastp contra la base de dades de NCBI mostra homologia del 82% amb GPx8 d’altres espècies properes com seria el cas de Myotis brandtii però no podem confirmar que aquesta regió codifica per la nostra proteïna per falta d’informació.
Per tant, amb les dades que tenim no podem assegurar que l’homòleg de cisteïna GPx8 es trobi en el genoma d'Eptesicus fuscus.

Hem fet un arbre filogenètic comparant les querys usades d’Homo sapiens provinents de SelenoDB amb les nostres prediccions de Genewise a Eptesicus fuscus. Podem observar que hem realitzat un bon anàlisi, ja que hem escollit bé els hits de cada membre de la família de GPx. En el cas de GPx1, GPx2, GPx3, GPx4, GPx7, GPx8 veiem que es corresponen correctament als nostres hits. Veiem al cas de GPx6 en què no s’ha trobat un bon hit, i això ens reforça la nostra hipòtesi que aquesta no es troba al genoma d’Eptesicus fuscus. També teníem dubtes pel que fa a GPx5. A l’arbre veiem que no s’associa amb el nostre hit. A més, els resultats del nostre anàlisi ens han fet suposar que aquesta proteïna no es troba ben anotada al nostre genoma. Per tant, gràcies a l’arbre filogenètic podem suposar que no hem seleccionat el hit correcte a GPx5.
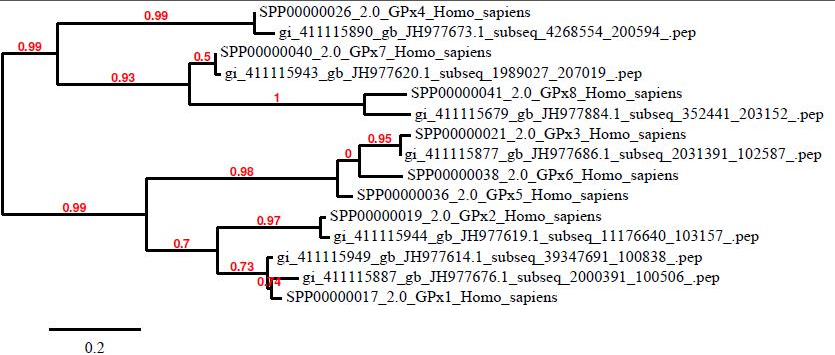
MSRA
MSRA és la metionina sulfòxid reductasa A. És un homòleg de cisteïna propi de mamífers que té com a funció la reducció del sulfòxid de metionina a metionina, per reparar el dany oxidatiu.
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
El Blast de la proteïna MSRA dóna un únic hit significatiu (E value superior o igual a 10-5 el qual es troba a l'scaffold JH977673.1, de la posició 7096454 a la 7487032 i està codificada en el sentit revers (-)).
Les dades obtingudes de SelenoDB de la MSRA de Myotis lucifigus a partir de la qual hem fet l’anàlisi després del corresponent T-Coffee té 6 exons. Com que no és una selenoproteïna, no conté l’aminoàcid selenocisteïna.
La proteïna predita per Exonerate té 390579 nucleòtids, incloent 6 exons i amb un raw score de 1205. Per altra banda, la predicció de Genewise dóna lloc a una proteïna de 390578 nucleòtids, amb 6 exons i un score de 496.47 bits. Per tant, tots dos mètodes prediuen la mateixa proteïna. A més, observem com ambdós T-Coffees (tant el d’Exonerate com el de Genewise) tenen un score de 100, ja que hi ha correspondència entre la query de Myotis lucifigus i el genoma d’Eptesicus fuscus.
Observant els resultats del Seci, veiem que n’hi ha un de predit que està convenientment col·locat a la posició 3’ de la proteïna i amb un bon valor d’energia lliure. Tot i això, no s’ha predit cap selenoproteïna perquè no conté l’aminoàcid selenocisteïna.
Els nostres resultats, doncs, demostren la conservació de l'aminoàcid cisteïna i un element SECI. La nostra hipòtesi és que MSRA inicialment era una selenoproteïna, però ha evolucionat canviant la selenocisteïna per la cisteïna i encara no s'ha perdut la maquinària pròpia de la selenoproteïna.
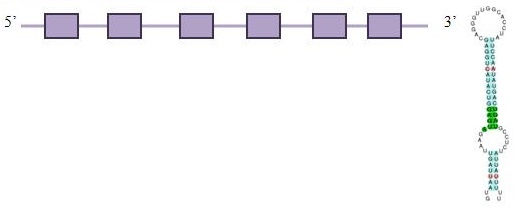
Sel15
El gen de la Sel15 (selenoproteïna de 15 kDa) està localitzat en el cromosoma 1p31, un locus genètic comunament mutat o deleccionat en càncers humans. Comparteix un 31% de la seva seqüència amb SelM, i ambdues es localitzen al reticle endoplasmàtic. Sel M té dominis Cys-X-X-Sec i Sel15 Cys-X-Sec. Es creu que totes dues tenen una funció en el control del plegament de les proteïnes en el reticle endoplasmàtic. A més, s’han dut a terme diversos estudis que relacionen Sel15 amb càncer.
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
Aquí ens trobem amb la proteïna Sel15 que es troba al scaffold JH977787.1. , aquest scaffold es troba situat a la regió 167686 fins a 139489 (BLAST) del genoma. Està codificada en el sentit invers (reverse). La predicció d’Exonerate té 28198 nucleòtids, que inclouen cinc exons i quatre introns i un raw score de 797; mentre que la predicció de Genewise té 28197 nucleòtids, que inclouen cinc exons i quatre introns i un score de 285,03 bits. En aquest cas, les prediccions són quasi idèntiques, a excepció que l’Exonerate que dóna una predicció més llarga, per tant sols un nucleòtid de més i es pot veure que els dos mètodes emprats ens donen uns bons alineaments del T-Coffee corresponent (score de 100).
La proteïna de Myotis lucifugus té una selenocisteïna i hem observat que aquesta es troba conservada tant a l’Exonerate com al Genewise, trobant-se al segon exó de les proteïnes predites per ambdós mètodes.
El programa SeciSearch ha trobat un possible element Seci a l’extrem 3’ de la cadena complementària del gen. En quant al programa Seblastian, també prediu un element SECIS, amb un grau A i situat a una distància de 791 nucleòtids de la selenocisteïna i a 584 del final del gen. També obtenim la predicció de la proteïna, per tant és una confirmació que aquesta selenoproteïna existeix en Eptesicus fuscus. Després d’haver analitzat tota la informació anterior, es podria predir una estructura de la proteïna Sel15:
GENEWISE I EXONERATE
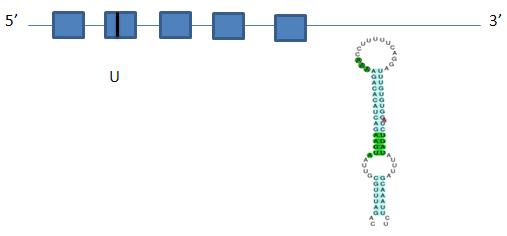
SelH
La selenoproteïna (Sel) és una proteïna de 14kDA, amb un domini tioredoxina i un motiu Cys-X-X-Sec conservat. La seva expressió és àmpliament distribuïda en una gran varietat de teixits i en un gran nombre d’etapes embrionàries durant el desenvolupament. En Drosophila, s’ha vist que Sel és essencial per la viabilitat i la defensa antioxidant.
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
La SelH disponible a SelenoDB que hem utilitzat com a query (en aquest cas és la query de Myotis, després de realitzar el corresponent T-Coffee) té un total de 3 exons, amb l'aminoàcid selenocisteïna al segon exó. Tenim 3 hits situats als següents scaffolds: JH977834.1, JH977762.1 i JH977651.1. Observant els valors d'E-value i el nombre d'exons dels diferents hits, ens quedem amb el primer scaffold, ja que tant l'Exonerate com el Genewise ens donen 3 exons, igual que la seqüència disponible a SelenoDB. A més, veiem que en tots dos casos, l'aminoàcid selenocisteïna es troba al segon exó.
L'scaffold triat (JH977834.1) està situat entre les posicions 969760 i 970232 corresponents al BLAST, i està codificada en direcció forward (+). L'Exonerate ha predit una proteïna de 554 nucleòtids, amb un raw score de 495 i, tal i com hem comentat anteriorment, amb 3 exons i l'aminoàcid selenocisteïna en el segon. Per altra banda, el Genewise prediu una proteïna de 553 nucleòtids, amb un score de 198 bits i també amb 3 exons i la selenocisteïna en el segon. Per tant, Exonerate i Genewise prediuen la mateixa proteïna, amb una diferència d'un únic nucleòtid entre ambdues tècniques.
El T-Coffee d'Exonerate dóna solapaments entre la query triada i el genoma d'Eptesicus fuscus, amb un score de 99. El mateix bon resultat observem en el T-Coffee de Genewise, amb un score de 99. A més, trobem l'element SECI convenientment situat a la posició 3'. Per tant, la Selenoproteïna H es troba conservada en Eptesicus fuscus.
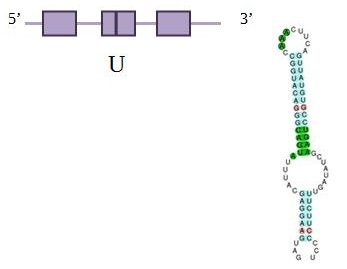
SelI
Recentment s’ha conegut la funció de SelI, a causa de l’homologia de la seva seqüència amb enzims implicats en la síntesi de fosfolípids, la qual es du a terme en la majoria de teixits, motiu pel qual SelI s’expressa en diverses localitzacions.
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
La proteïna Sel I es troba al scaffold JH977617.1, a la regió que va de 24417179 fins a 24372156 (BLAST) i està codificada en sentit invers (reverse). La predicció d’Exonerate té 45024 nucleòtids, que inclouen deu exons i nou introns i un raw score de 2004. Mentre que la predicció de Genewise té 45023 nucleòtids, que inclouen deu exons i nou introns i un score de 809.84 bits. En aquest cas les prediccions són quasi idèntiques a excepció que l'Exonerate dóna una predicció més llarga per només un nucleòtid de més. Es pot veure que els dos mètodes emprats ens donen uns bons alineaments dels T-Coffees corresponents (score de 100).
La proteïna de Myotis lucifugus té una selenocisteïna i hem observat que aquesta es troba conservada tant a l’Exonerate com el Genewise, trobant-se a l'últim exó de les proteïnes predites per Exonerate i Genewise.
El programa SeciSearch ha trobat tres possibles elements Seci a l’extrem 3’ de la cadena complementària del gen. Això dona suport a la hipòtesi que aquesta selenoproteïna es troba en Eptesicus fuscus.
En quant el programa Seblastian ens prediu un SECIS, amb un grau A i a una distància de 1303 nucleòtids de la selenoproteïna i a 1333 de la selenocisteïna. Per tant, això confirma que existeix aquesta selenoproteïna a E.fuscus.
Després d’haver analitzat tota la informació anterior, es podria predir una estructura de la proteïna SelI:
EXONERATE I GENEWISE:
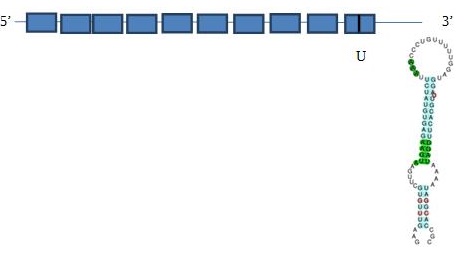
SelK
La selenoproteïna K és una proteïna petita de 16kDa, localitzada tant a la membrana del reticle endoplasmàtic com a la membrana plasmàtica. Està altament expressada al cor, on es creu que actua com a antioxidant. Vam agafar com a query SELK none 3 de Myotis lucifugus, ja que la selenoproteïna començava per metionina i tenia una selenocisteïna conservada.
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
Aquí ens trobem amb la proteïna Selk que es troba al scaffold JH977647.1, a la regió que va de 12803997 a 12805757 (BLAST) i està codificada en el mateix sentit que l’anotació (forward). La predicció d’Exonerate té 6591 nucleòtids, que inclouen quatre exons i tres introns i un raw score de 422. Mentre que la predicció de Genewise té 2953 nucleòtids, que inclouen tres exons i dos introns i un score de 164,50 bits. Les dues prediccions ni comencen ni acaben al mateix nucleòtid i tampoc tenen la mateixa estructura pel que fa a exons i introns, ja que la predicció d’Exonerate és més llarga.
La selenoproteïna de Myotis lucifugus té una selenocisteïna i hem observat que es troba conservada a l’últim exó (el quart) de la proteïna predita per l'Exonerate, però no en la proteïna predita pel Genewise, ja que s’acaba dos aminoàcids abans. El que hem observat podria ser perquè Genewise considera el codó TGA com a codó stop i per tant s’acabaria la predicció i no es detectaria la selenocisteïna. Amb tot això podríem pensar que aquesta proteïna presenta la selenocisteïna conservada. Per corroborar la nostre hipòtesi vam agafar també la Selk d’Homo sapiens i vam mirar si la proteïna predita pel Genewise també s’acabava abans de l’aparició de la selenocisteïna i així va ser.
El programa SeciSearch ha trobat dos possibles elements Seci a l’extrem 3’ del gen. En quant al programa Seblastian ens prediu un SECIS, amb un grau A, el qual es troba a una distància de 370 nucleòtids de la selenocisteïna i a 376 del final del gen. Això pot ser, ja que el Seblastian és més restrictiu a l'hora de predir els Secis.
La presència d'aquest SECIs dóna suport a la predicció que es tracta d'una selenoproteïna que està conservada. A més, vam mirar la selenoproteïna predita per aquest programa. Després d’haver analitzat tota la informació anterior, es podria predir una estructura de la proteïna SelK:
EXONERATE:
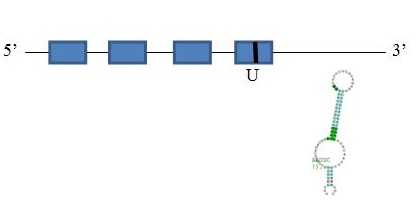

SelM
El gen de la selenoproteïna SelM s’expressa en una gran varietat de teixits. A més, codifica per una proteïna que està localitzada en el reticle endoplasmàtic. Igual que la proteïna de 15kDa, es una proteïna petita (de 15kDa també), amb un 31% d’identitat amb la seqüència de 15Kda, i ambdues es localitzen al reticle endoplasmàtic. Presenta un motiu Cys-X-X-Sec.
A SelenoDB hi havia dues proteïnes SelM així que vam emprar T-Coffee per saber quina de les dues havíem d’utilitzar per a fer l’anàlisi (veure arxiu). D’aquesta manera ens vam adonar que havien anotat dues vegades la mateixa selenoproteïna.
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
La proteïna SelM es troba al scaffold JH977628.1 i està codificada en el mateix sentit que l'anotació (forward). La predicció de l’Exonerate té 2493 nucleòtids que inclouen 5 exons i 4 introns, amb un raw score de 702, mentre que la predicció del Genewise és de 2407 nucleòtids que inclouen 5 exons i 4 introns, amb un score de 248,29 bits. En aquest cas, la predicció és més llarga en Exonerate. Es pot veure que els dos mètodes emprats ens prediuen el mateix número d'exons i introns i que obtenim uns bons alineaments del T-Coffee (score de 96 Exonerate i 97 de Genewise).
La proteïna d' Homo sapiens té una selenocisteïna i hem observat que aquesta es troba conservada tant a l’Exonerate com en el Genewise, trobant-se en el segon exó de les proteïnes predites per l’Exonerate i el Genewise.
En quant al programa SeciSearch3 ens prediu un SECIS, amb un grau B. Tot i això, per algun motiu que desconeixem, el programa Seblastian no prediu cap selenoproteïna. Després d’haver analitzat tota la informació anterior, es podria predir la següent estructura de la proteïna SelM:
EXONERATE I GENEWISE:
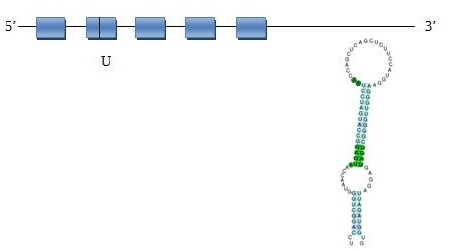
SelN
La Selenoproteïna N es una proteïna transmembrana localitzada a la membrana del reticle endoplasmàtic, amb un tamany de 70 kDA. Existeixen dues isoformes, la 1 que és el transcrit amb la llargada total i la isoforma 2, que exclou l’exó 3 de l’splicing. Tots dos transcrits es troben al múscul esquelètic, cervell, pulmó i placenta, amb la isoforma 2 sent la que es troba de manera majoritària.
Diversos estudis han relacionat mutacions en el gen de la SelN amb malalties musculars, com la malaltia del multiminocore i la distròfia muscular congènita amb rigidesa espinal i síndrome respiratori restrictiu.
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
Al fer el Blast, només hem obtingut un hit significatiu (E-value major o igual a 10-5) per SelN, el qual es troba a l’scaffold JH977655.1, de la posició 3888035 a la 3876508 i està codificada en sentit revers (-). A SelenoDB, veiem que la transcripció de la primera SelN humana (la qual hem usat per comparar en Eptesicus fuscus, després d’haver realitzat el T-Coffee corresponent: conté 13 exons i la selenocisteïna es troba al 3er exó. El resultat de l’Exonerate dóna un total de 178912 nucleòtids, amb 30 introns i 31 exons, amb un score de 137 bits. Per altra banda, el resultat del Genewise dóna un total de 11545 nucleòtids, amb 11 exons i 12 introns i un score de 918,64 bits.
Veiem que el resultat del T-Coffee de l’Exonerate té un score de 64 i no és gens informatiu, ja que hi ha poca correspondència entre la query humana i el genoma d’Eptesicus fuscus. Per altra banda, el T-Coffee de Genewise sí és informatiu, ja que té un score de 96 i hi ha un alt nivell de correspondència entre la SelN humana i el genoma d’Eptesicus fuscus. Però veiem que el Genewise comença a la posició 60, per tant, ens perdem la primera part de la proteïna. Pel que fa a la posició de les selenocisteïnes, a la SelN humana es troben en el tercer i en el desè exó. A l’Exonerate els trobem en el cinquè i el vint-i-cinquè, mentre que en el Genewise es troba en el segon i en el setè. Però aquests resultats del Genewise són compatibles amb el fet que hem explicat anteriorment que el genoma d’Eptesicus fuscus comença més tard que la query, motiu pel qual ens perdem els primers exons, i no es corresponen totalment amb la posició obtinguda de les dades de SelenoDB.
Pel que fa a l’anàlisi del Secis i del Seblastian veiem que la selenocisteïna SelN es troba conservada en Eptesicus fuscus, ja que té un element Secis, el qual es troba a la posició 3’, a la cadena reversa i amb valor A. Tal i com veiem al web, ens prediu dos elements Secis, però ens quedem amb el primer ja que té millor categoria (A) i es troba a la mateixa cadena on hem predit la proteïna (-). Per tant, a partir dels resultats del Genewise podem imaginar la següent estructura: (la primera caixa corresponent als exons significa que hi ha un nombre indeterminat d’exons que no s’han pogut predir perquè la predicció de Genewise no començava a la posició 0).
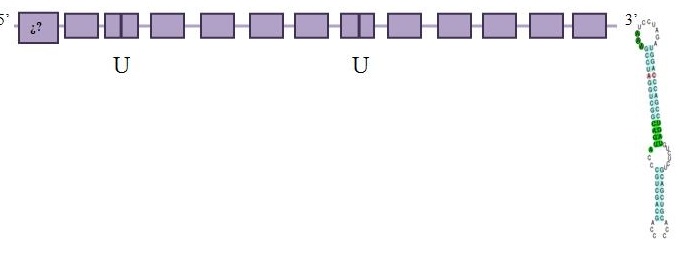
SelO
Tot i saber que la selenoproteïna humana consta de 669 aminoàcids i té un pes de 73,4 kDa, no es té informació sobre la seva distribució tissular o subcel·lular, així com la funció fisiològica. La presència del motiu Cys-X-X-Sec suggereix una funció redox, però no hi ha demostració experimental.
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
Al fer el BLAST, obtenim 4 hits significatius (E-value major o igual a 10-5), els quals es troben als següents scaffolds (ordenats de millor a pitjor scaffold): JH977730.1, JH977772.1, JH978053.1 i JH977626.1. A SelenoDB, observem que la SelO humana anotada que hem fet servir com a query (després d’haver analitzat les diferents seqüències de SelO fent un T-Coffee: té 9 exons, amb la selenocisteïna a l’últim exó.
Analitzant els T-Coffees tant d’Exonerate com de Genewise, observem com els d’Exonerate donen poques correspondències, mentre que principalment el T-Coffee de Genewise de l’scaffold JH977730.1 (codificat a la seqüència forward: +, de la posició 3429992 a la 3445764) dóna un valor de 96 i hi ha molts solapaments entre la query i el genoma d’Eptesicus fuscus. L’exonerate d’aquest scaffold dóna 15842 nucleòtids, amb 11 exons i 10 introns i un raw score de 2648. Per altra banda, el seu Genewise dóna 15832 nucleòtids, amb tabmbé 11 exons i 10 introns i un score de 1064,36 bits. Per tant, tots dos mètodes prediuen la mateixa proteïna, amb una diferència de 10 nucleòtids. Tal i com hem dit, el T-Coffee de Genewise és significatiu, amb un score de 96 i molts solapaments entre ambdues seqüències.
A més, veiem com la selenocisteïna, igual que a les dades disponibles a SelenoDB, es troba a l’últim exó de l’scaffold que hem triat. Pel que fa a l’anàlisi dels Secis i el Seblastian veiem com sí que presenta un element Secis, amb un bon valor d’energia lliure i situat convenientment a la regió 3’ de la proteïna predita.
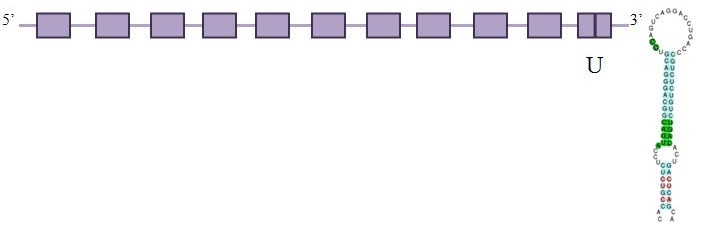
SelP
El gen de SelP codifica per una selenoproteïna que correspon a una glicoproteïna extracel·lular. És inusual ja que conté múltiples residus selenocisteïna en el seu transcrit. És una proteïna d’unió a l’heparina i que podria estar associada amb les cèl·lules endotelials. Pot funcionar com a antioxidant extracel·lular i es troba implicada en el transport de seleni a diferents òrgans i teixits.
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els Secis predits.
La selenoproteïna SelP es troba al scaffold JH977734.1, a la regió que va de 3670544 a 3667014 (BLAST) i està codificada en sentit invers (reverse). La predicció d’Exonerate té 3669 nucleòtids, que inclou cinc exons i quatre introns i un raw score de 1516; mentre que la predicció de Genewise té 3422 nucleòtids, incloent cinc exons i quatre introns i un score de 454.20 bits. En aquest cas observem que la predicció feta per l'Exonerate és més llarga mentre que la de Genewise és 247 posicions més curta. Observem com les catorze selenocisteïnes presents en Myotis lucifugus apareixen en la proteïna predita d’Eptesicus fuscus, i inclús apareix un nou TGA al primer exó. En quant al Genewise, s’observa que ens prediu una proteïna que acaba abans i per tant no podem saber si les últimes selenocisteïnes es conserven. S’observa com de les set que hi haurien de ser, fins a la proteïna predita només apareixen cinc ja que les altres dues no s’han conservat. Es pot veure que els dos mètodes emprats ens donen uns bons alineaments del T-Coffee corresponent, encara que la predicció del Genewise és més curta i no se sap si les últimes selenocisteïnes es conserven.
El programa SeciSearch ha trobat dos possibles elements Seci a l’extrem 3’ de la cadena complementària del gen, a l’igual que en la selenoproteïna de Myotis lucifugus. En quant el programa SeciSearch3 ens prediu dos SECIS, amb un grau A i una amb grau B, totes tres a la cadena complementària. En quant el programa Seblastian, no ens prediu cap selenoporoteïna i això podria ser per algun error procedimental. Després d’haver analitzat tota la informació anterior, es podria predir una estructura de la proteïna SelP:
EXONERATE:
GENEWISE:

Tornar a dalt


FAMÍLIA SelR
La selenoproteïna R és part de la família de proteïnes metionina sulfoxido reductasa (Msr), que inclou MsrA, MsrB2 i MsrB3. Els enzims d’aquesta família són enzims de reparació (redueixen els residus de metionina oxidats en les proteïnes). Aquesta selenoproteïna és expressada en una gran varietat de teixits adults i fetals, i es localitza tant en el nucli cel·lular com en el citosol. Vam obtenir diferents hits i utilitzant els valors d’E-value i la informació dels bits del BLAST vam poder assignar un hit a cada una de les proteïnes. Vam veure que es solapaven els scaffolds entre les diferents proteïnes de la família.
SelR1
En aquest cas no vam realitzar cap T-Coffee inicial perquè de totes les proteïnes SelR que hi havia disponibles a SelenoDB, únicament hi havia una que contingués l’aminoàcid selenocisteïna, motiu pel qual vam triar directament aquesta com a query.
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
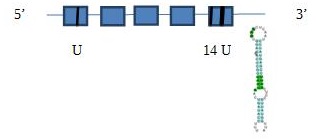
La proteïna SelR1 es troba al scaffold JH977648.1, que està a la regió de 675815 a 676429 (BLAST) i està codificada en el sentit invers (reverse). La predicció de l’Exonerate té 3480 nucleòtids que inclouen quatre exons i tres introns, amb un raw score de 566. Mentre que la predicció del Genewise és de 3479 nucleòtids que inclouen quatre exons i tres introns, amb un score de 206,32 bits. En la predicció feta pel Genewise obtenim una proteïna amb un nucleòtid menys en comparació amb l’Exonerate, però obtenim el mateix número d’exons i introns. Observem que tant en Exonerate com en Genewise la selenocisteïna present a la selenoproteïna d’Homo sapiens es troba conservada a l’exó tres.
El T-Coffee d'Exonerate ens dóna un molt bon alineament, amb un score de 98 i el de Genewise un alineament amb un score de 99. En quant el programa SeciSearch3 ens prediu un Secis amb un grau A. Seblastian, a més, prediu una selenoproteïna amb la selenocisteïna conservada a l’exó tres. Amb tota aquesta informació podem afirmar que aquesta selenoproteïna es troba conservada en Eptesicus fuscus. Després d’haver analitzat tota la informació anterior, es podria predir una estructura de la proteïna SelR1:
EXONERATE I GENEWISE:
SelR2
Per triar quina proteïna triar com a query de totes les disponibles a la base de dades de SelenoDB vam realitzar T-Coffee (veure arxiu).
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
En aquest cas vam obtenir dos hits molt bons i per a poder escollir quin dels dos emprar per a fer l’anàlisi posterior vam optar per mirar la filogènia (veure arxiu) i vam obtenir només un hit, que estava al scaffold JH977660.1.

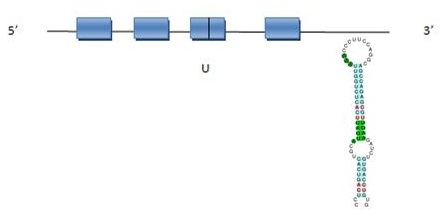
Aquí ens trobem amb la proteïna que es troba al scaffold JH977660.1, que està a la regió de 8472016 a 8483248 (BLAST) i està codificada en el mateix sentit que l’anotació (forward). La predicció de l’Exonerate té 11272 nucleòtids que inclouen quatre exons i tres introns, amb un raw score de 653; mentre que la predicció del Genewise és de 11259 nucleòtids que inclouen quatre exons i tres introns, amb un score de 257.79 bits. Observem que entre els dos mètodes, la proteïna predita només dista de 13 nucleòtids i que obtenim el mateix número d’exons i introns en els dos mètodes emprats.
El score del T-Coffee a partir de la proteïna d’Exonerate és de 99, encara que la primera part de la proteïna no s’observa per molt que haguéssim obert 100.000 posicions abans d’executar programa. El T-Coffee de Genewise presenta un alineament molt semblant. Com esperàvem, no hem trobat ni Secis ni selenoproteïnes amb el programa Seblastian.
Després d’haver analitzat tota la informació anterior, es podria predir una estructura de la proteïna SelR2:
EXONERATE I GENEWISE
SelR3
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
La proteïna SelR3 es troba al scaffold JH977659.1, que està a la regió de 7928581 a 8036737 (BLAST) i està codificada en sentit invers (reverse). La predicció de l’Exonerate té 178168 nucleòtids que inclouen cinc exons i quatre introns, amb un raw score de 653; mentre que la predicció del Genewise és de 178167 nucleòtids que inclouen cinc exons i quatre introns, amb un score de 266,52 bits. Observem que entre els dos mètodes, la proteïna predita només dista d’un nucleòtid i que obtenim el mateix número d’exons i introns en els dos mètodes emprats.
El score de T-Coffee fet a partir de la proteïna predita per Exonerate és de 98 mentre que el T-Coffee a partir de Genewise obtenim un score de 99 i s’observa com la part inicial de la proteïna no s’observa en la proteïna predita però sí que es conserva la resta. Com esperàvem, no hem trobat ni Secis ni selenoproteïnes amb el programa Seblastian.
Després d’haver analitzat tota la informació anterior, es podria predir una estructura de la proteïna SelR3:
EXONERATE I GENEWISE:
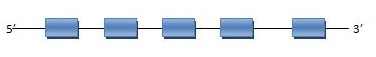
SelS
La selenoproteïna S és una proteïna transmembrana localitzada al reticle endoplasmàtic i a les membranes plasmàtiques que s’expressa en una gran varietat de teixits. S’ha suggerit la seva participació en l’eliminació de proteïnes malplegades per la seva degradació en el lumen del RE i en la protecció de les cèl·lules front el dany oxidatiu i de l’estrès del reticle endoplasmàtic induït per l’apoptosi. A més, certs SNPs en el promotor de SelS s’han vist que regulen nivells de citoquines inflamatòries com IL-1, TNF-alfa i IL-6. També s’ha vist com l’expressió de SelS està modulada pel metabolisme de la glucosa i l’estrès del reticle endoplasmàtic. Diverses malalties s’han associat amb variacions genètiques de SelS, incloent malalties cardiovasculars i embòlies, artritis reumatoide i càncer gàstric, tot i que no tots els estudis demostren aquestes troballes.
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
Al fer el Blast, únicament obtenim un hit significatiu (E-value major o igual a 10-5), situat a l’scaffold JH977624.1, de la posició 5807982 a la 5807468 i està codificada en sentit revers (-).
A SelenoDB, observem que la SelS humana triada com a query a partir dels resultats del T-Coffee conté 6 exons i 6 introns, i té l’aminoàcid selenocisteïna en el primer exó. L’Exonerate ens prediu una proteïna de 482 nucleòtids, amb un únic exó i un raw score de 526, amb la selenocisteïna en aquest únic exó. Per altra banda, el Genewise prediu una proteïna de 293 nucleòtids, distribuïts en 3 exons i amb un score de 227,84 bits, amb la selenocisteïna al primer exó.
El T-Coffee d’Exonerate té un score de 63, ja que no hi ha masses regions de solapaments entre la query humana i el genoma d’Eptesicus fuscus. El T-Coffee de Genewise té un millor score (95) ja que hi ha molt solapament entre ambdues seqüències, tot i que la query comença més tard i no agafa la part inicial del genoma d’Eptesicus fuscus. Per tant, com que els resultats de Genewise són més similars a l'estructura de SelS humana, ens quedme amb aquests resultats per a la interpretació.
Tot i que les posicions dels elements SECI ens portaria a pensar que no hi haurien d'haver, el Seblastian sí que ens prediu l'element SECI que observem a la figura.
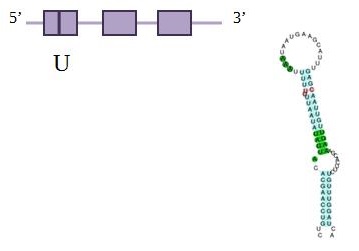
SelT
La selenoproteïna T es troba expressada de manera ubiqua des de l’etapa embrionària fins a l’etapa adulta a les rates, principalment en el reticle endoplasmàtic a través d’un domini hidrofòbic. SelT es membre una subfamília de selenoproteïnes (la qual inclou SelW, Sel i SelV) que presenten similaritat, ja que contenen un domini tioredoxina i un motiu Cys-X-X-Sec conservat. Es creu que SelT participa en la regulació de l’estrès cel·lular, tot i que són necessaris més estudis per demostrar aquesta funció. A mes, s’ha vist que SelT està implicada en vies de senyalització activades per PACAP, potser per la regulació del calci. Tot i això, es necessita més investigació en aquesta direcció.
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits. Al fer el Blast, obtenim dos hits significatius (E-value major o igual a 10-5), situats a l’scaffold JH977691.1 (codificat en sentit forward: +, de la posició 4440491 a la 4440982) i JH978008.1 (codificat en sentit revers: -, de la posició 128812 a la 149206), ordenats de millor a pitjor E-value. A partir de les dades disponibles a SelenoDB de la SelT humana que hem fet servir com a query després del T-Coffee corresponent , veiem que té 5 exons i el codó corresponent a la selenocisteïna està al segon codó. Per fer l’anàlisi seleccionem l’scaffold JH978008.1, ja que tant el seu Exonerate com el seu Genewise prediuen una proteïna amb 5 exons (mentre que l’Exonerate de l’altre scaffold només en prediu un, mentre que el seu Genewise en prediu 7).
L’Exonerate de l’scaffold seleccionat prediu una proteïna de 110395 nucleòtids, amb 5 exons (amb l’aminoàcid selenocisteïna al segon exó) i un raw score de 968. El Genewise d’aquest mateix scaffold prediu una proteïna de 110394 nucleòtids, amb 5 exons i un score de 386,68 bits. Per tant, tots dos mètodes prediuen la mateixa proteïna, amb un únic nucleòtid de diferència.
El T-Coffee d’Exonerate té un score de 100, ja que té una correspondència perfecta entre la query i el genoma d’Eptesicus fuscus. El mateix observem en el cas del T-Coffee de Genewise, que també té un score de 100 amb correspondència entre ambdues seqüències, també pel que fa a l’aminoàcid selenocisteïna. Veiem que hi ha un element Seci que es troba convenientment col·locat a la posició 3’ de la proteïna predita, amb un bon valor d’energia lliure, de manera que es confirma que Eptesicus fuscus conté la selenoproteïna Sel T.
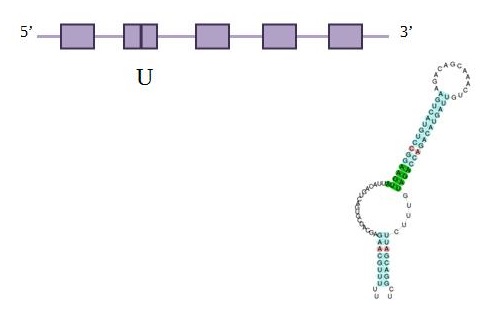
FAMÍLIA SelU
SelU és una selenoproteïna de la qual se’n desconeix la funció. En el cas de SelU1 hem comparat amb un T-Coffee les seqüències de Myotis lucifugus, Pteropus vampyrus i Homo sapiens i hem vist que són gairebé iguals.
Hem escollit com a query una seqüència de SelU1 humana perquè a més de ser molt semblant a la resta comença per metionina (veure arxiu).
En el cas de SelU2 també hem comparat amb un T-Coffee la SelU2 humana i la de Pteropus amb les diferents de Myotis lucifugus i hem vist semblances: la humana semblava millor anotada així que hem escollit com a query SelU2 d'Homo sapiens (veure arxiu).
SelU1
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
El millor hit d’aquesta proteïna es troba a l’scaffold JH977662.1 a la posició 10914305- 10905350 a la cadena forward. A SelenoDB, observem que SelU1 humana anotada que hem fet servir com a query no és una selenoproteïna, té 5 exons.
Analitzant els T-Coffees observem com el d’Exonerate dona més o menys bona correspondència T-Coffee Genewise. L’Exonerate d’aquest scaffold dóna 8956 nucleòtids, amb 5 exons i 4 introns i un raw score de 1042. Per altra banda, el seu Genewise dóna 8955 nucleòtids, amb 5 exons i 4 introns i un score de 427 bits. Per tant, tots dos mètodes prediuen la mateixa proteïna, amb una diferència d’1 nucleòtid.
Pel que fa a l’anàlisi del Seblastian veiem com no ens pot predir cap selenoproteïna però sí que presenta un element Secis, de grau A. Els nostres resultats, doncs, demostren la conservació de l'aminoàcid cisteïna i un element Seci. La nostra hipòtesi és que GPx5 inicialment era una selenoproteïna, però ha evolucionat canviant la Selenocisteïna per la Cisteïna i encara no s'ha perdut la maquinària pròpia de la selenoproteïna.
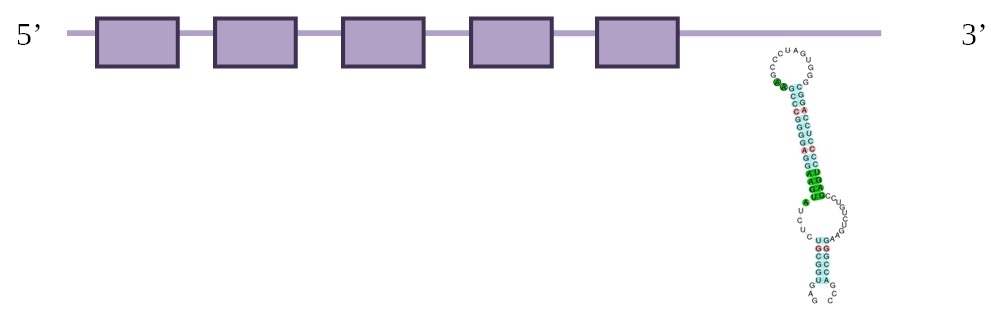
SelU2

En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
El millor hit d’aquesta proteïna es troba a l’scaffold JH977714.1, entre les posicions 3434670-3437632 a la cadena forward. A SelenoDB, observem que la SelU2 humana anotada que hem fet servir com a query no és una selenoproteïna i té 6 exons.
Analitzant els T-Coffees observem com el d’Exonerate dóna correspondència molt dolenta. L’Exonerate d’aquest scaffold dóna 2963 nucleòtids, amb 2 exons i 1 intró i un raw score de 405. Per altra banda, el seu Genewise dóna 8587 nucleòtids, amb 4 exons i 3 introns i un score de 186 bits. Hi ha una diferència molt gran entre les prediccions d’aquests dos mètodes.
Els resultats trobats no són massa significatius, perquè tal i com passa en el genoma de Myotis lucifugus, SelU2 no està ben anotada (després del T-Coffee, veiem que proteïnes no anotades en Myotis lucifugus sembla que es corresponen amb SelU2 humana), motiu pel qual no podem afirmar la presència de SelU2 en Eptesius fuscus.
A més, veiem com no hi ha selenocisteïna, igual que a les dades disponibles SelenoDB. No detectem cap selenocisteïna, ja que conté cisteïna enlloc de la selenocisteïna.
Pel que fa a l’anàlisi dels Secis i el Seblastian no s’han trobat elements SECIs.
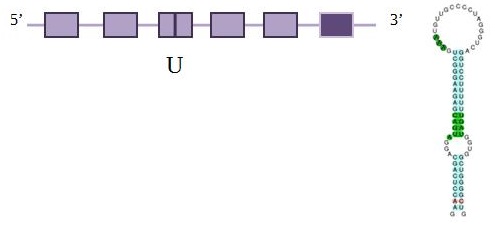
SelV
Tot i no conèixer la funció de la Selenoproteïna V, se sap que s'expressa de manera exclusiva als testicles. SelV presenta homologia amb altres selenoproteïnes com Sel, SelT i SelW, ja que totes aquestes tenen un domini tioredoxina i un motiu Cys-X-X-Sec conservat, suggerint funcions redox. Cal considerar que SelV és una duplicació de SelW.
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
Al fer el Blast, únicament obtenim un hit significatiu (E-value major o igual a 10-5), situat a l’scaffold JH977621.1, de la posició 1200843 a la 1198384 i està codificada en sentit revers (-).
A SelenoDB, observem que la SelV humana triada com a query (sense realitzar cap T-Coffee, perquè només hi ha una seqüència de proteïna SelV en humans, i no està disponible ni en Pteropus vampyrus ni en Myotis lucifigus) té 5 exons i 5 introns, amb el codó codificant per selenocisteïna (TGA) en el segon exó. L’Exonerate ens prediu una proteïna de 50528 nucleòtids, amb 11 exons i 10 introns i un raw score de 121, amb la selenocisteïna en el 5è exó. Per altra banda, Genewise prediu una proteïna de 3157 nucleòtids, amb 6 exons i 5 introns i un score de 191,99 bits, amb la selenocisteïna en el 3r exó. Cal considerar que els cinc primer exons són codificants, mentre que el 6è té l'última porció del 3'-UTR, incloent l'element Secis.
El T-Coffee d’Exonerate dóna un score de 59 i mostra pocs solapaments entre la query humana de SelV i el genoma d’Eptesicus fuscus. El T-Coffee de Genewise presenta més solapaments entre ambdues seqüències, motiu pel qual té un score de 88. Per tant, ens basem en els resultats del Genewise per a la interpretació. Les posicions dels elements Secis es troben a la regió 3' sempre i quan tinguem en compte les posicions del Genewise, ja que suposem que l'Exonerate agafa una regió massa ampla i per això no es troba l'element Secis a la regió correcta en l'Exonerate, però sí en el Genewise.
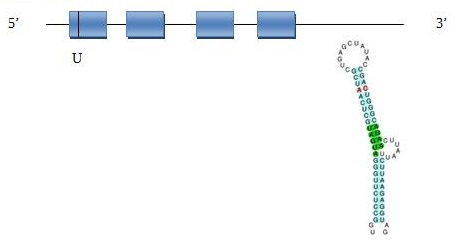
FAMÍLIA SelW
Sel W1
La selenoproteïna W és una selenoproteïna petita (9,5 kDa) que conté el motiu Cys-X-X-Sec. Aquesta selenoproteïna està altament expressada en múscul esquelètic i cor i pot estar involucrada en reaccions redox.
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
Vam obtenir diferents hits i utilitzant els valors d’E-value i la informació dels bits del BLAST vam poder assignar un hit a cada una de les proteïnes. A SelenoDB hi havia diverses querys, per tant, vam emprar T-Coffee per escollir quina havíem d’emprar com a selenoproteïna problema (veure arxiu).
Aquí ens trobem amb la proteïna SelW que es troba al scaffold JH977711.1, que va des de la regió 3474306 fins a 3474380 (BLAST) i està codificada en sentit invers (reverse). La predicció de l’Exonerate té 590 nucleòtids que inclouen quatre exons i tres introns, amb un raw score de 352. Mentre que la predicció del Genewise és de 480 nucleòtids que inclouen tres exons i dos introns, amb un score de 118,81 bits.
La proteïna d’ Homo sapiens té una selenocisteïna i hem observat que aquesta en Exonerate es troba conservada al primer exó de la proteïna predita mentre que no en Genewise, ja que aquest programa prediu una proteïna que comença cinc posicions més enllà de la selenoproteïna. En els T-Coffees obtenim bons alineaments i per tant, bons valors (scores). Tot i això, en el T-Coffee Genewise no es detecta la selenocisteïna, ja que l’alineament comença la proteïna predita posicions més enllà.
Hem emprat SeciSearch3 i ens ha predit un element Seci amb grau B, mentre que Seblastian no ens prediu una selenoproteïna, això podria ser degut a errors de procediment. Després d’haver analitzat tota la informació anterior, es podria predir una estructura de la selenoproteïna Sel W1:
EXONERATE:

Vam emprar el programa present a la web Phylogeny.fr per a fer la filogènia de SelW i ens vam adonar que el hit escollit no era el més adient, encara que igualment ens prediu la selenoproteïna, ja que obtenim un bon valor a la filogènia (veure arxiu).

Sel W2
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
La proteïna SelW2 es troba al scaffold JH977703.1, a la regió que de va des de 27943899 fins a 27944084, i està codificada en sentit invers (reverse). La predicció de l’Exonerate té 813 nucleòtids que inclouen quatre exons i tres introns, amb un raw score de 413; mentre que la predicció del genewise és de 812 nucleòtids que inclouen tres exons i dos introns, amb un score de 171,40 bits. Hem vist que l’Exonerate prediu una proteïna per només un nucleòtid de més en comparació amb Genewise. Hem vist que els T-Coffee corresponents a cada mètode presenten uns bons scores i bons alineaments.
No s’han trobat Secis, com esperàvem, ja que és una proteïna homòloga de cisteïna. Amb tota la informació anterior podem dir que aquesta proteïna es troba en Eptesicus fuscus.
Després d’haver analitzat tota la informació anterior, es podria predir una estructura de la proteïna SelW2:
EXONERATE I GENEWISE:

Tornar a dalt
FAMÍLIA TR
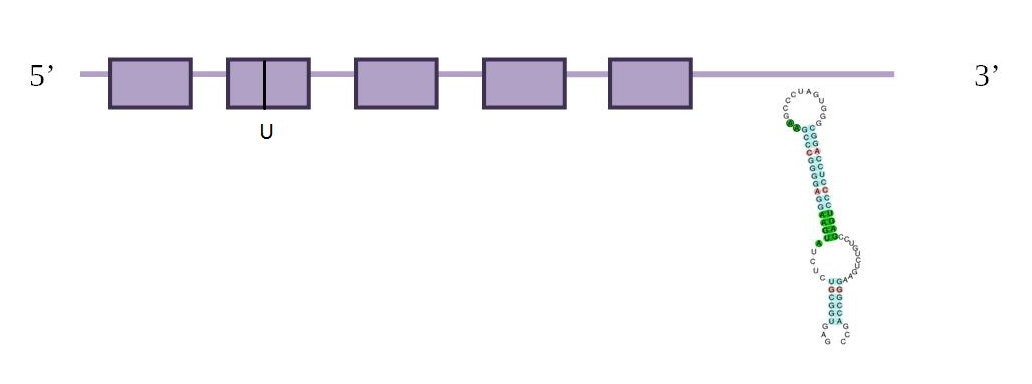
La família de selenoproteïnes TR (thioredoxin reductase) són oxidoreductases que usen NADPH per catalitzar la reducció de tioredoxina oxidat (Trx). La família té 3 membres: TR1, TR2 i TR3. A cada una d’elles a l’hora de fer el BLAST, vam obtenir diversos hits, la majoria dels quals estaven presents també als BLASTs de les altres proteïnes de la família. Mitjançant els valors obtinguts d’E-value, el percentatge d'identitat i la informació dels bits del BLAST hem pogut assignar un hit a cada una de les proteïnes de la família. A partir de les tres querys inicials s’han obtingut 5 seqüències, de manera que hi ha hagut una expansió d'aquesta família en el genoma d’Eptesicus fuscus.
SelTR1
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
Aquí ens trobem amb la proteïna TR1 (thioredoxin reductase), que es troba al scaffold JH977638.1, a la regió que va del 9351030 fins a 9280314 i està codificada en el sentit invers (reverse). La predicció d’Exonerate té 97417 nucleòtids, que inclouen 17 exons i 16 introns i un raw score de 2857, mentre que la predicció de Genewise té 97410 nucleòtids, que inclouen 17 exons i 16 introns i un score de 1087,2 bits. En aquest cas, les prediccions són quasi idèntiques, a excepció que l’Exonerate dóna una predicció més llarga per tan sols 7 nucleòtids de més i es pot veure que els dos mètodes emprats donen uns bons alineaments del T-Coffee corresponent (score de 99). La proteïna d'Homo sapiens té una selenocisteïna i hem observat que aquesta es troba conservada a l'últim exó de la proteïna predita per l'Exonerate, però no en la predita per Genewise, ja que s'acaba en un aminoàcid anterior.
Una hipòtesi que ens plantegem podria ser que el Genewise considerés el codó TGA com a codó STOP i per tant, acabi la predicció i no es detecti la selenocisteïna. Finalment, considerem que aquesta selenoproteïna té la selenocisteïna conservada. El programa SeciSearch ha trobat un possible element Seci a l’extrem 3’ de la cadena complementària. En quant el programa Seblastian, també prediu un Seci. La distància entre la selenoproteïna i l'element Seci és de 232 nucleòtids, mentre que la distància entre la selenocisteïna i l'element Seci és de 235 nucleòtids. També ens prediu una selenoproteïna i això ajuda a confirmar que aquesta selenoproteïna està present en Eptesicus fuscus. Després d’haver analitzat tota la informació anterior, es podria predir una estructura de la proteïna Tr1:
EXONERATE I GENEWISE

SelTR2
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
Aquí ens trobem amb la proteïna TR2 (thioredoxin reductase 2) que es troba al scaffold JH977628.1, a la regió que va des de 12804028 fins a 12843226 (BLAST) i està codificada en el mateix sentit que l’anotació (forward). La predicció d’Exonerate té 79454 nucleòtids, que inclou 19 exons i 18 introns i un raw score de 2019, mentre que la predicció de Genewise té 41986 nucleòtids, que inclou 14 exons i 13 introns i un score de 723,9 bits. En aquest cas, l’Exonerate cobreix una regió superior a l’inici en comparació amb Genewise. A l’hora d’analitzar els T-Coffees s’ha vist que en els dos casos hi ha hagut regions que faltaven, ja que podria ser que aquestes regions estiguessin localitzades en altres scaffolds, i per tant, no s’han pogut localitzar. El que també podria haver passat és que haguéssim escollit erròniament el hit. La selenoproteïna d’Homo sapiens té una selenocisteïna que s’ha conservat segons el mètode d’Exonerate a l’últim exó (al 19). No hem pogut observar-la en el programa Genewise ja que la predicció acaba aminoàcids abans.
El que hem observat és que una alanina de la query ha mutat a un stop codon (TGA). El programa SeciSearch ha trobat un possible element Seci a l’extrem 3’ de la cadena forward del gen, amb una energia lliure molt bona (-29). En quant el programa Seblastian, també prediu un element Seci a 1215 nucleòtids del final de la selenoproteïna i a 1218 nucleòtids de Sec-UGA. A més, ha predit una selenoproteïna, per tant, és una prova més que realment existeix aquesta selenoproteïna a E.fuscus. Després d’haver analitzat tota la informació anterior, es podria predir una estructura de la proteïna Tr2:
EXONERATE:


SelTR3
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
Aquí ens trobem amb la proteïna TR3 (thioredoxin reductase 3) que es troba al scaffold JH977646.1, que va de la regió 6790803 fins a la regió 6790955 i està codificada en el mateix sentit que l’ anotació (forward).
La predicció d’Exonerate té 183347 nucleòtids, que inclouen 4 exons i 3 introns i un raw score de 130, mentre que la predicció de Genewise té 155 nucleòtids, que inclou 1 exó i un score de 51,73 bits. Observem que la proteïna d’Homo sapiens té una selenocisteïna, però nosaltres no l’hem detectat, ja que les dues prediccions fetes acaben varis aminoàcids abans (3 en el T-Coffee d’Exonerate i 15 en el T-Coffee de Genewise). Per tant, no podem saber si es tracta d’una selenoproteïna o no.
El programa SeciSearch ha trobat un possible element Secis a la cadena complementària del gen, per tant, això donaria suport a la hipòtesi que no es tracta d’una selenoproteïna ja que l’element predit es troba a la cadena reverse. En quant el programa Seblastian, no ha predit cap selenoproteïna, per tant, això donaria suport a la nostra hipòtesi que aquesta selenoproteïna no es troba en E.fuscus. L’única manera de resoldre les relacions entre els membres de la família és fer un arbre filogènetic.
A l’haver fet la filogènia (veure arxiu) de la família TR en front les proteïnes hem pogut observar que hem escollit erròniament el hit d'aquesta selenoproteïna i que si haguéssim escollit el hit trobat a l’scaffold JH977615.1, llavors hauríem tingut uns resultats millors, ja que trobaria aquesta selenoproteïna en E.fuscus ja que s'ha conservat. Hem escollit el hit erròniament, ja que la selenoproteïna humana tenia una longitud molt curta i la longitud repercuteix substancialment en el valor d’E-value, obtenint valors menys significatius i per tant, obtenint pitjors valors en front de les altres proteïnes de la família.

Després d’haver analitzat tota la informació anterior, es podria predir una estructura de la proteïna Tr3:
MAQUINÀRIA
eEFSec
eEFSec és un eukaryotic elongation factor que permet la síntesi de selenoproteïnes.
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
Aquí ens trobem amb la proteïna eEFSEC (Eukaryotic elongation factor) que es troba al scaffold JH977615.1, el qual es troba situat a la regió 16286082 fins 16057229 del genoma. Està codificada en el sentit invers (reverse).
Només hem obtingut la predicció amb el programa Exonerate del primer scaffold, que té 228889 nucleòtids, que inclouen set exons i sis introns i un raw score de 2743. A l’hora de fer els Secis, no se n’ha trobat cap, ja que al no tractar-se d’una selenoproteïna, no esperem trobar-hi ni Secis ni selenocisteïnes. A l’utilitzar el programa Seblastian òbviament no hem trobat cap element Seci ni selenoproteïna, corroborant-nos definitivament el que haviem dit.
Després d’haver analitzat tota la informació anterior, es podria predir una estructura de la proteïna eEFSEC:
PSTK
La PSTK és una fosfoseril-tRNA kinasa localitzada al cromosoma 10.
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
El Blast de la proteïna PSTK dóna un únic hit significatiu (E value superior o igual a 10-5) el qual es troba a l'scaffold JH977742.1, de la posició 3421004 a la 3419639 i està codificada en el sentit revers (-). Les dades obtingudes de SelenoDB de la PSTK humana a partir de la qual hem fet l’anàlisi després del corresponent T-Coffee té 6 exons i 5 introns. Com que no és una selenoproteïna, no conté l’aminoàcid selenocisteïna.
A més, veiem que en l'Exonerate tenim una regió amb Ns i Unknowns al genoma d'Eptesicus fuscus, fet que indica que aquesta regió del genoma està mal anotada i, per tant, no hi ha res que es correspongui a la query. És per això que el T-Coffee d'Exonerate no és informatiu, ja que no ens ha permès identificar la PSTK en el genoma d'Eptesicus fuscus. Pel que fa al T-Coffee de Genewise, veiem que a la part central sí que hi ha solapament, tot i que no a la zona inicial i final (això és així perquè la seqüència de Genewise acaba més tard i acaba més aviat que la query). Com que no estàvem segurs dels resultats del T-Coffee vam realitzar un BlastP (veure arxiu) de la PSTK d’Eptesicus fuscus sobre les seqüències disponibles a NCBI, el qual ens mostra homologia de PSTK amb espècies relacionades amb Eptesicus fuscus, com és el cas de Myotis lucifugus.
Tot i això, no hi ha una perfecta homologia, motiu pel qual serien necessaris més estudis per determinar la presència o absència de PSTK a Eptesicus fuscus. No detectem cap selenocisteïna, ja que com en el cas de la PSTK humana, conté cisteïna enlloc de la selenocisteïna. A més, no hi ha cap element Seci possible (a la taula no mostrem cap valor d'element Seci perquè no es trobaven a la cadena corresponent de PSTK; resultats del web Seblastian (veure arxiu)). Per tant, amb les dades que tenim no podem predir la PSTK en el genoma d'Eptesicus fuscus.
SBP2
El gen de la SBP2 (SECIS binding protein 2) codifica per a una proteïna nuclear que funciona com a proteïna d’unió Secis. Les mutacions en aquest gen s’han associat amb una reducció en l’activitat d’un enzim que conté selenocisteïna i un metabolisme anormal de l’hormona tiroidea.
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
La proteïna SBP2 es troba al scaffold JH977714.1, a la regió que va de 4329006 a 4362476 (BLAST) i està codificada en el mateix sentit que l'anotació (forward). La predicció d’Exonerate té 18184 nucleòtids, que inclouen deu exons i nou introns i un raw score de 1528; mentre que la predicció de Genewise té 19414 nucleòtids, que inclouen nou exons i vuit introns i un score de 518.13 bits. En aquest cas es pot veure que entre els dos mètodes emprats hi ha una diferència de 1230 nucleòtids. Els dos mètodes utilitzats donen uns bons alineaments dels T-Coffee corresponents (score de 98 i 99 corresponent). Hem observat que igual que en Homo sapiens, aquest SECIS binding protein es troba conservat en Eptesicus fuscus.
El programa SeciSearch no ha trobat cap SECI, ja que no és una selenoproteïna. Tal i com esperàvem, SeciSearch3 tampoc va trobar Secis elements i Seblastian tampoc va poder predir cap selenoproteïna. Després d’haver analitzat tota la informació anterior, es podria predir una estructura de la proteïna SBP2:
EXONERATE:
GENEWISE:
Tornar a dalt


SECp43
SECp43 és la “tRNA Sec 1 associated protein 1”.
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
El blast de la proteïna SECp43 dóna 7 hits significatius (E value superior o igual a 10-5), que ordenats de millor a menor E-value es troben en els següents scaffolds: JH977655.1, JH977612.1, JH977645.1, JH977970.1, JH977753.1, JH977842.1 i JH977735.1. A SelenoDB, veiem que la SECp43 humana (seqüència que hem usat com a query per analitzar el genoma d'Eptesicus fuscus després d’haver realitzat el T-Coffee) no és selenocisteïna (té cisteïna) i té 9 exons i 8 introns. A més, al no tenir cap selenocisteïna, no és una selenoproteïna. Per tant, per determinar quins són els millors hits, compararem el que més s'assembla als resultats disponibles a SelenoDB. Només hi ha dos hits que coincideixin amb els valors de 9 exons i 8 introns, els quals són JH977655.1 i JH977645.1. Tot i això, considerem millor el primer hit, ja que el Genewise també dóna el mateix resultat de 9 exons i 8 introns, mentre que el Genewise del segon hit només detecta 1 exó.
El hit escollit es transcriu en direcció reversa (-) i va de la posició 1893320 a la 1881131. La predicció d'Exonerate pel hit que hem seleccionat dóna un total de 12946 nucleòtids, incloent, com ja hem dit, 9 exons i 8 introns (igual que la informació disponible a SelenoDB) i un raw score de 1492. Per altra banda, la predicció de Genewise dóna un total de 12945 nucleòtids, també amb 9 exons i 8 introns, amb un score de 595,07 bits. Per tant, tant Exonerate com Genewise prediuen la mateixa proteïna, ja que només difereixen per un nucleòtid i presenten la mateixa estructura exònica i intrònica.
A més, analitzant tant el T-Coffee de l'Exonerate com el del Genewise, veiem que tots dos donen scores informatius (100), de manera que SECp43 s'ha trobat al genoma d'Eptesicus fuscus. Tal i com la SECp43 humana usada com a query, a Eptesicus fuscus conté una cisteïna enlloc de selenocisteïna, motiu pel qual tampoc no hem pogut predir elements Secis reals (es troben bé a l'extrem 5' o dins de la seqüència predita i en cap cas a l'extrem 3' com cabria esperar tal i com podem comprovar a la taula. No hi ha disponible cap link perquè directament el web ha dit que no detectava elements Secis).

SecS
SecS és una selenocisteïna sintasa.
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
Al fer el Blast, només hem trobat un hit significatiu (E-value major o igual a 10-5) per SecS, el qual es troba a l'scaffold JH977622.1, entre les posicions 5606690 i 5642192 i codificada en sentit revers (-). A SelenoDB, veiem que la SecS humana no és una selenoproteïna (ja que té una cisteïna enlloc de selenocisteïna) (query que hem usat per fer els anàlisis a Eptescius fuscus després d’analitzar els resultats del T-Coffee):té 11 exons i 10 introns.
El resultat de l'Exonerate dóna un total de 35915 nucleòtids, amb 11 exons i 10 introns i un raw score de 2271. Per altra banda, el resultat del Genewise dóna un total de 35914 nucleòtids, amb 11 exons i 10 introns i un score de 938,64. Per tant, tant Exonerate com Genewise reconeixen la mateixa proteïna, només diferint per un nucleòtid i presentant la mateixa estructura exònica i intrònica. Analitzant tant el T-Coffee d'Exonerate com el de Genewise, obtenim resultats significatius (score de 99), de manera que SecS s'ha identificat al genoma d'Eptesicus fuscus. Tal i com a la SecS humana usada com a query, a Eptesicus fuscus hi ha una cisteïna, motiu pel qual tampoc hem pogut predir elements Secis reals (no mostrem els resultats a la taula perquè estaven a la cadena +, mentre que el nostre hit es troba a la cadena -).
SPS2
(En aquest treball hem considerat SPS2 com a maquinària però segons la literatura també es pot considerar una selenoproteïna).
Vam agafar SPS2 d'Homo sapiens ja que la selenoproteïna començava per metionina i tenia dues selenocisteïnes.
En aquesta taula es pot observar a quin scaffold es troba la proteïna, en quina direcció es transcriu respecte l’anotació, les coordenades de la proteïna predites per l’Exonerate, l’score del T-Coffee, Genewise i el seu respectiu score de T-Coffee i finalment els SECIS predits.
Vam obtenir diferents hits i utilitzant els valors d’E-value i la informació dels bits del BLAST vam poder assignar un hit. La proteïna SPS2 es troba al scaffold JH977617.1, en la regió que va de 215348 a 215937 (BLAST) i està codificada en el mateix sentit que l’anotació (forward).
La predicció d’Exonerate té 970 nucleòtids, que inclou un exó i un raw score de 1135; mentre que la predicció de Genewise té 422 nucleòtids, que inclou un exó i un score de 541.81 bits. Segons la literatura, això seria el que esperaríem trobar ja que SPS2b prové d'una pèrdua exònica de SPS2a, la qual és multiexònica. SPS2b és una selenoproteïna amb un únic exó, apareguda a partir de la pèrdua exònica de SPS2a, la qual era multiexònica.
La selenoproteïna d'Homo sapiens té dues selenocisteïnes, mentre que no les hem pogut trobar en l' Exonerate. En canvi, sí que hem trobat totes dues en el Genewise. No hem obtingut cap Seci, mitjançant el programa SeciSearch3 i el programa Seblastian tampoc ens ha predit una selenoproteïna.
Per tant, es necessiratien més anàlisis per corroborar la presència de SPS2 en Eptesicus fuscus.
Després d’haver analitzat tota la informació anterior, es podria predir una estructura de la proteïna sps2: