INTRODUCCIÓ
El seleni és un micronutrient essencial per a moltes formes de vida tant en bacteris com en arqueobacteris i eucariotes. No obstant, no es troba mai en forma lliure (degut a la seva toxicitat) sinó que el trobem conjugat dins de les selenoproteïnes. El dèficit de seleni comporta la malaltia de Keshan mentre que el seu excés és tòxic i genera selenosi.
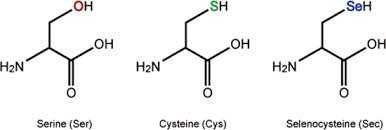
Així doncs, en els éssers vius el seleni resideix dins les cèŀlules formant part de l'aminoàcid selenocisteïna (SeCys). La selenocisteïna, l'aminoàcid número 21, està codificat pel codó TGA i es representa amb la lletra U. La selenocisteïna té la mateixa estructura que una cisteïna (Cys), però s'ha substituït el radical de sofre (SH) per un de seleni (SeH). El seleni i el sofre tenen propietats químiques semblants, i per això es poden trobar proteïnes homòlogues a les selenoproteïnes que en comptes de tenir una selenocisteïna contenen cisteïna. [1]
En mamífers, les selenoproteïnes acostumen a ser enzims redox, implicats en reaccions d'oxidació-reducció, gràcies a l'alta reactivitat de l'àtom de seleni situat al centre catalític, i per tant, tenen una possible capacitat protectora antioxidant i anticancerígena. [2]
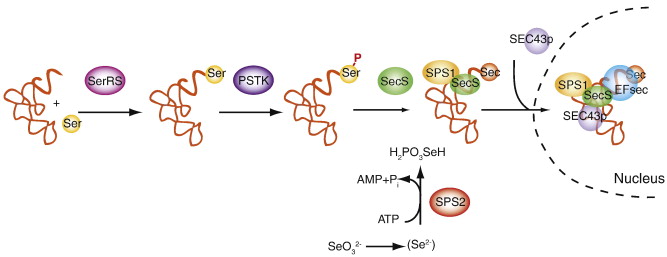
La selenocisteïna no és un aminoàcid lliure sinó que la seva biosíntesi parteix de l'addició d'una serina al tRNAsec específic, gràcies a la Seryl-tRNA sintetasa (SerRS). Tot seguit, el residu incorporat es fosforila per l'acció de la O-fosforil tRNAsec quinasa (PSTK). Després, la selenocisteïna sintasa (SecS) converteix el Ser-tRNAsec a Sec-tRNAsec, fent servir com a substrat per a aquesta reacció el monoselenofosfat (H2PO3SeH). Aquest substrat s'obté gràcies a l'acció de la selenofosfat sintetasa 2 (SPS2). Per últim, s'ha comprovat que la SECp43 pot direccionar tot el complex (SPS1, SecS i Sec-tRNAsec) cap al nucli. [1]
No obstant, els bacteris no segueixen aquesta via de síntesi, sinó que simplement converteixen la serina a selenocisteïna en el tRNAser.
El codó TGA – que codifica per la selenocisteïna – és també un codó d'STOP. Per aquest motiu, la maquinària de traducció del DNA ha de tenir un element diferencial que permeti discernir entre aquestes dues situacions. Aquest element s'anomena SECIS (SelenoCysteine Insertion Sequence) i és un loop d'RNA que resideix al 3'UTR dels mRNAs de les selenoproteïnes que impedeix que el codó TGA sigui reconegut com a un codó d'STOP, evitant així la terminació de la traducció.
La seqüència dels SECIS està molt poc conservada, però tot i així en destaquen quatre parells de bases consecutius (coneguts com “el quartet”) que són essencials per la incorporació de les selenocisteïnes in vivo. Contràriament, l'estructura secundària dels SECIS està molt conservada - només varia la part apical del loop - i per això s'ha usat diverses vegades com un element de cerca de selenoproteïnes. [3]
El SECIS és necessari per a la incorporació de la selenocisteïna però cal remarcar que la seva presència no porta necessàriament a la seva incorporació.
Aquest element té com a funció reclutar la proteïna SECIS binding protein (SBP2) per tal que aquesta s'uneixi a un factor d’elongació específic de selenoproteïnes (eEFsec). Llavors, eEFsec s'encarrega d'apropar el tRNA específic amb la selenocisteïna (tRNAsec) al mRNA i així es continuï la traducció. [3]
Filogènia de les selenoproteïnes
Les selenoproteïnes estan presents en els tres regnes (Prokarya, Archea i Eukarya) però no totes les espècies n'utilitzen. Per exemple, els llevats i les plantes han perdut la maquinària d'inserció de les selenoproteïnes durant l'evolució. Per altra banda, s'ha observat que els organismes aquàtics compten amb un major nombre de selenoproteïnes que no pas les espècies terrestres.
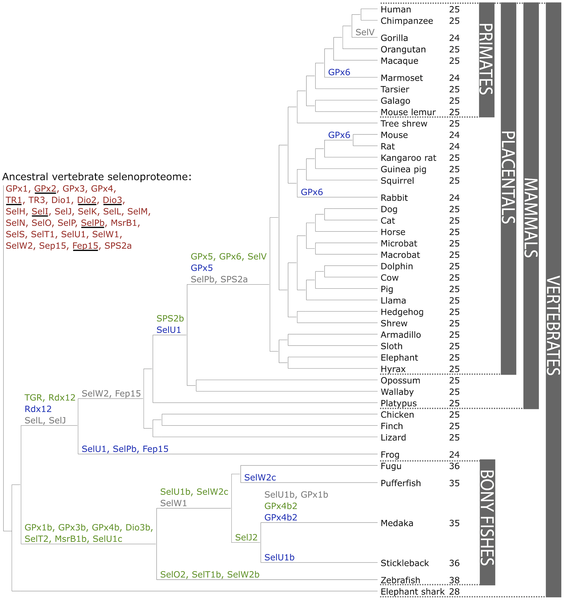
Així, s'han comptabilitzat fins a 45 selenoproteïnes diferents en vertebrats, però mai totes es trobaven en una sola espècie. Les espècies de vertebrats amb més selenoproteïnes són els teleostis, d'entre els quals destaca el peix zebra, amb un total de 38. Per altra banda, s'han trobat 28 selenoproteïnes en l'ancestre dels vertebrats i 25 en l'ancestre dels mamífers així com en humans.
Per tant, hi ha hagut una reducció de les selenoproteïnes durant l'evolució (sobretot en el moment de colonitzar la superfície terrestre) i s'ha apuntat que el gran nombre d'aquestes proteïnes en els peixos ossis és degut a la duplicació que van patir els genomes d'aquest clade. [4]
L'espècie Chelonia mydas és una tortuga marina herbívora de grans dimensions que habita en les aigües del Carib i del Pacífic. Actualment està reconeguda com a espècie en perill d'extinció per la Unió Internacional per a la Conservació de la Natura (IUCN) degut a l'activitat humana i a una disminució del seu hàbitat natural.
La tortuga verda marina rep aquest nom pel color verdós de la seva closca. és una de les espècies de tortugues marines més grans i poden arribar a pesar uns 250 kg i viure uns 75 anys. Diferenciem les dues subespècies Chelonia mydas mydas i Chelonia mydas agassizii.
Hàbitat: La tortuga verda marina habita en aigües tropicals arreu del món i les femelles només abandonen l’aigua en el moment de pondre ous.
Reproducció: L’aparellament succeeix a dins el mar cada 3-6 anys i després de la còpula la femella anirà a pondre els ous a les platges (on ella mateixa va néixer) i farà un forat durant hores on hi dipositarà uns 200 ous. Finalment taparà el forat i tornarà al mar. Després d’un període de 40 a 72 dies (depèn de la localitat) naixeran les cries, que hauran d’espavilar-se soles.
Alimentació: Les tortugues verdes són principalment marines tot i que en edat juvenil també s’alimenten de meduses, cargols, crancs i cucs [5].
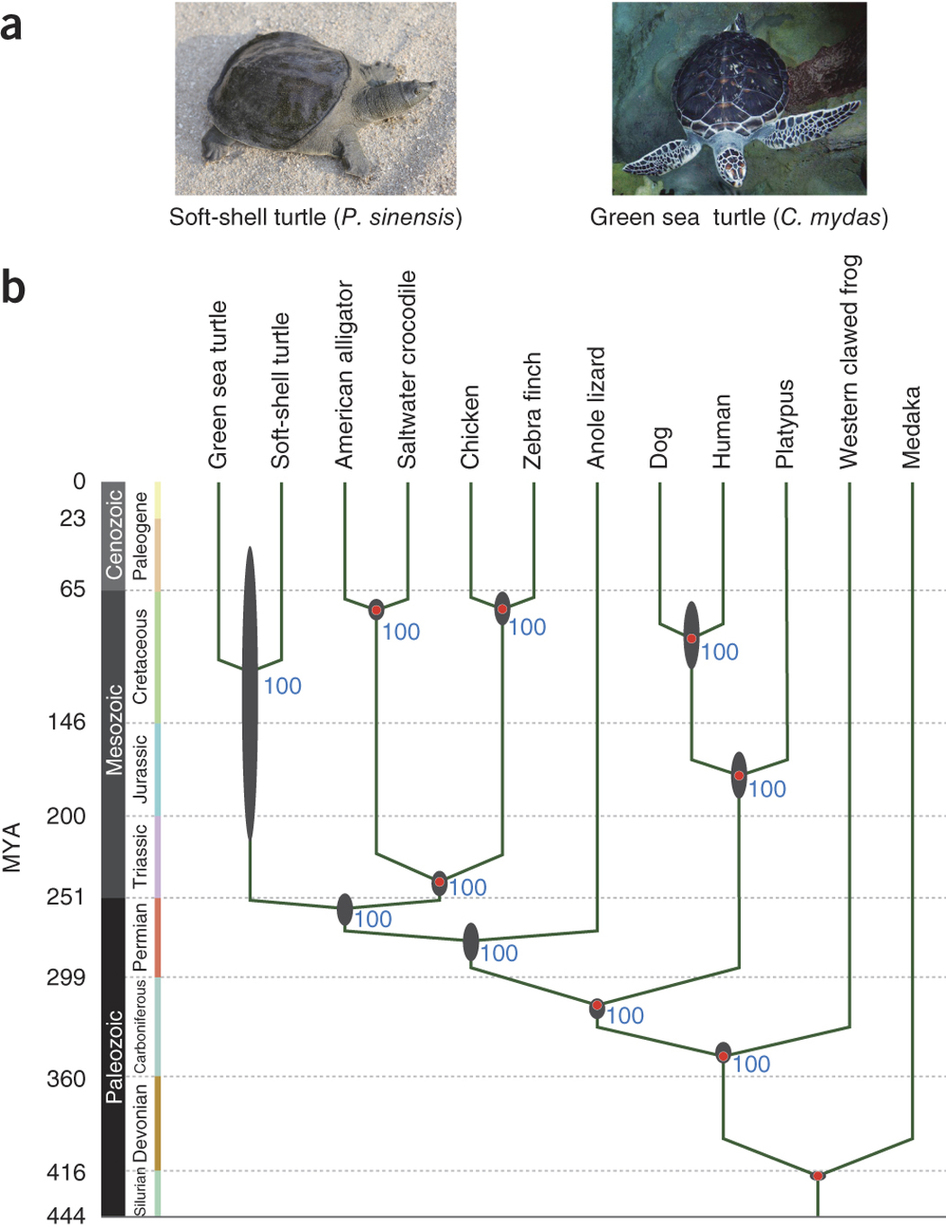
L’origen evolutiu de les tortugues ha estat àmpliament debatut donades les seves característiques úniques en la natura (com per exemple la disposició de l’escàpula, que és diferent a la d’altres tetràpodes amb closca). No obstant, sembla clar que pertanyen a un llinatge – anomenat anàpsids – que va divergir dels rèptils molt d’hora en l’evolució (fa 251 milions d’anys) però és proper evolutivament al clade de les serps i les sargantanes (Lepidosauria). A més a més, està relacionat amb el llinatge dels cocodrils i dels ocells. [6]
Tria dels genomes de referència
Com a genomes de referència, s’han triat l’Anole lizard (Anolis carolinensis) i la Soft-shell turtle (Pelodiscus sinensis).
La Soft-shell turtle és l’espècie (disponible a la base de dades SelenoDB) més propera filogenèticament a Chelonia mydas. Per altra banda, es va optar per l’Anole lizard per ser una espècie llunyana però certament emparentada amb Chelonia mydas. A més, per tal de tenir tot el ventall del selenoproteoma a l'abast i poder així identificar totes les selenoproteïnes possibles en el genoma problema, s'ha obtat per seleccionar queries també d'Homo sapiens, Danio rerio i Xenopus tropicalis.