Discussió
Família GPx
Les Glutatió Peroxidases són la principal defensa enzimàtica antioxidant en humans ja que participen en l'eliminació del peròxid d'hidrogen, modulen la síntesi d'eicosanoides modificant la inflamació i disminueixen el dany oxidatiu de lípids, lipoproteïnes i DNA. S'han descrit vuit proteïnes homòlogues per aquesta família les quals totes són selenoproteïnes, a excepció de GPx5, GPx7 i GPx8 que són homòlogues en Cisteïna).
La localització de cadascuna s'indica a continuació:
Durant l'anàlisi d'aquesta família de selenoproteïnes, es va observar que el nom de la selenoproteïna no es conserva segons la espècie. És per això que es va realitzar un alineament múltiple amb el programa T-coffee, observant que proteïnes amb el mateix nom, diferien en la seva seqüència entre diferents espècies. En aquesta imatge podeu apreciar l'alineament múltiple de les selenoproteïnes GPx d'humà i pollastre:
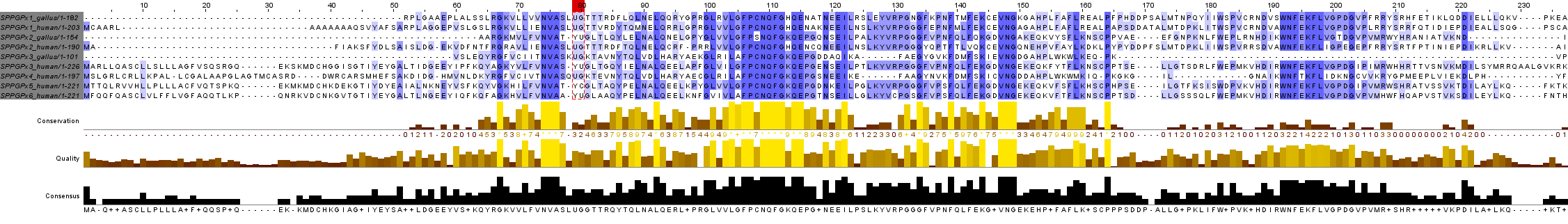
Observat aquest fet, es va decidir extrapolar els resultats a partir de la nomenclatura de les selenoproteïnes GPx humanes, ja que tenien les 8 GPx fins ara descrites ben anotades i provenien d'una base de dades fiable (SelenoDB). A més a més només es van obtenir 3 querys GPx de Gallus gallus.
Degut a que és una família de selenoproteïnes amb varis membres, amb un grau d'homologia molt elevat, va resultar complicat decidir quina era la posició exacta en el genoma de cadascuna per separat. Es van observar molts hits significatius del blast que es repetien en l'anàlisi individual de cadascun dels membres. Tot i així, van servir d'ajuda els resultats predits pel programa Selenoprofiles per escollir la regió genòmica més plausible. Es va trobar un hit significatiu per les 4 primeres selenoproteïnes (GPx1, 2, 3 i 4). En canvi, les GPx5 i 6 mostraven un hit significatiu en la mateixa posició del mateix scaffold que GPx3. S'ha estudiat l'origen d'aquestes dues selenoproteïnes, GPx5 i 6, sent les GPxs que han evolucionat més recentment, i es creu que apareixen com a resultat d'una duplicació en tàndem de la GPx3 en l'arrel dels mamífers placentaris. A més, no s'ha descobert cap forma de GPx5 que presenti una selenocisteïna en la seva seqüència peptídica [4]. Aquests descobriments reforcen la nostra idea, de tal manera que es creu que l'anotació en el genoma de la selenoproteïna predita correspon a la GPx3 i les GPx5 i 6 no estan presents en el genoma de C. picta bellii.
Cal afegir que s'ha aconseguit anotar els dos homòlegs en cisteïna, GPx7 i 8, en el genoma de la tortuga. Aquests dos homòlegs han evolucionat d'una selenoproteïna GPx4 ancestral abans de la separació entre mamífers i peixos [4]. D'aquesta manera, la nostra anotació es veu reforçada per la història filogenètica d'aquestes proteïnes. Tot i així, el programa Selenoprofiles no dóna cap resultat del blast per GPx7 i 8 i no es pot saber si és perquè no troba un bon alineament o bé perquè no té querys per aquests homòlegs en la seva base de dades interna.
En quant a la recerca dels elements SECIs, se'n van trobar en les GPx1 i GPx2; així com també en la GPx8 (però a 8000 nucleòtids en posició 3' del gen). Per aquest motiu s'ha cregut que es trobava massa lluny del gen i no s'ha tingut en compte. Tot i no trobar elements SECIs en la GPx3 i GPx4, es creu que els resultats dels programes de predicció de selenoproteïnes són prou significatius i es consideren com a tal.
Tornar a daltFamília DI
La família DI (Iodotironina Deiodinases) està formada per una subfamília d'enzims que s'encarreguen de l'activació i la desactivació de les hormones tiroidees. Concretament, catalitzen la transformació del precursor T4 (tiroxina) en T3 (triiodetironina) activa. Actualment s'han descobert 3 enzims coneguts d'aquesta família en mamífers, anomenats: DI1, DI2 i DI3. Tots tres presenten una estructura terciària característica que correspon a un plegament en tiorredoxina necessària per la seva funció. Degut a aquest fet, presenten un elevat grau d'homologia entre ells. A més, s'hi afegeix el fet que s'ha trobat una duplicació de DI3 en els peixos osteïctis (Dio3b). Curiosament, tots els gens que codifiquen per DI3, en totes les espècies només contenen un exó i no presenten introns [17].
En la nostra recerca, es van localitzar els gens per les 3 proteïnes d'aquesta família en el genoma de C. picta bellii. Els resultats del programes de predicció van ser els mateixos tant per la query humana com per la de gall. En l'apartat de resultats es pot trobar la posició genòmica exacta que codifica per cada selenoproteïna. Les tres són selenocisteïnes però no es va identificar cap element SECIs en la DI3. En canvi, el programa Selenoprofiles sí que en prediu un a 327 nucleòtids de distància de l'última regió codificant. Per últim, el fet d'haver trobat només un exó en la DI3, tal com indiquen els estudis prèviament realitzats, reforça la nostra hipòtesi que en el genoma de C.picta bellii podem localitzar les tres selenoproteïnes DI.
Tornar a daltFamília Msr i SelR
Aquesta família està formada per enzims amb funció antioxidant que s'encarreguen de la reparació d'altres enzims que han patit pèrdua de funcionalitat degut a oxidacions de metionines en la seva seqüència, o per la desaparició dels ROS. Se'n coneixen dos, MsrA i MsrB. Aquestes proteïnes no comparteixen cap homologia ni en seqüència ni en estructura, per aquest motiu es creu que van aparèixer independentment en la història evolutiva [19].
L'enzim MsrA va ser el primer en ser descobert i és l'única proteïna coneguda en mamífers capaç de reduir el methionine-S-sulfoxide. També pot reduir altres molècules. És una selenoproteïna que es troba majoritàriament en organismes procariotes com les algues verdes i alguns bacteris, ja que contenen una Sec en el domini catalític en comptes d'una Cys. En canvi, la proteïna MsrA que es troba en mamífers ja conté cisteïnes en el domini catalític [20]. Per aquest motiu, es va comprovar si en Chrysemys picta bellii també hi era present. El resultat obtingut no mostren un alineament complet ja que, els primers 70 aminoàcids no apareixen en la proteïna resultant dels programes de predicció. A més, presenta introns molt grans. Ara bé, sí que es va aconseguir alinear la cisteïna característica del centre catalític, que es prediu també com una cisteïna. El programa proporciona un element SECIs en l'interior del gen, així que no es considera correcta aquesta predicció.
D'altra banda, el segon enzim d'aquesta família, MsrB, també conegut com SelR, presenta tres subproteïnes en humans: SelR1 (amb Sec en el centre catalític), R2(Cys), i R3(Cys). Altrament anomenades MsrB1, MsrB2 i MsrB3 respectivament. En Gallus gallus només es va obtenir una seqüència de SelR. Els resultats dels programes de predicció obtenen hits significatius per la SelR1, que es troba conservada com a selenoproteïna en el genoma de la tortuga (corresponent a SelR de gallus que també conté la selenocisteïna). Els resultats de SelR2 i R3 no mostren tanta homologia com els casos anteriors. Es podria dir que el gen de SelR3 sembla estar present en el genoma de C.picta. Tot i així, aquest correspon a una seqüència molt més curta que la humana. Es podria hipotetitzar que la proteïna s'estigui perdent o bé que mostri homologia amb un domini comú en aquesta família. En aquest cas, la seqüència predita de SelR3 no correspondria al gen d'aquesta proteïna realment. L'alineament observat pel programa t-coffee cobreix la cisteïna del centre catalític. En un altre scaffold es troba un resultat del blast significatiu per la primera part de la proteïna R3 humana, però els programes de predicció (Exonerate i Genewise) no proporcionen cap alineament.
Cal afegir que també es pot observar un element SECIs en la regió 3' del gen de la SelR1, i aquest és inexisteint en les seqüències de les proteïnes homòlegues en cisteïna. Podem concloure que la tortuga Chrysemys picta bellii conserva aquesta família de proteïnes en el seu genoma, tot i que sembla que MsrB hagi patit certs canvis respecte les proteïnes en mamífers.
Tornar a daltSel15/Sep15
La selenoproteïna 15, Sep 15 o Selenoproteïna 15 kDa és una selenoproteïna de funció desconeguda que es troba expressada principalment en ronyons, tiroides i paratiroides. Es coneix que Sel15 està implicada en la formació d'enllaços disulfurs i en el plegament de les proteïnes. El gen que codifica per aquesta proteïna consta de cincs exons i quatre introns [21].
S'han utilitzat diversos programes de predicció per confirmar els resultats. En tots ells s'han obtingut resultats molt diferents i no s'ha pogut obtenir una anotació evident. El programa Selenoprofiles predia un alineament amb la Sel15 de Bos taurus en la cadena directa de DNA, amb un element SECIs en 3' del gen però amb un intró de 33000 parells de bases. El nostre programa alineava la Selenocisteïna de la Sel15 humana amb una Glicina degut a que introduïa varis gaps en la seqüència i el programa Genewise no alineava la posició on es troba la Sec de la Sel15 humana. Per últim es van analitzar manualment la Sel15 de Tetraodron nigroviridis i es va obtenir l'alineament de la Sec amb un codó TGA en el genoma de la tortuga. Tot i així, predia un element SECIs en mig de la regió codificant per la proteïna. En tot cas, la regió del genoma de C.picta bellii on s'anota aquesta proteïna és el mateix, però les posicions varien en funció de la llargada de l'alineament. En conclusió, no es pot confirmar amb exactitud que la proteïna Sel15 estigui codificada en el genoma de Chrysemys picta bellii. Tornar a daltSelH
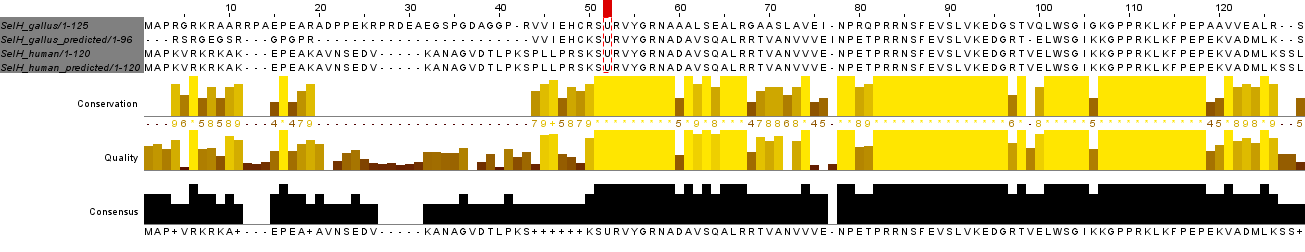
La SelH és una selenoproteïna que presenta un plegament de tioredoxina en la seva estructura terciària, conservant un motiu -CXXU- (correspon a una cisteïna separada per dos altres residus i una selenocisteïna a continuació). El motiu -CXXC- és comú en la família de les tioredoxines, suggerint que aquesta selenoproteïna té una funció d'oxidoreductasa nuclear [22].
Tots els anàlisis realitzats per anotar la SelH en el genoma de Chrysemys picta bellii proporcionen els mateixos resultats. Es van utilitzar com a querys les SelH humana, de pollastre i la de cavall (aquesta última es va obtenir del programa Selenoprofiles). Després dels anàlisis es pot afirmar que la SelH està codificada en el genoma de la tortuga com a selenoproteïna, conservant el motiu -CXXU- i l'element SECIs en posició 3' del gen predit pel programa Selenoprofiles. El nostre programa no va ser capaç de predir cap element SECIs però sí que mostra un alineament múltiple altament homòleg entre la predicció i les querys.
Tornar a dalt
SelI
La Selenoproteïna I és una de les últimes selenoproteïnes estudiades. Curiosament, conté un domini CDP-alcohol fosfatidiltransferasa altament conservat, que es troba de forma habitual en els enzims colina fosfotransferases (CHPT1). Aquesta selenoproteïna conserva els dominis transmembrana corresponents a les topologies CHPT1 i CEPT1, i regions crítiques entre segments transmembrana que contenen 3 àcids aspàrtics essencials per la seva funció.[4] Vegeu la següent imatge:

La principal diferència entre SelI i les seves homòlogues és l'extensió C-terminal que conté la Selenocisteïna (Sec). Precisament, un cop obtingudes les dades definitives, tant els alineaments aconseguits per Homo sapiens com per Gallus gallus (amb scores en T-coffe de 100), contenen l'extenció C-terminal característica. Segons les prediccions d'Exonerate, T-coffe i Genewise (a excepció de la query humana d'aquest últim), sembla indicar que SelI és una selenoproteïna present en el genoma de Chrysemys picta belli. Tot i així, no apareix cap element SECIs. Aquest fet no descartaria la possibilitat de que fos una selenoproteïna, ja que com hem esmentat, el genoma de Chrysemys picta belli no es troba totalment ensamblat i l'element SECIs podria localitzar-se en una regió encara no anotada del genoma. Els resultats de Selenoprofiles semblen recolzar la nostra hipòtesi.
Tornar a daltSelK
La família de selenoproteïnes K (SelK) es troba en nombroses espècies d'eucariotes superiors. Té una mida d'uns 95 aminoàcids i està formada per proteïnes transmembrana que també es poden localitzar al reticle endoplasmàtic. En funció de l'espècie, la proteïna compleix diferents funcions: s'està investigant el seu paper en la resposta oxidativa a l'estrés del reticle endoplasmàtic [23].
Es va analitzar la presència d'aquesta selenoproteïna en el genoma de la tortuga a partir de dues querys diferents: la humana i la de pollastre. Els resultats obtinguts a partir de la query de pollastre són bastant significatius i semblen indicar que el genoma de la tortuga codifica per la SelK com a selenoproteïna d'uns 90aa. Però, en contraposició a la nostra hipòtesi, no es va trobar cap element SECIs en la regió 3' del gen predit. En canvi, sí que se'n va localitzar un en la cadena complementària i a més, a una distància de molts parells de bases downstream de la regió 3' del gen.
Tornar a daltSelM
De la mateixa manera que Sel15, aquesta selenoproteïna forma part de la família de tiol-disulfit oxidoreductases. També presenta el plegament constituït per una làmina β central rodejada per α-hèlixs, característic de les tiorredoxines i els motiu Cys-X-X-Sec.
Es va trobar una regió del genoma de la tortuga que codifica per aquesta selenoproteïna on es conserva el motiu Cys-X-X-Sec. Els resultats són molt significatius, amb puntuacions molt elevades en els programes de predicció utilitzats (Exonerate i Genewise). A més, també coincideix la predicció del nostre programa automatitzat amb la del programa Selenoprofiles. La proteïna predita conté 5 exons i uns 128 aminoàcids aproximadament ja que no comença amb metionina. L'alineament que ens mostra en totes les querys utilitzades d'humà, gallus i gos comença per l'aminoàcid 5 de la proteïna de referència. A més, en tots els casos, s'ha obtingut la predicció d'un element SECIs a uns 300 aminoàcids de la regió codificant en posició 3'.
Tornar a daltSelN
Aquesta selenoproteïna està formada per dues isoformes. Aquestes, s'expressen en múscul esquelètic, cervell, pulmó i placenta, i a més, la isoforma 2 és expressada també en cor, diafragma i estómac. El gen que codifica per aquesta selenoproteïna s'anomena SEPN1 i els seus defectes causen la distròfia congènita muscular amb rigidesa espinal (RSMD1), un desordre muscular. Mentre que, quan la proteïna està mutada apareixen problemes respiratoris.
Es va analitzar aquesta proteïna utilitzant dues querys: una procedent d'Homo sapiens i l'altra de Gallus gallus. Es pot observar com la proteïna humana conté dues U en la seva seqüència (en les posicions 127 i 462). En canvi, la de pollastre només presenta l'última U situada en la posició 403 de la seva seqüència. En ambdós casos la proteïna predita conserva la U i es troba en la mateixa regió i posició genòmica sense considerar la part inicial de la proteïna. En la proteïna obtinguda a partir de la query humana es va obtenir el primer intró de 18000 nucleòtids, fet que fa variar els valors inicials de localització en el genoma.
Cal comentar que la proteïna predita per Exonerate presenta una glicina (Gly) en l'aminoàcid 127 que corresponia a una Sec en la SelN humana. Contràriament el programa Genewise prediu una leucina (Leu) en la mateixa posició. Creiem que pot ser degut a que ambdós programes introdueixen gaps per obtenir el millor alineament i poden modificar el marc de lectura.
Tornar a daltSelO
SelO és una selenoproteïna molt gran formada per 669 aminoàcids en humans i es troba àmpliament distribuïda en animals, bacteris, llevats i plantes. Els homòlegs de SelO en vertebrats tenen una selenocisteïna localitzada en la penúltima posició de l'extrem C-terminal. En proteïnes homòlogues de SelO d'altres organismes, la selenocisteïna és substituïda per una cisteïna. Una publicació recent intenta aclarir la funció d'aquesta selenoproteïna, proposant que SelO adopta un plegament tridimensional semblant al plegament de les proteïnes quinases. A més, també es creu que conserva l'activitat fosfotransferasa tot i no conservar el residu d'aspàrtic en el motiu His-Arg-Asp en el domini catalític. Per últim, es discuteix una possible activitat oxidoreductasa[24].
Es creu que aquesta selenoproteïna està conservada en el genoma de Chrysemys picta bellii ja que els anàlisis de predicció realitzats són altament significatius. Es prediu una proteïna de 638 aminoàcids aproximadament (no es mostra l'alineament dels primers 20 aminoàcids) que conserva la Sec en l'avantpenúltima posició. A més, el programa Selenoprofiles prediu un element SECIs a 306 nuclèotids downstream del gen.
Tornar a daltSelP
La selenoproteïna P és una glicoproteïna extracel.lular que es troba en abundància en el plasma, unida a les cèl.lules endotelials. En humans consta de 381 aminoàcids i conté 10 residus de selenocisteïna, els quals es troben distribuïts en dos dominis: N-terminal (domini enzimàtic que conté una Sec) i C-terminal (domini de transport amb 9 Secs). El seu gen consta de cinc exons. S'ha estudiat la seva funció en l'homeòstasi del seleni i en la defensa oxidant en l'espai extracel.lular. A més, també s'ha vist que és una proteïna que s'uneix a heparina i a metalls pesants com el mercuri. Es creu que la selenoproteïna P només s'expressa en vertebrats.[21]
Es poden distingir dues variants d'aquestes selenoproteïnes, la SelP i SelPb. En el cas de la SelP (o SelPa) es va identificar el gen que codifica per aquesta selenoproteïna en el genoma de la tortuga i un element SECIs a 280 nucleòtids downstream d'aquest gen, predit per Selenoprofiles. Es pot observar com manté el codó TGA que codifica per Sec en la mateixa posició que les querys analitzades (humana i pollastre). El programa automatitzat també prediu un segon element SECIs però es localitza més allunyat d'aquesta regió. A més, es va observar que la longitut de la proteïna resultant pels programes de predicció diferia segons la query utilitzada. Per aquest motiu, es va realitzar un alineament múltiple per comprovar si entre les querys també variava molt la longitut de la seqüència peptídica.

En aquesta figura s'observa com la longitut proteica varia tot i que hi ha un domini molt conservat on es troba la Sec en tots els casos.
D'altra banda, el genoma de la tortuga també codifica per la SelPb, que fins al moment no s'ha observat en mamífers placentaris. Tot i que la longitut de les querys utilitzades (Takifugu rubripes, Danio rerio i Gallus gallus) varia bastant, la Sec es troba en la regió alineada comú en totes elles, per tant, podem saber que es tracta d'una selenoproteïna. A més, també vam predir un element SECIs en la posició corresponent. La seva seqüència es pot trobar en l'apartat de resultats.
Tornar a daltSelS
La Selenoproteïna S està formada per 189 aminoàcids en humans i és codificada pel gen SEPS1. És una proteïna de membrana del reticle endoplasmàtic, que participa en la retrotranslocació de les proteïnes mal plegades cap al citosol, on seran degradades. En situacions d'estrés, l'expressió del gen SEPS1 es veu up-regulada per tal d'evitar l'acumulació de proteïnes mal plegades, sovint relacionat amb procesos inflamatoris. A més a més, l'up-regulació d'aquest gen també s'ha relacionat amb la privació de glucosa. Aquesta selenoproteïna es troba associada a la inflamació, la susceptibilitat al càncer colorectal, a malalties coronàries, artritis, diabetis, etc[25].
El programa Selenoprofiles va predir aquesta selenoproteïna en el genoma de Chrysemys picta bellii a partir d'una query de Taeniopygia guttata. L'alineament comença a partir de l'aminoàcid 26 fins al final i obté l'alineament de la Sec corresponent. A més, prediu els 5 exons i un element SECIs a 850 parells de bases de distància de la regió codificant en posició 3' del gen. El nostre programa inicialment no predia aquesta selenoproteïna però, després de rebaixar l'e-value del blast fins a 1e-1, els programes de predicció van extreure un bon alineament amb la mateixa regió que predia el Selenoprofiles i, a més; també es va obtenir el mateix element SECIs.
Tornar a daltSelT
La selenoproteïna T és una proteïna altament conservada des de plantes fins a humans. Es localitza en el reticle endoplasmàtic a través d'un domini hidrofòbic, tot i que la seva funció és desconeguda en l'actualitat[26].
Es va trobar aquesta selenoproteïna codificada en el genoma de la tortuga. Les evidències que van portar a pensar que es trobava codificada en el genoma d'aquest organisme eren: la conservació dels mateixos exons, la longitut de la proteïna aproximada i l'obtenció dels mateixos resultats per diferents querys analitzades. Ara bé, en tots els casos no es prediu cap SECIs correcte. El nostre programa automatitzat en prediu un però es considera que és erroni perquè està a més de 5000 nucleòtids downstream del gen que codifica per la SelT. Es creu que l'absència de SECIs, pot ser degut a errors en el programa de predicció de SECIs o bé que aquesta selenoproteïna s'està perdent en la tortuga. Es creu però que l'opció més verídica és la primera.
Tornar a daltSelU
La presència de Sec o Cys en la família de les SelU té una distribució poc uniforme en les diferents espècies. Les seqüències obtingudes de mamífers, plantes terrestres, artròpodes, cucs, amfibis i tunicats tenen homòlegs de selenoproteïna amb cisteïna, mentre que les de peixos, aus, equinoderms, algues verdes i diatomees contenen selenocisteïna. En principi, els llevats i les mosques no tenen selenoproteïnes de cap tipus, i en destaca només un protist, T.Pseudonana, que sí que en té [27].
Es va estudiar la possible presència de tres proteïnes de la família Sel U (Sel U1, U2 i U3). Es van analitzar les tres proteïnes que són homòlogues en cisteïna originalment de dues maneres: pel programa automatitzat i segons els resultats de Selenoprofiles. Ambdós programes de predicció donen resultats similars i s'ha pogut localitzar cadascuna de les proteïnes en una regió del genoma de C.picta bellii. En l'apartat dels resultats es mostren les localitzacions exactes. De manera anàloga al resultat exposat per la SelT, tots els alineaments proporcionen puntuacions molt altes, però en aquest cas es van trobar elements SECIs en tots els tres tipus de proteïnes. Com que són homòlegs en cisteïna no esperaríem trobar SECIs però en el cas de SelU2 vam trobar un element SECIs a 8000 nucleòtids de la regió codificant en 3' del gen. No es va considerar un bona predicció d'element SECI, ja que es localitzava molt lluny del gen.
Tornar a daltSelV i W
SelV és una selenoproteïna humana que prové de la duplicació de SelW en mamífers. Les funcions d'aquestes dues no es coneixen però del tot, però se sap que SelV s'expressa exclusivament en testicles mentre que SelW s'expressa en varis òrgans. Totes dues presenten la mateixa estructura gènica. Tot i així, no s'ha pogut anotar la selenoproteïna SelV en el genoma de la tortuga tal i com s'esperava pels resultats obtinguts en altres articles.
Recentment s'han trobat homòlegs de la SelW. En el nostre treball s'han utilitzat les seqüències de les SelW1 i W2 humanes de la base de dades SelenoDB. De forma inesperada s'ha obtingut SelW2, descrita com a proteïna humana. Aquesta presenta una cisteïna en la posició de la Sec de la selenoproteïna W2 dels peixos osteïctis. Basant-nos en un article publicat aquest any per Mariotti. M et al. es creu que s'hauria d'anomenar Rdx12, que correspon a l'homòleg de SelW2 present en mamífers que alinea una cisteïna en la regió on es troba el residu Sec de SelW2. En el nostre cas s'ha localitzat el gen de la Rdx12 codificat en el genoma de la tortuga però no la SelW2, fet que coincideix amb la teoria exposada en l'article publicat per Mariotti. M et al [4]. La query utilitzada per cercar la SelW2 s'ha obtingut de Danio rerio i no s'observa cap resultat significatiu.
En el cas de la SelW1 sí que es va poder anotar aquesta selenoproteïna en el genoma de la tortuga segons els resultats del programa Selenoprofiles, que mostraven una homologia molt alta utilitzant com a query la SelW1 d'ovella. A mé es predia un element SECIs a 1300 nucleòtids de la posició 3' del gen (segons el Blast realitzat en les querys d'humà i pollastre, no mostraven hits estadísticament significatius).
Tornar a daltFamília TR
Les Thioredoxin Reductases (TR o TrxRs) són una família de flavoproteïnes que tenen el mateix mecanisme i seqüència idèntica, inclosa una domini redox catalític conservat de seqüència -Cys-Val-Asn-Val-Gly-Cys-, a les Glutathione reductases. Les TR catalitzen la reducció depenent de NADPH de les proteïnes Tioredoxines, entre d'altres compostos endògens o exògens. L'àmplia especificitat de substrat de les TR de mamífers és deguda a un segon domini redox catalític situat en C-terminal i de sequència -Cys-SeCys- (on SeCys és una selenocisteïna). En mamífers existeixen tres variants amb diferent funció i localització d'aquesta selenoproteïna: TR1, TR2(TGR) i TR3. TR1 es troba principalment al citosol, TR2 a nivell mitocondrial (on neutralitza l'estrès oxidatiu de la cèl.lula causat pels mitocondris) i TR3 es troba principalment en el cervell[18].
En els nostres anàlisis es mostren 3 scaffolds altament significatius amb regions que codifiquen per proteïnes d'aquesta família. Totes elles són altament homòlogues degut al domini redox catalític comú. D'aquesta manera, es creu que és correcte trobar tres scaffolds significatius en el resultat del blast de cada selenoproteïna. Però, coincidint tres regions i tres proteïnes es va concloure que segurament no hi havia cap duplicació i que cada regió codificava per una TR diferent. Els resultats obtinguts pel programa Selenoprofiles han estat d'ajuda per anotar les selenoproteïnes en el genoma de Chrysemys picta bellii. Totes elles es mantenen com a Selenoproteïnes amb la SelCys (U) en la penúltima posició.
Tot i així, es va obtenir que la TGR de Gallus gallus és perfectament homòloga a la TR3 humana i prediu una proteïna que presenta una puntuació molt elevada (99) de T-coffee. Es creu que pot ser degut a l'elevada homologia d'aquesta família i al domini comú que presenten. A més, el Selenoprofiles utilitza la TR3 de Gallus gallus com a query per predir la TR3 de Chrysemys picta bellii i no s'assembla a la TR3 de la nostra llibreria de selenoproteïnes. Cal afegir també que, els resultats obtinguts utilitzant la TR3 de Gallus gallus com a query són molt similars als obtinguts amb la TR2 d'Homo sapiens com a query.
Per últim, el Selenoprofiles detecta elements SECIs en les selenoproteïnes TR1 i 3, ambdós a uns 250 nucleòtids de la regió 3' del gen. En canvi, els programes de predicció utilitzats prediuen un SECIs per la TR2 a 2000 nucleòtids de la regió codificant en 3' del gen.
Tornar a daltAltres selenoproteïnes analitzades
Es va voler analitzar algunes selenoproteïnes presents exclusivament en peixos osteïctis originades a partir de la duplicació del seu genoma. A mesura que s'anava analitzant cada selenoproteïna, ens adonàvem que la gran majoria donaven com a resultat un hit que es localitzava en la mateixa regió que la proteïna humana o de pollastre respectiva. S'han afegit els resultats del nostre anàlisi en una taula que es pot trobar en l'apartat de resultats 'Altres'. Alguns exemples són: no s'ha trobat cap hit del Blast per la SelL, SelJ, SelWb; els hits trobats en SelW2a coincideixen amb la localització de la SelW2 provinent de la query humana i alinea la posició de la Sec amb una Cys. Aquest és el mateix resultat que es va obtenir en l'anàlisi de la SelW2 amb el nostre programa.
Gràcies a Selenoprofiles, s'ha obtingut una nova proteïna anotada, la SelG, que és un homòleg en glicina i en Drosophila melanogaster, és una selenoproteïna rica en glicina.
Maquinària de Selenoproteïnes
Es va analitzar la presència de maquinària de síntesi de selenoproteïnes en el genoma de Chrysemys picta bellii. Utilitzant com a querys la maquinària de síntesi humana, es va aconseguir localitzar la regió genòmica de SPS1 (selenophosphate synthetase 1), SPS2 (selenophosphate synthetase 2), Secp43 (tRNA selenocysteine associated protein), eFSec (selenocysteine-specific elongation factor), SBP2 (Selenocysteine Binding Protein 2) i SecS (SPCS). Inicialment, es creia que la proteïna pstk (O-phosphoseryl-tRNASec kinase) no es trobava en el genoma de la tortuga ja que els alineaments que mostraven presentaven molts gaps i l'homologia observada en el T-coffee era molt baixa. Tot i així, el resultat del Blastp contra la base de dades de proteïnes no redundant d'NCBI, mostra homologia amb pstks d'altres espècies. Podem concloure doncs, que no hi ha suficient informació per confirmar que aquesta regió genòmica codifica per pstk.
Altres resultats també són dubtosos. La SBP2 presenta certa controvèrsia ja que el programa Genewise no prediu un bon alineament, i el programa SECISearch prediu un SECIs dins la regió codificant d'aquesta proteïna de la maquinària de síntesi. A més, l'alineament d'Exonerate mostra un codó STOP en l'aminoàcid 13 i el T-coffee no presenta una elevada homologia. Tot i així, mostra una gran homologia en el domini catalític, fet que porta a pensar que aquesta proteïna es troba codificada en el genoma però amb modificacions respecte la humana. D'altra banda, la part final de la proteïna SecS no surt alineada perquè sortia de l'scaffold degut a que és una proteïna molt gran. Tot i així, amb les dades obtingudes podem concloure que està present en el genoma de la tortuga analitzada.
Tornar a dalt