INTRODUCCIÓ
Selenoproteïnes
1. Què són les selenoproteïnes?
Les selenoproteïnes són proteïnes que incorporen un aminoàcid anomenat selenocisteïna (aminoàcid vint-i-u). Aquest aminoàcid es troba a la major part d’espècies (archaea, procariotes i eucariotes). No obstant, el número de selenoproteïnes varia molt per als diferents taxons. Homo sapiens té vint-i-cinc selenoproteïnes, Drosophila melanogaster en té tres, mentre que el cuc Caenorhabditis elegans només en té una.
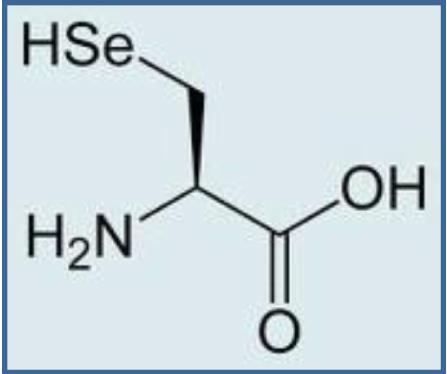
La selenocisteïna (Sec) és un anàleg de la cisteïna (Cys) que en comptes de tenir un àtom de sofre (S), conté un àtom de seleni (Se). Aquests dos elements tenen una reactivitat semblant i presenten unes propietats químiques similars.
A vegades, l’ortòleg d’una selenoproteïna conté una cisteïna enlloc d’una selenocisteïna. Molt sovint trobem que la mateixa proteïna en una espècie determinada és una selenoproteïna, en canvi en una altra espècie, en la mateixa posició, en comptes d’una selenocisteïna hi ha una cisteïna, per tant tenim un homòleg en cisteïna. Fins i tot, dins de la mateixa espècie trobem un gen amb selenocisteïna i una còpia del mateix amb una cisteïna (paràleg).
Normalment les selenoproteïnes tenen només un aminoàcid selenocisteïna, però hi ha excepcions i algunes en tenen més d’un.
2. Quin és el paper del seleni?
El seleni és un element traça, que no és molt abundant a la terra però que és essencial per als animals, per molts microorganismes i per altres éssers vius. La deficiència de seleni pot donar lloc a determinades malalties.
Tot i que no podem viure sense seleni, un element rar, estrany i poc abundant, un excés de seleni és tòxic.
En el cas dels animals, dels eucariotes i dels protists, el seleni es troba en les cèl·lules formant part de selenoproteïnes, no trobem seleni lliure.
3. Biosíntesi
L’aminoàcid selenocisteïna és codificat pel codó UGA, el qual és, pràcticament sempre, un codó de terminació de la traducció.
La recodificació del codó UGA com a codó per la selenocisteïna és mediada per una estructura tridimensional en l’extrem 3’ no traduït (UTR) dels gens de les selenoproteïnes. Aquesta estructura rep el nom de SElenoCystein Insertion Sequence (SECIS Element).
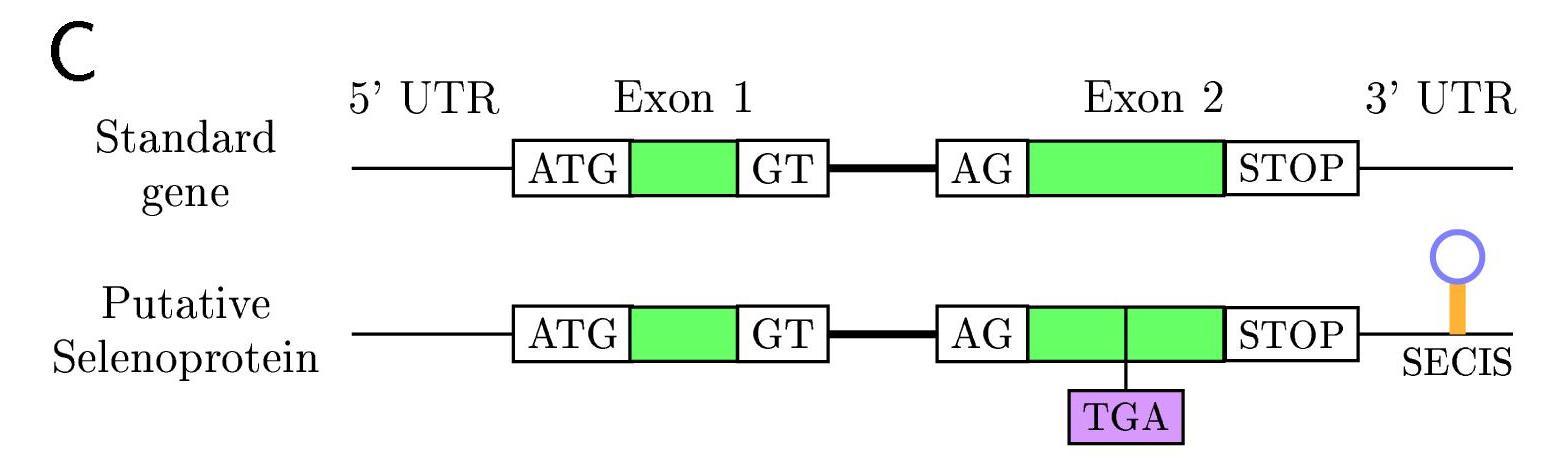
Aquest element recluta una proteïna anomenada SECIS Binding Protein (SBP2), la qual s’uneix a un factor d’elongació específic de selenoproteïens (Efsec). Efsec apropa el tRNA de la selenocisteïna (tRNASec) al codó UGA del mRNA de les selenoproteïnes i en determina la seva traducció a selenocisteïna; la traducció del mRNA continua fins que el ribosoma reconeix el següent codó de terminació. D’aquesta manera, el ribosoma, que normalment quan es trobés l’UGA acabaria la traducció, el que fa és reconèixer l’UGA per un codó amb l’aminoàcid selenocisteïna i continua la lectura fins el següent codó de terminació, que pot ser qualsevol dels tres codons (UAG, UAA o UGA).
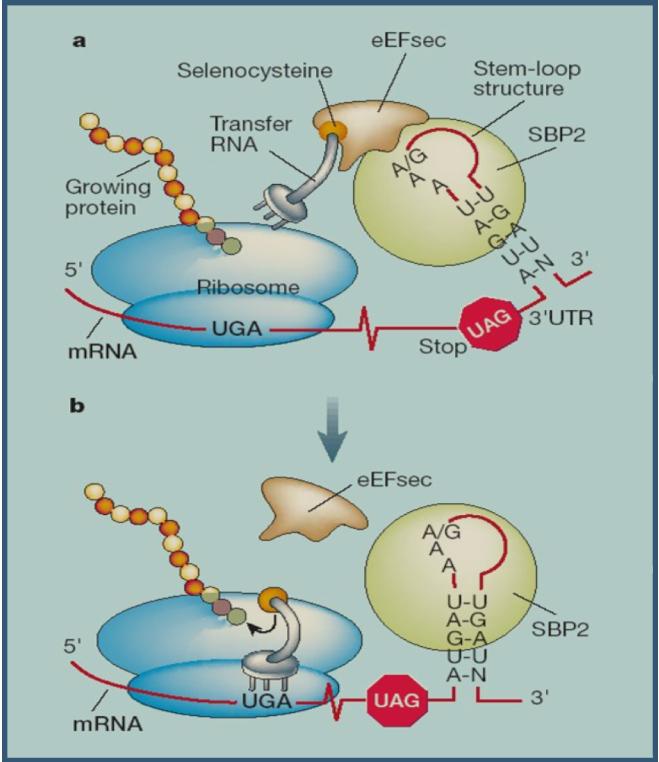
Altres proteïnes participen específicament en la síntesi de selenocisteïna. Entre aquestes es troben la Selenofosfat Sintetasa (SPS1 i SPS2), la Selenocisteïna Sintasa (SLA/LP), la Phosphoseryl tRNA Kinase (PSTK) i la Selenocysteine associated protein (SECP43).
4. Funció
D’una gran part de les selenoproteïnes no se’n coneix la funció, però hi ha un gran grup que actuen com a enzims d’oxidoreducció, per la qual cosa hi ha indicis d’un possible paper antioxidant, com és la neutralització de radicals lliures i l’equilibri redox intracel·lular. Un exemple serien la Tioredoxina reductasa i la Glutatió peroxidasa.
Per altra banda, també s’han trobat selenoproteïnes que participen en la mateixa síntesis de les selenocisteïnes o en la activació i inactivació de l’hormona tiroide, com el cas de les Deiodinases.
5. Per què és difícil identificar selenoproteïnes?
La utilització excepcional del codó UGA per codificar selenocisteïnes (Sec), fa molt difícil la identificació de les selenoproteïnes perquè pràcticament tots els programes bioinformàtics assumeixen que aquest codó codifica per una senyal de terminació de la traducció. És per aquest motiu que la identificació de selenoproteïnes requereix la utilització d’altres mètodes. A més, sense supervisió humana normalment resulta molt difícil caracteritzar el selenoproteoma d’un determinat genoma. Per aquesta raó, és tan desconeguda l’existència de les selenoproteïnes en la majoria dels genomes seqüenciats fins ara.
Família TR
Les Thioredoxin reductases (TrxRs) són una família de pyridine nucleotide-disulphide oxido-reductases que contenen seleni. Presenten una seqüència i un mecanisme d’acció que també trobem a les Glutathione reductases, incloent un domini catalític -Cys-Val-Asn-Val-Gly-Cys- altament conservat.
Les TrxRs catalitzen la reducció depenent de NADPH de les proteïnes Thioredoxins, com també d’altres components endògens o exògens. La gran especificitat de substrat de les TrxRs de mamífers és degut a un segon domini redox a l’extrem C-terminal: -Cys-SeCys- (on SeCys és una selenocisteïna) que no es troba en la glutathione reductase ni en la TrxR d’E.coli.
Els electrons són transferits de NADPH via FAD al domini actiu disulphide de TrxR, i llavors, es redueix el substrat.
Les principals funcions són subministrar equivalents reductors a enzims com la ribonucleotid-reductasa i la thioredoxin peroxidase. També s’ha vist que tenen funcions com a factors de creixement cel·lular i d’inhibició de l’apoptosis ja que TrxR activa directament el supressor tumoral p53. TrxR i Trx estan sobreexpressats en molts tumors agressius i participen en carcinogènesis, progressió cancerígena i resistència a medicaments. Molts fàrmacs usats en clínica (cisplatin o arsenictrioxide) i agents quimicopreventius s’ha demostrat que inhibeixen TrxR.
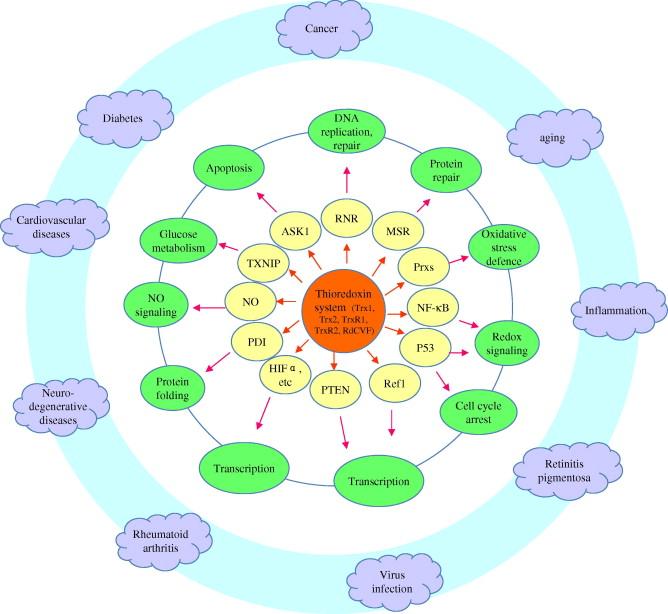
TrxR és un selenoenzim amb tres isoformes, TrxR1 al citosol i extracel·lular, TrxR2 a mitocòndries i TrxR3 o TGR (thioredoxin glutathione reductase) present primàriament en cervell. Comparat amb el sistema Trx procariota, l’evolució més remarcable és que TrxR és un selenoenzim gran, diferent dels petits enzims específics presents en tots els bacteris, fongs i plantes.
Família Fep15
La majoria de selenoproteïnes eucariotes conegudes es troben representades al selenoproteoma de mamífers. A més, moltes d’elles presenten ortòlegs en cisteïna. Fep15 és un homòleg relativament distant de la família de selenoproteïnes Sep15 que només es troba en peixos.
Es creu que Fep15 va ser originada per duplicació gènica de SelM en animals, molt probablement en peixos. A més, és present en aquests organismes només en forma de selenoproteïna (no es troben homòlegs en cisteïna). La disponibilitat de seleni al mar, pot ser un factor que hagi influït a l’evolució d’aquesta família de selenoproteïnes. Aquesta selenoproteïna es troba localitzada al RE i possiblement al Golgi ja que presenta un domini RDEL localitzat al C-terminal.