SELENOPROTEINAS en Dictyostelium purpureum

|
 |
| { | Presentación | | | Organismo | | | Genoma | | | Selenoproteínas | | | Métodos | | | Información | | | Nosotros | } |
|
| Selenoproteínas | | | SECIS | | | tRNASec | | | Selenoproteoma: | |
Enlaces


|
¿Qué son las selenoproteínas?
Tal vez sea el código genético uno de los elementos de la vida que menos variaciones ha admitido a lo largo del tiempo. A menudo, pensar en la vida es pensar en la diversidad. Por eso, cuando fijamos nuestra atención en el conjunto de los 64 tripletes del código y vemos la universalidad de su significado no podemos hacer otra cosa que intentar entender esta excepción. Pero en biología siempre hay excepciones, incluso en las excepciones: algunos organismos presentan variaciones del código universal. Quizá la más sorprendente de estas variaciones sea la del doble significado del codón UGA, que, pese a que la inmensa mayoría de las veces determina la parada de la traducción, en algunos casos codifica un aminoácido que también es excepcional: la selenocisteína.
La mayoría de las veces, la presencia de un codón UGA en un RNA mensajero (mRNA) significa la finalización de la cadena peptídica que se está ensamblando en los ribosomas. Una proteína llamada RF1 (Release Factor 1), que mimetiza la estructura de los RNAs de transferencia (tRNAs), es capaz de interaccionar con el ribosoma cuando en éste se encuentra un codón de terminación (como UGA), y este hecho conlleva la terminación de la traducción. Sin embargo, en ocasiones, el codón UGA, en lugar de reclutar a RF1, interacciona con un tRNA unido a selenocisteína, de modo que la traducción no sólo no termina, sino que se sintetiza una proteína con un aminoácido poco habitual en su cadena polipeptídica. El hecho de que el codón UGA actúe como codón de terminación o como codón para selenocisteína depende de una región de la molécula de mRNA que se encuentra en el lado 3' respecto del marco abierto de lectura (en algunas bacterias, se sitúa dentro de la secuencia codificante o incluso en el lado 5' respecto del marco abierto de lectura). Esta región se conoce con el nombre de elemento SECIS (Selenocysteine Insertion Sequence). Las características que ha de tener cierta región de un mRNA para ser elemento SECIS no son fáciles de definir ni de identificar. Tanto la distancia a la que los elementos SECIS se encuentran de los respectivos codones UGA como sus propias secuencias nucleotídicas varían considerablemente. Esta diversidad sugiere que la probabilidad de una región de mRNA de operar como elemento SECIS depende de la estructura secundaria que adopte. Como es lógico, este hecho hace difícil la búsqueda de selenoproteínas, ya que analizar estructuras secundarias es mucho más complejo que buscar secuencias de caracteres en una molécula de mRNA. No obstante, la presencia de elementos SECIS en el genoma de un organismo no necesariamente implica que codifique selenoproteínas. La expresión de selenoproteínas depende de la presencia de una serie de proteínas específicas sin las cuales la incorporación de selenocisteína no sería posible. Por tanto, la búsqueda de selenoproteínas en un genoma no sólo debe incluir la identificación de genes candidatos y de elementos SECIS, sino también el encuentro de los genes que codifican estas proteínas efectoras.
La selenocisteína (Sec o U) es una cisteína en la que el átomo de azufre ha sido sustituido por un átomo de selenio. Debido a la estructura electrónica del selenio, la selenocisteína es más ácida que la cisteína, es decir, el anión de selenocisteína es más estable que el de cisteína. Este hecho implica que la energía necesaria para ionizarse es menor y tiene, por tanto, una mayor capacidad de atraer sustancias electrófilas. Esto explica por qué prácticamente todas las selenoproteínas descritas en eucariotas participan en reacciones de oxidación-reducción y presentan, en sus centros activos, los residuos de selenocisteína. En ocasiones, las selenoproteínas son homólogas a proteínas de otras especies que en lugar de selenocisteína presentan cisteína (a pesar de que el potencial reductor de la cisteína es menor que el de la selenocisteína, estos aminoácidos exhiben propiedades químicas muy similares). En bacterias, el papel de las selenoproteínas parece ser algo distinto (muchas selenoproteínas de bacterias participan en procesos catabólicos, por ejemplo). Por esta y otras razones, algunos autores rechazan la hipótesis de un origen único para todas las selenoproteínas.
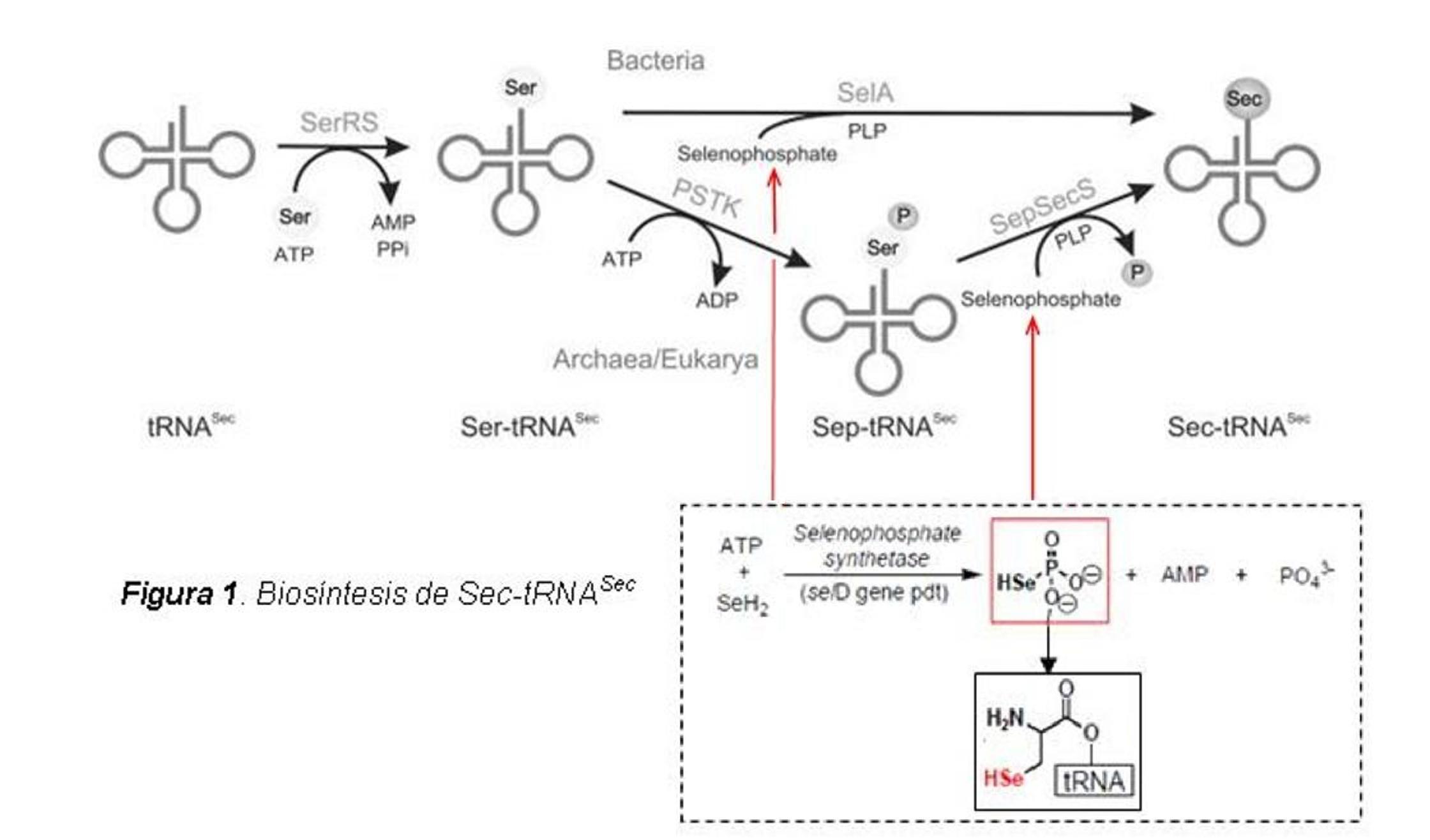
Síntesis de selenoproteínasSíntesis de selenocisteína En la síntesis de una selenoproteína, la selenocisteína (Sec) se incorpora a la cadena polipeptídica en formación mediante un RNA de transferencia específico: tRNASec. Tanto en procariotas como en eucariotas, el tRNASec presenta algunas particularidades estructurales respecto los RNAs de transferencia convencionales y sufre modificaciones post-transcripcionales específicas que son determinantes para su función. A diferencia de lo que ocurre con la mayoría de aminoácidos, la selenocisteína se sintetiza directamente sobre su RNA de transferencia. En primer lugar, una seril-tRNA sintetasa convencional (SerRS) cataliza la unión de un aminoácido serina con el tRNASec para dar lugar a seril-tRNASec (Ser-tRNASec). Más tarde, la serina del Ser-tRNASec es modificada a selenocisteína, de forma que se obtiene selenocisteil-tRNASec (Sec-tRNASec), que ya puede intervenir en la traducción de mRNAs. En eubacterias, esta reacción depende de una selenocisteína sintasa (SecS o SelA). En eucariotas y en arqueobacterias, el proceso incluye reacciones intermedias. Primero, el Ser-tRNASec es fosforilado a fosfoseril-tRNASec por la enzima PSTK (phosphoseryl tRNASec kinase), y, posteriormente, un conjunto de proteínas entre las cuales se encuentra una selenocisteína sintasa (SepSecS) permite la incorporación de selenofosfato.
Para la incorporación de los distintos aminoácidos a las proteínas en crecimiento son necesarios ciertos factores de elongación que se unen a los tRNA y permiten su interacción con los ribosomas. En el caso del Sec-tRNASec, esta función la lleva a cabo un factor de elongación específico: EFSec (selenocysteine elongation factor). EFSec de procariotas (también llamado SelB) es una proteína dependiente de GTP con un dominio de factor de elongación específico para Sec-tRNASec en su extremo N-terminal y un dominio de unión a SECIS en su región C-terminal. EFSec de eucariotas es similar al anterior pero no se une al elemento SECIS directamente, sino a través de la proteína SBP2 (SECIS binding protein 2). SBP2 tiene dominios de unión al elemento SECIS, al complejo EFSec-Sec-tRNASec y a la subunidad mayor de los ribosomas. Se ha visto que la afinidad de SBP2 por los elementos SECIS es variable y depende de la estructura de estos elementos. Es posible que éste sea uno de los factores por los cuales la tasa de traducción de las distintas selenoproteínas no es la misma. Otra proteína que interviene en el proceso de traducción del codón UGA como selenocisteína en eucariotas es L30. L30 está asociada a los ribosomas y se une a la misma región del elemento SECIS que SBP2 (compite, por tanto, con él). Aunque se desconoce el papel preciso de estas proteínas en el proceso, las complejas interacciones que se establecen entre ellas, el elemento SECIS, el Sec-tRNASec y otras proteínas acaban permitiendo que el Sec-tRNASec se una al ribosoma para que la selenocisteína sea incorporada a la proteína en formación.
|
|
