Glutathione Peroxidases (GPx)
Són selenoproteïnes que tenen una funció de protecció cel·lular gràcies a catalitzar la reducció del peròxid d'hidrogen i peròxids orgànics mitjançant el glutatió. En l'espècie humana se'n distingeixen fins a 8 GPx diferents (algunes de les quals no contenen selenocisteïna sinó que són homòlegs en cisteïna). Altres espècies però, no les tenen totes i no sempre són selenoproteïnes.
L'anàlisi de les GPx en P. berghei comença per obtenir les seqüències conegudes de les diferents proteïnes que componen aquesta superfamília en el Selenodb i realitzar un tBLASTn contra el genoma del nostre organisme. S'obtenen bons hits (E-values significatius) per algunes de les proteïnes però no de totes. Tot i això destaca que en tots els casos en què l'E-value és significatiu, el contig alineat és el mateix (gi|56491127|emb|CAAI01005912.1) cosa que sembla indicar que P. berghei pot tenir només una GPx.
Per corroborar aquesta teoria fem el següent: Analitzar individualment alguns dels hits obtinguts.
Per començar s'analitza GPx2 d'Homo sapiens. Primer s'agafa el contig amb el que s'ha obtingut un bon hit amb el BLAST. Es realitza un exonerate contra el genoma de P. berghei sense cap resultat. Per això fem un genewise (Frame negatiu), en el que es veu com s'alinea la Sec (U) humana amb una Cisteïna (codó TGT).
Com que la proteïna que s'obté no està completa es decideix fer un pBLAST de la seqüència obtinguda contra tota la base de dades del NCBI, per veure si realment la proteïna predita pot ser una GPx o, pel contrari, és una altra de diferent.
Els hits resultants del pBLAST són GPx de diverses espècies, entre les que destaquen espècies de Plasmodium com P. chabaudi, P. knowlesi i inclús del mateix P. berghei, però aquesta última no està ben anotada, ja que no comença per una metionina. Per tal d'analitzar aquest últim fet i anotar correctament la proteïna predita amb el genewise s'utilitzarà com a seqüència de comparació la seqüència aminoacídica GPx de P. knowlesi (que està ben anotada) en comptes de la de Homo sapiens, ja que és molt més semblant al nostre organisme que aquesta última.
Es repeteix el procés anterior utilitzant la GPx de P. knowlesi en comptes de la d'Homo sapiens (tBLASTn) anotant el gen de P. berghei amb l'exonerate i el genewise. En aquest cas la proteïna comença per una metionina i conté dos introns en comptes d'un com s'obtenia abans.
Finalment per tal de comparar la proteïna obtinguda amb la de P. knowlesi i d'Homo sapiens es realitza un T-Coffee entre les tres espècies, veient com la Sec s'alinea amb cisteïna dels dos plasmòdiums. També es va realitzar un alineament entre els dos plasmòdiums per veure com efectivament són molt més semblants entre elles que amb la seqüència humana.
Per acabar d'assegurar els resultats obtinguts es fa el mateix amb GPx4 d'Homo Sapiens. Amb el contig obtingut amb el tBLASTn s'utilitza la seqüència aminoacídica de GPx4 d'Homo sapiens per fer un exonerate ,sense resultat, i genewise, que torna a donar una proteïna incompleta, pel que es repetirà l'estratègia anterior, fer un pBLAST amb la seqüència obtinguda contra la base de dades del NCBI. Al igual que en el cas anterior, els resultats del pBLAST donen com a hit diverses GPx de plasmòdiums, inclòs el propi P. berghei. Es torna a realitzar un exonerate i un genewise utilitzant com a query la GPx de P. knowlesi i s'alinea mitjançant T-Coffee la proteïna obtinguda amb l'obtinguda abans quan estàvem mirant GPx2. Observant que la proteïna obtinguda és idèntica en els dos casos.
Amb aquests resultats creiem que P. berghei només conté una GPx en el seu genoma, ja que en tots els alineaments entre les diferents GPx de les diverses espècies es realitzaven sobre el mateix contig, i analitzant-ne dues més exhaustivament, GPx2 i GPx4 d'Homo sapiens, es va obtenir com a resultat la mateixa seqüència proteica.
Per últim queda analitzar l'anotació de la GPx del NCBI, que com s'ha dit abans no sembla que estigui ben anotada ja que no comença per una metionina. Per mirar-ho es va realitzar un T-Coffee entre la seqüència de GPx de P. berghei del NCBI, la GPx de P. knowlesi del NCBI i la GPx obtinguda mitjançant el genewise. Els resultats indiquen que el problema està en que davant de la metionina està anotada una treonina, però la resta de la seqüència és exactament igual a la obtinguda pel genewise.
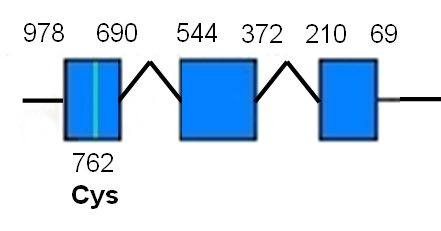
En aquest esquema es pot apreciar l'estructura del gen de la GPx.
Descarregar l'anotació del gen en format GFF.
Descarregar el cDNA del gen.
Descarregar la seqüència de la proteïna.
Aquí podeu descarregar tota la documentació referent a GPx.
