Basant-nos en l'article de Lobanov et al.vam poder identificar una nova possible selenoproteïna que no estava present a la base de dades SelenoDB. L'article menciona la presència de la selenoproteïna selTryp a Leishmania major. Vam procedir a cercar la seqüència peptídica de la proteïna en un organisme proper al nostre en la base de dades GeneDB. L'organisme triat va ser Trypanosoma brucei brucei, també emprat en l'article citat anteriorment per fer els diferents aliniaments amb les selenoproteïnes que s'hi esmenten.
La seqüència de la proteïna en T. brucei és:

on, segons la figura següent, extreta de l'article citat anteriorment, l'asterisc * representaria la selenocisteïna.

Una vegada vam tenir guardada en un arxiu la seqüència aminoacídica de la proteïna selTryp de Trypanosoma brucei brucei, vam fer un TBLAST contra la base de dades de Leishmania a GeneDB. Es pot observar que l'e-value és extraordinàriament baix (2.5e-122) i per tant els nostre aliniament molt signitificatiu, i que el nostre possible gen es troba al cromosoma 34:

A continuació vam fer un TBLASTN contra tot el genoma de Leishmania, de manera que poguessim veure en quina regió genòmica es troba el nostre gen.
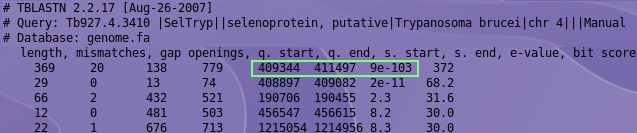
Com podem veure, també vam obtenir un hit amb un e-value molt baix (9e-103). El que es va fer a continuació va ser tallar el tros d'interès de la seqüència mitjançant el programa fastasubseq, de manera que vam agafar una regió cromosòmica que englobés la proteïna i també el possible SECIS. Es van tallar 5000 parells de bases a partir de la posició nucleotídica 408000 ja que, segons el TBLASTN de la terminal, l'inici del gen es trobava al voltant de la posició 409344. Es va redirigir aquesta informació a un arxiu nou que contenia només aquest tros de seqüència.
El següent pas va consistir en analitzar aquesta regió cromosòmica amb el programa de predicció GeneWise. Com que es necessita una proteïna query per comparar amb la nostra seqüència nucleotídica i així predir la nostra proteïna, vàrem agafar la selTryp de Trypanosoma brucei brucei.
Resultats Genewise
Proteïna predita
Com es pot apreciar, el GeneWise interpreta el nostre codó TGA com un codó STOP i per tant, la nostra proteïna predita no és exactament com la que es reflexa en l'article de Lobanov et al., si no que li manquen els tres últims aminoàcids. De fet, el programa tampoc ens prediu els primers aminoàcids de la proteïna. La següent imatge ens mostra la hipotètica proteïna selTryp que apareix a la base de dades GeneDB.
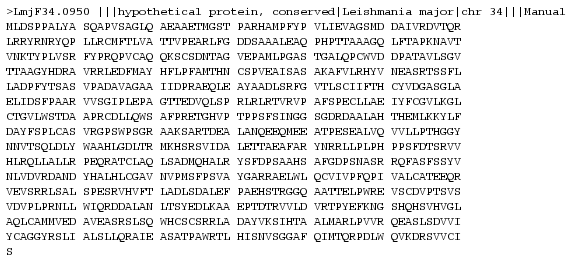
El mateix problema sorgeix alhora d'aliniar amb el programa T-COFFEE: degut a que la nostra proteïna predita no inclou la selenocisteïna, l'aliniament entre selTryp de Trypanosoma brucei brucei i la predicció de Leishmania major no inclou els aminoàcids següents al codó TGA. A més, tampoc s'alineen les Metionines inicials ni tan sols els primers aminoàcids. Ho podem comprovar als següents aliniaments:
Per poder predir l'estructura del gen, també vam utilitzar GeneWise. Com ja hem vist anteriorment, el gen codificant per selTryp conté dos exons i un intró. Concretament, el primer exó aniria des de la posició 408786 fins a la 409977 (hem de tenir en compte que en el nostre aliniament de GeneWise la proteïna predita no estava completa si no que faltàven aminoàcids de l'inici, concretament 37, per tant hem de comptar com a inici del gen 111 nucleòtids més upstream). L'intró correspon a la regió entre els nucleòtids número 409978 i 410970, i el segon exó va des de la posició 410971 a la 412274.
Per altra banda, vam procedir a la búsqueda de l'element SECIS corresponent. En aquest sentit vam tenir problemes per què el programa SECISearch no era capaç de trobar-lo, tot i provant amb tots els patrons de búsqueda. Cal remarcar que en l'article que hem utilitzat de referència es mencionava el fet de que els SECIS de T. brucei i de L. major no es van poder trobar mitjançant el programa SECISearch, si no que aquest es va haver de modificar o bé es van trobar manualment utilitzant altres programes. Tot i això, com que sabíem la seqüència del SECIS per què l'article ens la proporcionava, vam provar de cercar-la manualment, fent servir l'opció crtl+F (find) de l'editor Emacs. En aquest cas sí vam poder trobar la seqüència corresponent, a 4395 bases del codó TGA codificant per selenocisteïna. Per fer això va ser necessari tallar un altre cop la seqüència però aquesta vegada vam tallar 10500 nucleòtids, per poder englobar el SECIS.
L'estructura general del nostre gen, per tant, seria:

Pel que fa a la funció i dominis de la nostra proteïna, el programa utilitzat va ser InterProScan, que prediu dominis funcionals de seqüències proteïques determinades. El resultat el mostrem a continuació:
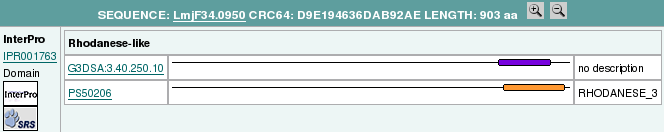
Com podem veure, ens prediu un domini rodanasa. La rodanasa és un enzim mitocondrial implicat en la detoxificació de tiocianat a cianur mitjançant reaccions redox. Això coincidiria amb la idea que aporta l'article de Lobanov et al. de que el motiu CxxU (és a dir, una cisteïna seguida de dos aminoàcids qualsevol i, a continuació, una selenocisteïna) present en algunes selenoproteïnes com selTryp, estaria implicat en processos redox mitjançant la formació d'enllaços no covalents selenilsulfit.
Per concloure, ens vam qüestionar si realment estàvem davant d'una selenoproteïna o no. Com que no teniem cap dada decisiva, vam voler fer una última prova. Abans de tot, però, explicarem el nostre raonament.
La imatge 3 mostra la seqüècia SECIS predita en l'article de Lobanov et al. per les diferents espècies. El que es pot apreciar clarament és que els SECIS de Trypanosoma brucei brucei i de Leishmania major són molt diferents a la resta.
Nosaltres vam voler predir l'estructura de la nostra seqüència SECIS. Com que SECISearch no ens va ser útil la primera vegada, vam provar a utilitzar un altre programa de predicció de plegament de RNA. Per dur a terme això, primer de tot vam haver de buscar manualment la seqüència exacta del SECIS i, a continuació, transcriure-la a RNA mitjançant la següent plana web.
La seqüència específica de DNA és:
CCCTGCCGCCTGCACTGGACGTGGTGAGCCGTGAGGGCGCGGCGCAGGTGGTGTCGACGGTGGTGTGCCGCG
CGTGAGGGCCGCTGTGCCCTCTCCCG
I la corresponent de RNA és:
CCCUGCCGCCUGCACUGGACGUGGUGAGCCGUGAGGGCGCGGCGCAGGUGGUGUCGACGGUGGUGUGCCGCG
CGUGAGGGCCGCUGUGCCCUCUCCCG
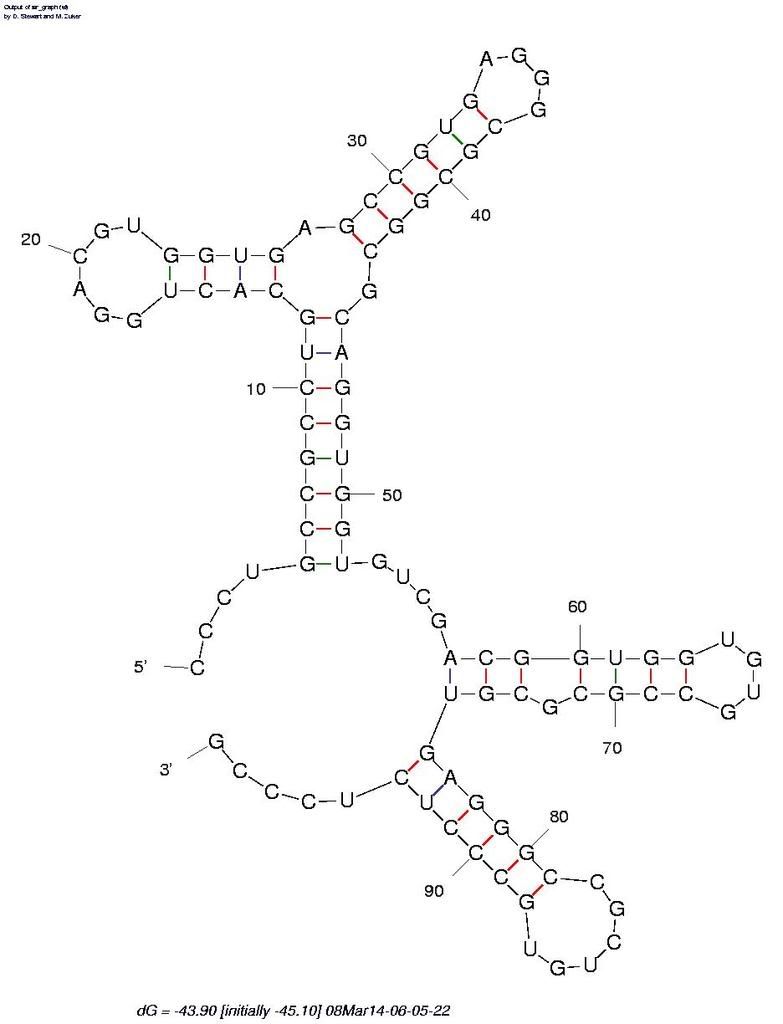
Mitjançant aquesta seqüència de RNA vam fer córrer el programa MFOLD.
La seqüència predita és la que es pot veure a la imatge 4. No podem concloure que sigui un SECIS perquè la seva estructura no correspon al que seria estríctament un SECIS, si no que el plegament observat és força diferent. Per tant, això ens conduiria a pensar que no estem realment davant d'una selenoproteïna.
A més, segons els autors de l'article, de totes les selenoproteïnes que es van trobar, selTryp va ser l'única que no van poder expressar mitjançant mètodes experimentals.
Així que, la diferència abans esmentada en L. major en quant a la diferent seqüència del SECIS i la falta de proves experimentals, ens fan pensar que, tot i la nostra idea inicial, possiblement no estiguem davant d'una selenoproteïna, si no d'un gen amb un codó STOP TGA que no codifica per cap selenoproteïna.




Resultats Genewise
Proteïna predita