Estudi de la proteïna predita
Un cop obtinguts els resultats amb la seqüència extesa s'ha realitzat un BLAST per tal de trobar possibles proteïnes homòlogues a les predites. Com que després d'analitzar els resultats veiem que la predicció més fiable és la del geneid, agafem la proteina que ens prediu i realitzem un blastp.
Com podem observar en els resultats del blastp, s'han obtingut diferents aliniaments significatius amb la proteïna predita. Trobem diversos hits corresponents a phospholipases C, tant d'Homo sapiens com d'altres organismes (Mus musculus, C. elegans...). Veiem també que apareixen diferents dominis conservats que corresponen a phospholipases, de manera que obtenim una evidència de funció. Per tal de trobar el percentatge de similitud entre la seqüència proteica predita pel geneid i la predita pel genscan amb el hit del blastp que presenta major score i un e value de 0.0 ( KIIA1902) realitzem un aliniament per parells basat en l'algorisme de programació dinàmica (Needleman and Wunsch) amb CLUSTAL (KIAA1092 amb la proteïna predita pel geneid, i per altra banda KIAA1092 amb la proteïna predita pel genscan). Això ho fem així ja que si realitzem un aliniament m˙ltiple progressiu, com que les seqüències de les dues prediccions són més semblants entre elles que amb la proteïna obtinguda pel blastp, els resultats es veurien afectats. Els resultats obtinguts amb el clustal es mostren a continuació:
- Aliniament de la proteïna KIAA1092 obtinguda a través del blastp i la predita pel geneid
- Aliniament de la proteïna KIAA1092 obtinguda a través del blastp i la predita pel genscan
Veiem en els resultats que els dos aliniaments tenen puntuacions molt altes; tot i que en el cas del geneid encara és millor (similitud 95% vs 88%). D'altra banda observem també que els exons centrals coincideixen amb les posicions dels exons corresponents a la proteïna KIAA1092 de manera força exacta. En el cas de la proteïna predita pel geneid, i també pel genscan, veiem que determinats exons ( exó 5 pel geneid i 6,7,8 pel genscan) codifiquen per un nombre d'aminoàcids més elevat que els de la KIAA1092; això és degut a que els programes prediuen aminoàcids falsos positius ja que el potencial codificant d'aquests exons és major repecte als reals per una manca d'especificitat en la predicció. Finalment veiem que on es presenten més diferència és en el primer exò, on les dues prediccions comencen 91 aminoàcids després del primer aminoàcid de la KIAA1092, i presenten dues regions amb gaps. Això ens reforça la idea que els exons inicials predits són falsos positius. De tota manera la predicció en la resta d'exons és molt bona, sobretot la obtinguda pel geneid.
Vistos els bons resultats de l'alineament hem buscat informació sobre la proteïna KIAA1092 hom˛loga a les predites pels programes de predicció de gens, en diferents bases de dades per tal de comprovar si la seva localització corresponia amb la mateixa regió del cromosoma que la nostra seqüència i d'aquesta manera obtenir una evidència més de que la proteïna predita correspongui a aquesta, i efectivament així ha estat.
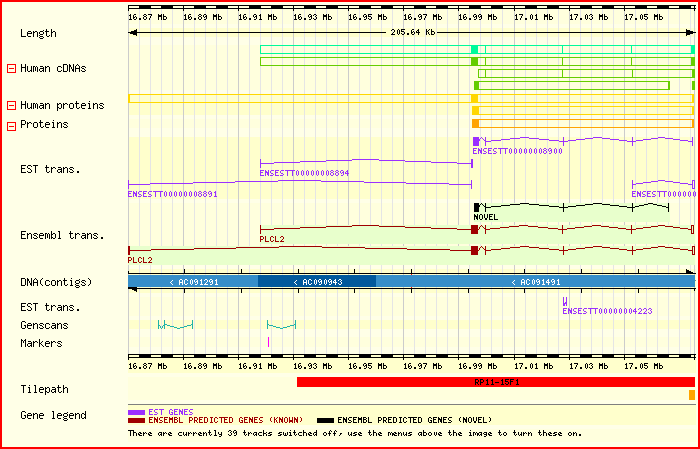
Fig 11. Podem observar que la proteïna KIAA1092 és una phospholipasa C i es troba
situada en gran part en la nostra seqüència (AC091491)
Veiem en aquestes imatges que efectivament l'exó inicial de la proteïna no correspon al predit pels programes de predicció de gens, i que en canvi apareix molt llunyà respecte la resta d'exons. Veiem també amb la informació que proporciona l'Ensembl que es prediuen dos trànscrits diferents que corresponen a cDNAs específics i/o seqüències de proteïnes, en els quals la diferència principal són els exons inicials, que es troben en diferents posicions per cada un dels trànscrits. Amb aquests resultats obtinguts a partir de l'Ensembl hem decidit representar gràficament els exons corresponents als dos trànscrits de la proteïna juntament amb les prediccions dels gens obtingudes i a partir d'aquí extreure'n conclusions.
Per tal de realitzar això hem transformat les diferents posicions dels exons de la proteïna obtingudes a partir de l'ensembl a posicions corresponents a la nostra seqüència des de la primera base.
-
Informació dels exons del primer transcrit de la PLCL2
-
Informació dels exons del segon transcrit de la PLCL2
| n║ exó | Exons trànscrit 1 | Posicions a la seqüència | Exons trànscrit 2 | Posicions a la seqüència |
| 1 | 16866333-16866746 | 28658-29071 | 16914463-16914664 | 76788-76989 |
| 2 | 16991047-16993533 | 153369-155858 | 16991047-16993533 | 153369-15585 |
| 3 | 16996081-16996284 | 158406-158609 | 16996081-16996284 | 158406-158609 |
| 4 | 17024247-17024322 | 186572-186647 | 17024247-1702432 | 186572-186647 |
| 5 | 17049329-17049438 | 211654-211763 | 17049329-17049438 | 211654-211763 |
| 6 | 17071106-17071967 | 233431-234292 | 17071106-17071968 | 233431-234293 |
Taula 1. Posicions en la seqüència dels diferents exons de la proteïna
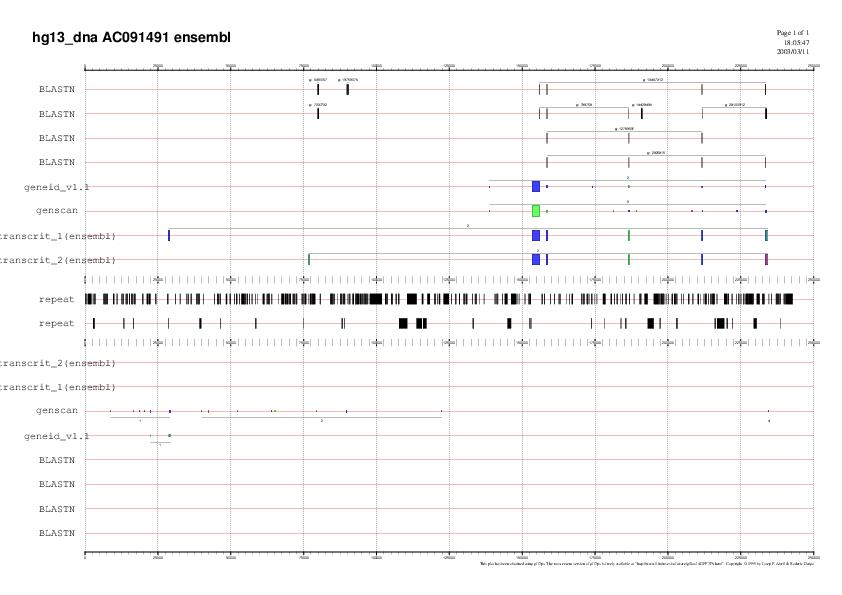
Un cop obtingudes les posicions construim dos fitxers gff (trànscrit_1.gff i trànscrit _2.gff) partir dels quals podrem representar a través del gff2ps un gràfic conjuntament amb els gens predits en la seqüència extesa i els ESTs trobats.
| Fig 12. Com podem veure els exons inicials de les dues variants de la proteïna kiaa1092 es troben en regions força distants a la resta d'exons, i tot i que l'estratègia d'extendre la seqüència era bona, perquè com veiem cauen dins de la regió ampliada, els programes de predicció de gens no n'han predit cap dels dos. |