![]() 1. ANÀLISI DE LA SEQÜÈNCIA DE PROTEÏNES
1. ANÀLISI DE LA SEQÜÈNCIA DE PROTEÏNES
Per definir els membres pertanyents a la familia ADH es va entrar a la base de dades Swissprot. De les 1345 entrades obtingudes entrant la paraula “alcohol dehydrogenase”, només 7 corresponien a l’espècie humana. Per a definir la família inequívocament es va fer un BLAST (Blastp, protein-protein) amb dues proteïnes per separat, cadascuna pertanyent a una classe diferent per comprovar que en els resultats apareixien les mateixes proteïnes amb scores similars (paràmetres: no-redundant, matriu Blosum 62, pairwise alignment). Així, es va triar la Adh2, pertanyent a la Classe I i la Adh6, pertanyent a la Classe V. S’observà que en ambdós casos l’score queia bruscament d’uns 400bits a uns 60-80 bits, en arribar a la seqüència de la Sorbitol deshidrogenassa, de manera que quedava establert el límit de pertanyença a la família. L’enzim sorbitol deshidrogenassa va ser usat per fer l’anàlisi com a control negatiu per comparar els membres de la familia.
Aquesta mateixa cerca en blast ens tornà un domini conservat en totes les seqüències trobades, el domini d’unió a zinc, característic:
![]()
Aquest domini l’estudiàrem més en detall fent cerques en
diferents bases de dades per a comparar la informació: Prosite,
Interpro, Reverse
Position specific Blast i Pfam. En
totes, el domini d’unió a Zn és el que indiscutiblement es troba conservat; Prosite ens tornava una mitjana de 5 dominis diferents per a cada seqüència, però podria
tractar-se de falsos positius, ja que aquesta base de dades fa prediccions, i
és per això que ens basàrem únicament en el domini
caracteritzat en Pfam per a
continuar la nostra anàlisi.
El pròxim pas consisteix en aliniar les seqüències
mitjançant el programa Clustal W,
d’aquesta manera obtenim una nova comprobació de similaritat
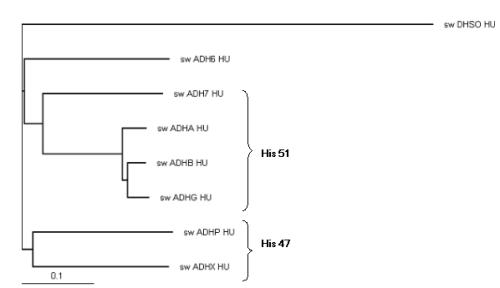
i un arbre, que ens dóna una idea de com s’agrupen les
diferents proteïnes i que permetrà un anàlisi funcional.
Els substrats varien en les diferents Adh (veure Taula de
Informació general). No observem una relació
directa entre els tipus de substrat i la similaritat de seqüència; en canvi, sí
que observem un agrupament relacionat amb la posició del residu crític de His.
L’associació obtinguda respon al següent esquema: