INTRODUCCIÓ
Miniopterus natalensis
Descripció
Miniopterus natalensis és una espècie de ratpenat que pertany a la família dels minioptèrids. És un animal carnívor que s'alimenta d'animals invertebrats. El seu pes oscil·la entre els 10 g i la seva longitud amb les ales esteses és de 45 cm.
Taxonomia
|
 |
Hàbitat i ecologia
Aquesta espècie habita en zones semidesertes amb climes secs, zones humides de la sabana i en zones amb vegetació mediterrània amb alta presència d'arbusts.
Distribució geogràfica
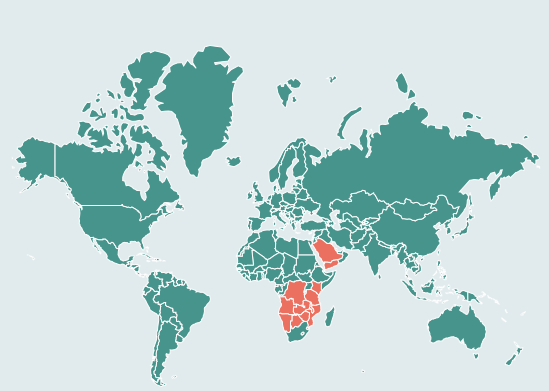
Podem trobar Miniopterus natalensis a l'Àfrica. Concretament és natiu de l'Angola, Botswana, la República Democràtica del Congo, Kènia, Lesotho, Malawi, Moçambic, Namíbia, Aràbia Saudí, Swazilàndia, Tanzània, República Democràtica del Yemen, Zàmbia i Zimbabwe.
Per a mes informació sobre aquesta espècie consultar la seva pàgina de
Selenoproteïnes
Què són les selenoproteïnes?
El seleni és un micronutrient essencial de gran importància biològica [7], que actua per mitjà de la selenocisteïna (Sec), aminoàcid que s’incorpora al centre catalític d’unes proteïnes anomenades selenoproteïnes [7, 8]. La selenocisteïna s’incorpora a les proteïnes davant d’un codó UGA, conegut prèviament únicament com a codó stop, i es considera el 21è aminoàcid [8]. La seva estructura és com la de la cisteïna, però es diferencien per un sol àtom, presenta seleni en lloc de sofre. Malgrat tots dos tenen propietats químiques molt semblants, la Ka de la selenocisteïna i la seva major activitat nucleofílica la fan molt més reactiva [9]. Com a conseqüència de la seva gran homologia amb la cisteïna, a les cèl·lules no hi ha un reservori de selenocisteïnes, ja que es podrien incorporar erròniament en lloc d’una cisteïna. A més, igual que tots els compostos amb seleni, reacciona amb l’oxigen i la tioredoxina i tioredoxina reductasa de mamífers, resultant en una ràpida oxidació de NADPH i la formació d’espècies reactives amb oxigen [9].
Biosíntesi
La incorporació d’aquestes proteïnes es dóna tan en bacteris com en eucariotes, però ens centrarem només en els segons.
Per poder incorporar el seleni ha d’estar en forma Se2-. Les formes inorgàniques ho fan gràcies a les vies de la glutatió-glutaredoxina i tioredoxina, mentre que les formes orgàniques són metabolitzades per la selenocisteïna liasa (Scly) o per trans-selenació. Aleshores, es produeix selenofosfat (SePO33-) en una reacció catalitzada per la selenofosfatasa sintasa 2 [10].
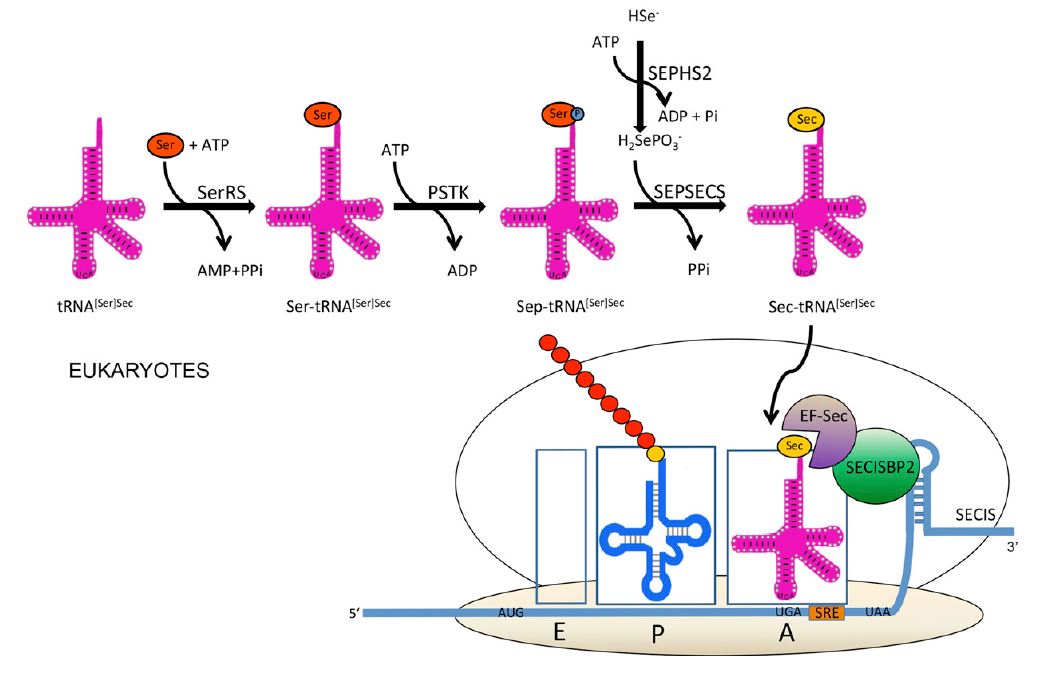
La selenocisteïna s’incorpora a les proteïnes gràcies al seu tRNA (tRNA[Ser]Sec) que, com hem dit, codifica pel codó UGA [10]. Aquest codó codifica per la Sec i no per un stop perquè al mRNA de la selenoproteïna hi ha una seqüència d’inserció de selenocisteïna (SECIS), un loop que actua en cis, a la posició 3’ de la regió no traduïda (UTR) del mRNA de la selenoproteïna, plataforma per la unió de les proteïnes al RNA [8, 10, 11]. En alguns mRNA de selenoproteïnes i prop del codó UGA, també podem trobar un element de redefinició de la selenocisteïna (SRE), un reminiscent d’un SECIS bacterià [8].
El SECIS forma complexes amb dos elements: el factor d’elongació específic de Sec (eEF-Sec) i la proteïna d’unió al SECIS 2 (SBP2), que permeten seguir l’elongació de la traducció. A més, els SECIS també s’uneixen a altres proteïnes, com L30 ribosomal, un factor d’iniciació eucariota (eIF4A3) i la nucleolina [10].
La eEF-Sec interacciona amb la SBP2 per reclutar la tRNA[Ser]Sec al ribosoma i seguir la síntesi de la proteïna. A més, en alguns mRNA de selenoproteïnes també podem trobar el que es coneix com a element de redefinició de la selenocisteïna (SRE), prop del codó UGA, un reminiscent d’un SECIS bacterià [8].
Les selenocisteïnes son sintetitzades directament unides al seu tRNA. D’aquesta manera, la serina és carregada en el Ser-tRNA[Ser]Sec per la seril tRNA sintasa. A continuació és fosforilada per la PSTK, per donar lloc a la fosfoseril(Sep)-tRNA[Ser]Sec i, finalment, es converteix en Sec-tRNA[Ser]Sec per l’acció de la selenocisteïna sintasa, utilitzant com a substrat la selenofosfatasa [8].
Evolució
Les selenoproteïnes les podem trobar en bacteris, arqueobacteris i eucariotes, però no en plantes ni llevats [11]. Tot i això, moltes espècies d’eucariotes i procariotes no tenen selenoproteïnes, però sí que tenen proteïnes homòlogues, sense que comporti cap desavantatge evolutiu [8, 12]. Aquestes proteïnes homologues tenen cisteïna en lloc de selenocisteïna i existeixen per la majoria de selenoproteïnes [11].
Les diferents famílies d’aquest tipus de proteïnes tenen una distribució filogenètica molt diferent: les llombrius tenen només una selenoproteïna, les mosques tres, els eucariotes en tenen 25 -tot i que la distribució varia molt entre els diferents tàxons- [11] i un màxim de 38 en els peixos [13]. També cal destacar que, per exemple, hi ha selenoproteïnes que només es troben en procariotes, mentre que altres només en eucariotes. A més, algunes famílies tenen una distribució molt restringida i no les podem trobar a altres espècies, ni tan sols amb la forma amb cisteïna [12].
Si ens centrem en el cas dels vertebrats, es defineix el vertebrat ancestral amb 28 selenocisteïnes i el mamífer amb 25. Gràcies a duplicacions (20 casos), delecions (10 casos) i substitucions de les selenocisteïnes per cisteïnes (12 casos), s’aconsegueix la complexa distribució entre totes les espècies [12].
Actualment, s’han trobat 45 subfamílies de selenoproteïnes en vertebrats, sent 27 d’elles exclusives d’aquest subfílum. 28 d’aquestes proteïnes es troben en mamífers -encara que cap espècie les té totes i com a molt se’n presenten 25 en una mateixa- i 41 en els peixos -sent el peix zebra el que en té més, 38-. Les següents 21 selenoproteïnes es troben a tots els vertebrats: GPx1-4, TR1, TR3, Dio1-3, SeIH, SelI, SelK, SelM, SelN, SelO, SelP, MsrB1, SelS, SelT1, SelW1, Sep15. Les altres es troben només en alguns llinatges [13].
Identificació de selenoproteïnes al genoma
Com que el codó UGA codifica tant per un stop com per una selenocisteïna, la identificació de noves selenoproteïnes amb un programa automàtic és molt complicada i ha comportat errors en l’anotació d’alguns genomes [11]. Això es deu al fet que el progràma assigna automàticament a aquest codó la finalització de la proteïna quan, en aquests casos, no és així.
Per anotar-les correctament, el més fàcil és comparar el genoma que estem estudiant amb la seqüència de selenoproteïnes més propera filoganèticament ja conegudes. A més, també s’han de buscar els elements SECIS, a la regió 3’UTR del gen, com bé ja hem dit anteriorment. Tot i que aquesta aproximació ha sigut d’utilitat en diferents casos, la baixa especificitat en complica la cerca, ja que donen un gran nombre de prediccions quan s’estudien genomes eucariotes [11]. D’aquesta manera, podrem identificar tant les selenoproteïnes com les seves homòlogues amb cisteïna.