Introducció
LES SELENOPROTEÏNES
Les selenoproteïnes són un tipus de proteïnes que incorporen a la seva seqüència un aminoàcid anomenat Selenocisteïna (Sec), el qual correspon a l’aminoàcid 21, que es representa amb la lletra U. Aquest aminoàcid és idèntic a una cisteïna(Cys), però ha substituït el radical de sofre (SH) per un de seleni (SeH).
En els organismes, el seleni no es troba mai en forma lliure, sinó lligat a aquest tipus de proteïnes. Aquest mecanisme ha estat desenvolupat pels éssers vius per tal d'evitar la intoxicació per seleni i així poder-lo mantenir com a element essencial. Les selenoproteïnes acostumen a ser enzims redox, implicats en reaccions d'oxidació-reducció, gràcies a l'alta reactivitat de l'àtom de seleni situat al centre catalític, i per tant, tenen una possible capacitat protectora antioxidant. [7]
El seleni i el sofre ocupen la mateixa columna a la taula periòdica dels elements, fet que els confereix propietats químiques similars. Aquest fet explica que en la recerca de gens ortòlegs de selenoproteïnes en altres organismes, a vegades es troben Cys enlloc de Sec, ja que en ambdós casos les proteïnes tenen funcions similars.
L’aminoàcid selenocisteïna és codificat pel codó UGA el qual és, normalment, un codó de terminació de la traducció. És per això que, per a què es doni una re-codificació del codó UGA, es necessita una maquinària específica.
- La SECIS Binding Protein (SBP2) és una proteïna llarga, de 854 aminoàcids. La seva regió N-terminal és essencial per a la funció d’incorporar selenocisteïnes. Forma part d’un complex de proteïnes implicat en la síntesi de selenocisteïnes i participa en la incorporació dels factors eEFsec, SECp43, SPS1 i SLA (explicat en l'apartat Síntesi de Selenocisteïna). Té altres dominis funcionals com una senyal de localització nuclear rica en lisines (NLS) en la regió N-terminal, un NLS potent en la regió C-terminal, dues senyals d’exportació nuclear riques en leucina (NES), un domini funcional d’unió a elements SECIS i un domini d’interacció a ribosomes.
- El factor d’elongació específic de selenoproteïnes (eEFsec) té una funció complexa. Participa tant en la biosíntesi com en la incorporació de selenocisteïnes, i a més, interacciona amb altres proteïnes i RNA de la maquinària de síntesi de selenoproteïnes, com les dues isoformes de la tRNAsec i SBP2. En el cas de tRNAsec, és necessari perquè SBP2 i eEFsec formin un complex tant al nucli com al citoplasma
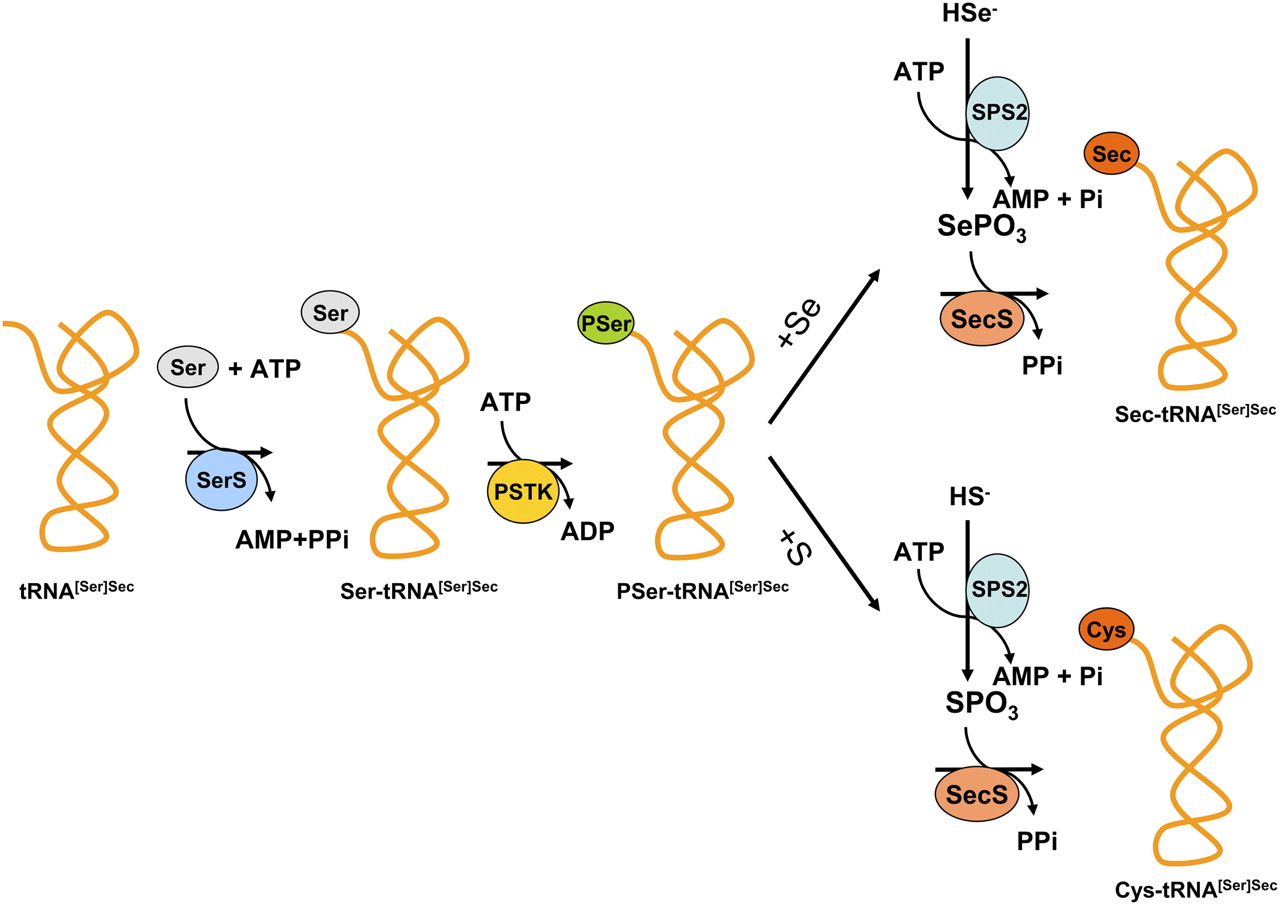
- La tRNAsec regula la síntesi de totes les famílies de selenoproteïnes, ja que tradueix el codó UGA a selenocisteïna. La fosfoseryl-tRNAsec kinasa fosforila específicament la molècula seryl-tRNAsec en la síntesi de selenocisteïna
Maquinària i procés de síntesi de les Selenoproteïnes
La majoria d’aquests elements són característics i exclusius dels organismes que contenen selenoproteïnes i poden ser utilitzats en la cerca d’aquestes proteïnes a través de diferents genomes. [23]
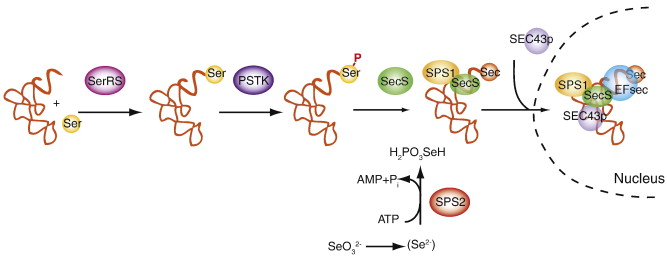
- La Selenofosfat Sintetasa 1 (SPS1) que com forma un complex amb proteïnes involucrades en la biosíntesi de selenocisteïna, es creu que probablement no té un paper essencial en el metabolisme de les selenoproteïnes. D’altra banda, es pensa que podria tenir un paper en el reciclatge de selenocisteïna. De totes maneres, la seva funció no implica la síntesi de selenofosfat.
- La Selenofosfat Sintetasa 2 (SPS2) s’ha demostrat en diversos estudis que la SPS2 sintetitza selenofosfat a partir de seleni i que probablement està involucrada en la síntesi de novo. A més, és essencial per generar el donant de seleni per la síntesi de selenocisteïna
- la Selenocysteine Associated Protein (SEC43p) reconeix motius de RNA gràcies a que té dos dominis d’unió a ribonucleoproteïna (RNPs). A més, forma un complex amb el tRNAsec, participa en la síntesi de selenocisteïna, en la regulació dels nivells d’expressió de tRNAsec metilat i és estabilitzador del complex eEFsec-SBP2-tRNAsec.
- La fosfoseril-tRNA kinasa (PSTK) és una proteïna que es troba altament conservada al llarg de la evolució, fet que suggereix que juga un paper important en la biosíntesi i/o regulació de selenoproteïnes.
Maquinària i síntesi de la Selenocisteïna
La Selenocisteïna se sintetitza directament en el seu tRNA perquè no es troba lliure a la cèl•lula, i per a la seva formació, seran necessaris els següents elements:Totes les proteïnes esmentades tindràn un paper en el procès de biosíntesi de la Sec. En primer lloc una serina s’uneix mitjançant l'acció de la seril-tRNA sintetasa al tRNA. A continuació, la PSTK fosforila la Serina unida al tRNA. Més tard l’enzim Selenocisteïna Sintasa (SLA/LP), amb l’ajuda de la SEC43p i les SPS1 i 2, transformaràn la serina en una Selenocisteïna i crearan la seqüència EF, on s’unirà el tRNA al factor de transcripció EFsec. [1]
Les Selenocisteïnes i el seu problema en l’anotació de genomes
El principal problema d’aquest tipus de proteïnes és a l’hora d’identificar els seus gens amb un programa automàtic. De manera sistemàtica, aquest reconeixerà un codó UGA com un codó de stop.Això fa que normalment les selenoproteïnes estiguin mal anotades i per tant, això també repercuteix en l’anotació del genoma de l’espècie.
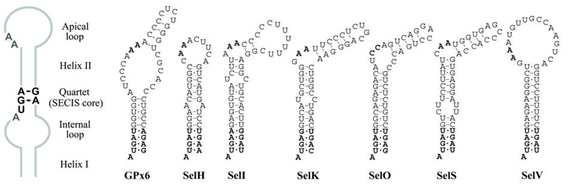
Una manera de trobar aquest tipus de proteïnes és mitjançant la identificació de SECIS a la regió 3’, amb l'objectiu de saber quins codons UGA no codifiquen per codons de stop sinó per Selenocisteïnes. El principal problema, és que no s’ha trobat cap patró de seqüència SECIS, cosa que dificulta la seva recerca i s’ha de dur a terme mitjançant programes especials.
Inicialment es creia que entre tots ells només tenen conservats un quartet de nucleòtids (AUGA), però posteriorment s'han identificat quartets diferents. Els SECIS que tenen el quartet AUGA s'anomenen canònics i la resta no canònics.
S’ha observat que tot i que la seqüencia no és la mateixa, l'estructura tridimensional sí que és la mateixa en tots els casos (loop-helix-loop), i per tant, aquesta serà l’estratègia per trobar aquests SECIS. Els programes utilitzats buscaran seqüències que es puguin plegar i formar aquesta estructura 3D coneguda. PatScan és un dels programes que es fan servir. S'introdueix el patró estructural i un conjunt de seqüències i el programa mostra quines d’aquestes poden adoptar aquesta estructura. Per fer una predicció eficient de la localització de selenoproteïnes es sobreposen la informació d'elements SECIS amb la localització d'exons del gen que estem estudiant. [20] [3]
MUSTELA PUTORIUS FURO
L'objectiu del nostre estudi és l'anotació de les selenoproteïnes del genoma de la Mustela putorius furo, genoma anotat recentment i que encara no ha estat corregit.
La Mustela putorius furo, més comúnment anomenada fura, és un mamífer carnívor de la varietat domèstica del furó comú. Aquests mustèlids solen tenir un pelatge marró, negre, blanc o mesclat. Tenen un mida d'aproximadament uns cinquanta centímetres (incloent-hi una cua d'uns tretze centímetres) i pesen aproximadament un quilogram. Poden arribar a viure entre set i deu anys. Aquest mamífer pertany a l’ordre Carnivora de la familia de la Mustelidae.

CLASSIFICACIÓ TAXONÒMICA
DE LA MUSTELA
Regne: Animalia
Fílum: Chordata
Classe: Mammalia
Ordre: Carnivora
Família: Mustelidae
Gènere: Mustela
Espècie: M. putorius
Subespècie: M. p. furo
Tornar a l'inici de la pàgina
