
Resultats i Discussió de G. niphandrodes
Fent el tBlastn de la query d’EhSEP2 d’Emiliania huxleyi contra tot el genoma de G.niphandrodes s’obtenen 5 hits estadísticament significatius (E.value < 1·10-4).
Hit 1
L’anàlisi amb Exonerate d’aquest scaffold troba dos possibles alineaments, mentre que Genewise prediu un segment del primer alineament.
- El primer és un alineament revers que prediu un gen de 2 exons i 1 intró en el que la selenocisteïna de la query es canvia per una cisteïna al primer exó; per tant s’hauria trobat un gen homòleg amb cisteïna. El resultat del Tcoffee és molt bo i quan es fa un Blastp amb la proteïna predita contra una base de dades no redundant de proteïnes del NCBI els hits trobats corresponen a proteïnes de la família de les Tioredoxina-Disulfit Isomerasa (TDI).
- El segon alineament prediu un gen també a la cadena reversa que presenta un sol exó on la selenocisteïna s'alinea amb una cisteïna. El Tcoffee obtingut presenta un resultat molt òptim però més curt que l’anterior. Tot i això, el Blastp amb NCBI retorna proteïnes de la família de les TDI.
El programa SECISearch no ha trobat cap element SeCIS. Els dos alineaments predits per Exonerate se solapen entre ells, de tal manera que l'exó del segon alineament se situa a l’intró predit al primer alineament, tal i com mostra la imatge:
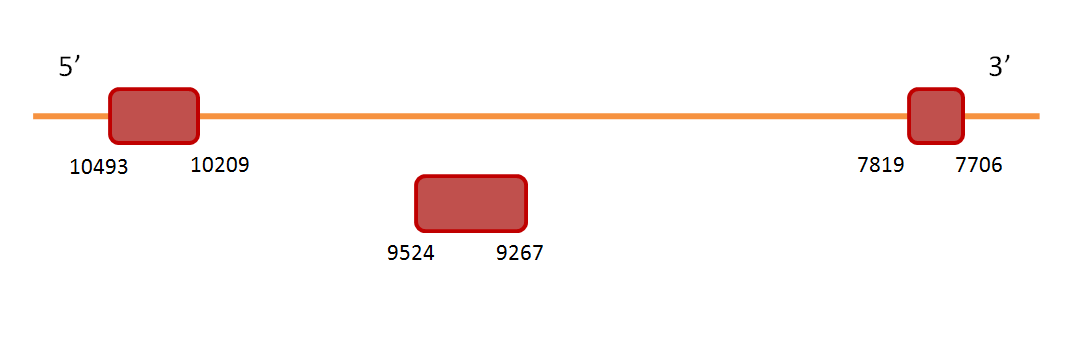
Figura 1.
Aquest resultat fa pensar en un cas d’isoformes en que la proteïna final podria estar codificada per diferents combinacions d’exons. És per això que manualment s'ha creat una proteïna formada per la combinació de l’exó predit al segon alineament amb el segon exó del alineament. El resultat del Tcoffee obtingut per aquesta proteïna és tant òptim com el de l’alinemant 2 però més llarg. Les proteïnes trobades quan es fa un Blastp d’aquesta proteïna amb NCBI són també de la família de les TDI.
Amb tot, es conclou que és probable que en aquest scaffold hi hagi un sol gen homòleg a EhSEP2 amb cisteïna que presenta 3 exons i que la proteïna final s'obté de la combinació per splicing alternatiu de l’exó 1 o el 2, unint-lo al final amb l’exó 3.
Hit 2
Exonerate i Genewise prediuen un mateix alineament en aquest scaffold, que consisteix en un gen amb un sol exó situat a la cadena reversa i en el que s'ha canviat la selenocisteïna per una cisteïna. El Tcoffee obtingut és bo i quan es fa el Blastp amb NCBI les proteïnes que es retornen són de la família de les TDI.
El programa SECISearch ha reportat 2 elements SeCIS consecutius. El primer es troba unes 8 Kb downstream del gen predit, i el segon aniria a continuació del SeCIS predit anteriorment.
Els resultats permeten predir amb alta probabilitat la presència d'un gen homòleg amb cisteïna d’EhSEP2 en aquest scaffold. El fet d'haver trobat element SeCIS podria ser degut a una predicció errònia de SECISearch, hipòtesis reforçada pel fet que els dos elements SeCIS estan localitzats massa a prop per poder contenir un exó entremig. Una altra possibilitat és que hi hagi un altre gen a continuació que codifiqui per una selenoproteïna.
Hit 3
Mentre Exonerate troba un alineament revers en aquest scaffold, Genewise obté el mateix però l’allarga una mica més per l'extrem 5'. Així, el gen predit per Genewise està format per un sol exó i la selenocisteïna s'alinea amb una cisteïna. El resultat del Tcoffee és força bo i, usant la proteïna predita per fer un Blastp amb NCBI, els hits obtinguts són Disulfit Isomerases de diversos organismes. No s’ha trobat elements SeCIS amb SECISearch.
Els resultats avalen que, amb força probabilitat, en aquest scaffold s’hi troba un gen homòleg a EhSEP2 que ha canviat la selenocisteïna per una cisteïna.
Hit 4
L’anàlisi amb Exonerate no retorna cap alineament en aquest scaffold, però Genewise sí que prediu un gen a la cadena reversa format per un sol exó, en el qual s’ha canviat la selenocisteïna de la query per una cisteïna. El resultat del Tcoffee és bo i la proteïna predita permet detectar altres proteïnes de la família de les TDI en fer un Blastp a l’NCBI. SECISearch no ha torbat cap element SeCIS.
Els resultats obtinguts permeten predir amb certa seguretat que en aquest scaffold hi ha un gen homòleg d'EhSEP2 amb cisteïna.
Hit 5
Exonerate no troba cap alineament, en canvi Genewise sí que en prediu un. El gen obtingut es localitza a la cadena directa, està format per un sol exó i és un homòleg d’EhSEP2 amb cisteïna. El Tcoffee obtingut és molt curt, però aquesta petita proteïna predita és suficient perquè en fer un Blastp amb NCBI ens retorni proteïnes de la família de les TDI. No s’ha trobat cap element SeCIS amb SECISearch.
Per tant es pot afirmar amb força probabilitat que hi ha un gen homòleg d'EhSEP2 amb cisteïna en aquest scaffold.
Conclusió
En conclusió, s’ha vist que G.niphandrodes no conté cap EhSEP2 com a selenoproteïna, sinó que tots els gens homòlegs predits contenen cisteïna en el seu lloc. En total s’han pogut predit 5 gens homòlegs a EhSEP2 repartits ens diferents scaffolds del genoma d’aquest organisme.




