Discussió
| SelI | MSP | |
| Albugo laibachii_Nc14 | ||
| Astrammina rara | ||
| Crithidia fasciculata | ||
| Dictyostelium discoideum_AX4 | ||
| Dictyostelium fasciculatum | ||
| Fragilariopsis cylindrus | ||
| Gregarina niphandrodes | ||
| Ichthyophthirius multifiliis_strain_G5 | ||
| Leishmania donovani_BPK282 | ||
| Leishmania tarentolae | ||
| Phytophthora capsici | ||
| Physarum polycephalum | ||
| Sphaeroforma arctica | ||
| Trypanosoma congolense |
SelI
A. laibachii Nc14
Els resultats mostren que hi ha dues pautes de lectura a partir de les quals s'obtenen dues proteïnes que s'alineen amb una elevada similitud amb la query. Cada proteïna s'alinea amb una regió diferent de la query i, a més, aquestes regions són complementàries. Això ens fa pensar que s'hagi pogut produir un error de seqüenciació que ha fet canviar la pauta de lectura, fet que explicaria perquè obtenim dues pautes de lectura amb el mateix nombre de codons STOP i perquè les proteïnes predites seguint dues pautes de lectura diferents s'alineen amb una elevada identitat amb regions diferents de la query. Així doncs, només es pot alinear la totalitat de la proteïna combinant els alineaments obtinguts amb les dues pautes de lectura diferents.
Tenint en compte els resultats i el possible error de seqüenciació, podem deduir que hem trobat la selenoproteïna SelI en A.laibachii. El motiu pel qual pensem que Exonerate no ens detecta aquesta regió com a exònica i hem hagut d'allargar manualment el darrer exó per tal d'aconseguir alinear la selenocisteïna amb un gap és perquè llegeix un codó STOP precedit d'una regió poc conservada, de manera que finalitza l'exó precoçment. A través de l'alineament que obtenim amb Tcoffee podem pensar que A.laibachii Nc14 ha patit una inserció propera a l'extrem C-terminal.
Resta de genomes
En general, hem obtingut hits significatius en el tBLASTn per a tots els genomes, excepte A.rara. Tanmateix, cap dels alineaments contenia la selenocisteïna però com eren bons hits els hem continuat analitzant. Ni amb Exonerate, Genewise o Tcoffee hem aconseguit alinear la selenocisteïna. En alguns casos hem intentat allargar manualment el darrer exó predit per Exonerate per intentar determinar amb Tcoffee si les regions properes a la selenocisteïna s'alineaven amb certa similitud amb la query. Tanmateix, els resultats són semblants als que obteníem sense allargar els exons.
Tcoffee ens dóna, per alguns genomes, resultats força bons i ens alinea regions extenses de la proteïna amb scores força elevats. En altres genomes, però, només alinea regions molt curtes i creiem que això no és gaire significatiu.
Després d'analitzar els genomes dels 14 protistes i d'haver fet un BLASTp amb les proteïnes predites contra la base de dades de l'NCBI, hem arribat a la conclusió que cap dels genomes analitzats té gens que codifiquin per a la selenoproteïna SelI o per a homòlegs amb cisteïna, excepte A.laibachii Nc14. Tanmateix, la majoria dels genomes presenten regions d'homologia a la selenoproteïna SelI.
Se sap que SelI té un domini CDP-alcohol fosfatidiltransferasa i que el domini de la superfamília de les CDP-alcohol fosfatiltransferasa presenta certa homologia amb altres dominis transferasa, tals com etanolamina fosfotransferasa, 1,2-diacilglicerol etanolamina i colinatransferasa. Aquesta és la raó per la qual creiem que la cerca de SelI en aquests organismes ens retorna proteïnes amb regions d'homologia amb aquesta selenoproteïna que es corresponen amb dominis transferasa. Tanmateix, en cap cas trobem selenoproteïnes, ja que la regió C-terminal de SelI, on es troba la selenocisteïna, no està conservada en cap d'aquests organismes.
MSP
D. discoideum AX4
Tal i com esperàvem, hem trobat la selenoproteïna MSP en el genoma de D.discoideum AX4 ja que la query que hem utilitzat és del mateix organisme. L'alineament del Tcoffee no és perfecte ja que es detecta una regió de 11 aminoàcids presents en el genoma però no a la query. Això pot ser degut a que l'organisme a partir del qual s'ha extret la query i el que s'ha seqüenciat per obtenir el genoma són diferents i presenten certes difències en la seves seqüències.
D.fasciculatum
En els resultats obtinguts amb l'alineament de Tcoffee podem observar que la proteïna predita en D.fasciculatum presenta una elevada identitat amb la query exceptuant la regió N-terminal que està lleugerament menys conservada. A més, trobem les dues selenocisteïnes alineades amb codons STOP. D'altra banda, el BLASTp amb la proteïna predita contra la base de dades de NCBI ens retorna com a hits una membrane selenoprotein (MSP) de D.discoideum_AX4 (la query inicial) així com de D.fasciculatum. Tot això ens fa concloure que D.fasciculatum codifica per a la selenoproteïna MSP.
P.polycephalum
A partir dels resultats obtinguts no podem estar segurs que el genoma de P.polycephalum codifiqui per la selenoproteïna MSP, ja que només hem trobat una regió central de la proteïna conservada amb una bona identitat i que, a més, presenta una de les dues selenocisteïnes. Tanmateix, no s'ha aconseguit alinear la primera selenocisteïna present a la regió N-terminal. Al realitzar un BLASTp amb la selenoproteïna predita de P.polycephalum contra la base de dades de NCBI, els millors hits que ens reporta són membrane selenoprotein, és a dir, MSP. Per això creiem que P.polycephalum codifica per a la selenoproteïna MSP però que aquesta ha degenerat en algunes regions no importants per la funció d'aquesta proteïna i, a més, ha perdut una de les dues selenocisteïnes.
S.arctica
Els resultats obtinguts ens mostren que S.arctica presenta unes petites regions amb certa similitud amb la selenoproteïna MSP, però com que aquestes regions no es corresponen amb les posicions amb selenocisteïnes ni són molt extenses, podem concloure que S.arctica no codifica per a la selenoproteïna MSP.
Altres genomes
Amb la resta de genomes no hem obtingut cap hit significatiu amb el tBLASTn. Per aquest motiu, no hem continuat l'anàlisi i concloem que no tenen la selenoproteïna MSP.
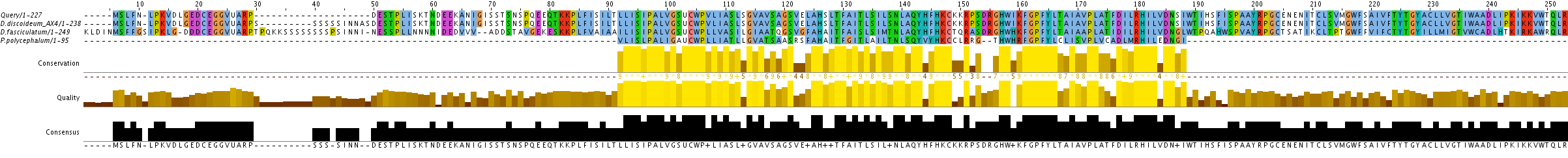
Alineament múltiple amb Tcoffee
Aquest alineament múltiple és l'alineament simultani de la query de la proteïna MSP (de D.discoideum_AX4) que hem utilitzat durant tot el procés d'aquest treball, la selenoproteïna de D.discoideum_AX4 que hem predit en aquest treball, la selenoproteïna MSP predita de D.fasciculatum i la selenoproteïna MSP predita de P.polycehalum.
Tal i com podem observar a la imatge de l'alineament múltiple, els aminoàcids selenocisteïna es troben ocupant exactament les mateixes posicions en les cinc proteïnes de l'alineament, en excepció de P.polycephalum, que com ja havíem predit, ha perdut la primera selenocisteïna i una petita regió final de la selenoproteïna MSP. Alhora, si observem la resta de residus, podem apreciar que la seqüència es troba força conservada en els tres genomes, amb una similitud bastant elevada a la totalitat de la proteïna, menys per P.polycephalum que concerva la regió central de la proteïna.
Maquinària de síntesi i traducció de selenoproteïnes
En general, podem dir que en la majoria de protists en els quals hem trobat alguna de les selenoproteïnes MSP o SelI també hem pogut determinar que presenten els gens que codifiquen per a algunes proteïnes implicades en la maquinària de traducció i síntesi de selenoproteïnes. Com sabem, aquesta maquinària és necessària per a poder obtenir les esmentades proteïnes i, per tant, haurien d'estar presents en els genomes de les espècies en que vulguem demostrar l'existència de selenoproteïnes
L’anàlisi bàsic per a la cerca de maquinària de traducció s’ha fet en tota la llista de protists d’aquest any. La taula s'ha omplert en base als resultats obtinguts i seguint els següents criteris:
- Resultats de BLAST amb uns hits estadísticament significatius.
- Prediccions amb Exonerate i/o amb Genewise de l'estructura exònica dels gens trobats en la regió delimitada pel hit.
- Alineacions amb alta identitat, utilitzant Tcoffee, de la query amb la que es fa la cerca i la proteïna predita a partir dels programes anteriors
S’ha elaborat la següent taula, on es veu de manera més o menys aproximada la possibilitat de la presència de les proteïnes implicades en la traducció i síntesi de selenoproteïnes en cada organisme.
| SPS2 | SBP2 | Pstk | Secp43 | SecS | eEFsec | |
| Albugo laibachii_Nc14 | ||||||
| Astrammina rara | ||||||
| Crithidia fasciculata | ||||||
| Dictyostelium discoideum_AX4 | ||||||
| Dictyostelium fasciculatum | ||||||
| Fragilariopsis cylindrus | ||||||
| Gregarina niphandrodes | ||||||
| Ichthyophthirius multifiliis_strain_G5 | ||||||
| Leishmania donovani_BPK282 | ||||||
| Leishmania tarentolae | ||||||
| Phytophthora capsici | ||||||
| Physarum polycephalum | ||||||
| Sphaeroforma arctica | ||||||
| Trypanosoma congolense |
A l'apartat de resultats ja s'ha comentat per als protists Albugo laibachii Nc14, Sphaeroforma arctica, Physarum polycephalum, Dictyostelium fasciculatum i Dictyostelium discoideum AX4 (enllaç a aquests resultats) la possible presència de cadascuna de les proteïnes de la maquinària.
A més d'haver trobat la maquinaria de transcripció i síntesi en els organismes que ens interessaven, també podem extreure algunes conclusions addicionals a partir d'aquesta taula. Així doncs, podem observar que protists com F.cylindrus, P.capsici, T congolense o Leishmania presenten quasi totes les proteïnes bàsiques implicades en la producció de selenoproteïnes. Això pot indicar que, tot i que no haguem trobat les selenoproteïnes SelI i MSP en concret, existeix la possibilitat que aquests organismes codifiquin per a selenoproteïnes i que, per tant, en puguin presentar d’altres famílies. D’altra banda, trobem organismes com G.niphandrodes i A.rara que no presenten cap de les proteïnes necessàries per a la traducció i síntesi de selenoproteïnes. Això, unit al fet que tampoc s’ha revelat la presencia de les selenoproteïnes SelI i MSP, ens permet hipotetitzar que potser aquestes espècies en concret no presentin cap selenoproteïna en el seu genoma, ja que els hi manca les eines moleculars necessàries per a sintetitzar-les.
Cerca d'elements SECIS
En aquest estudi, la cerca de SECIS s’ha realitzat en les selenoproteïnes trobades però després s’ha ampliat a la resta dels protistes del nostre estudi. Els resultats mostren que no hi ha cap element SECIS si utilitzem els paràmetres predeterminats que ens dóna el programa. Hem utilitzat els fitxers de fatasubseq de la cerca de selenoproteïnes que haviem realitzat a partir dels hits trobats en tots els genomes en que apareixien hits raonablement bons per a que predis si hi havia algun element, i no hi ha hagut cap resultat. Com no hem modificat els paràmetres, el fet de que no haguem trobat elements SECIS no vol dir que no hi hagin, sinó que pel caràcter restrictiu de les variables que es tenen en compte en aquest software i amb les seqüències introduïdes no hem obtingut cap resultat.