Identificación de selenoproteínas en protistas
Brugada V, Frías S, Fuentes P, García C.
El Selenio es un elemento esencial para los organismos
El selenio se considera un nutriente esencial en la dieta de los animales, microorganismos y algunos eucariotas, aunque es un elemento poco abundante. Es un componente esencial en muchas vías metabólicas: metabolismo de la hormona tiroidea, sistemas de defensa antioxidantes y en la función inmune. Puede causar problemas tanto por exceso como por defecto. La deficiencia de selenio en sangre tiene implicaciones en problemas potenciales de la salud pública. Por otro lado, el exceso de selenio también puede ser tóxico y, por ello, los organismos han generado mecanismos moleculares para prevenir la toxicidad del selenio.
Las selenoproteínas
Las selenoproteínas son proteínas que se encuentran en los tres reinos que incorporan el aminoácido selenocisteína en su secuencia aminoacídica.
Las selenoproteínas se caracterizan por presentar una selenocisteína, codificada por un codón UGA
La primera selenoproteína se descubrió en 1973 y desde aquel momento se han identificado más de 30 familias. Hoy en día, se sigue investigando para caracterizar selenoproteínas en los tres reinos: Eucarya, Archea y Prokarya. No todos los organismos tienen selenoproteínas, pero podemos encontrar homólogos con cisteína. Por ejemplo, nos podemos encontrar con el caso de que en una especie de pez haya una selenoproteína, mientras que en humanos la proteína ortóloga presenta una cisteína.
Las selenoproteínas tienen un papel com enzimas redox y realizan reacciones de oxidación-reducción.
Síntesis de SelenoproteínasLas síntesis requiere de una maquinaria específica
Como hemos visto la codificación por el codón UGA presenta un problema, ya que también codifica para un codón STOP. Por esto se requiere de una estructura en el extremo 3' de la secuencia codificante para identificar el UGA como selenocisteína. Este elemento se denomina SECIS (Selenocystein Insertion Sequence) y consiste en una estructura secundaria tridimensional de RNA. Este elemento recluta a la proteína SBP2 (SECIS Binding Protein) que, a su vez, recluta el factor de elongación (Efsec). Efsec se encarga de reclutar el tRNA específico de selenocisteína. Así, el codón UGA codificará para una selenocisteína. La traducción seguirá hasta que el ribosoma reconozca el nuevo codón de terminación. Además hay otras proteínas que participan específicamente en la síntesis de selenocisteínas, destacando entre ellas la Selenophosphate Synthetase (SPS1 y SPS2), Selenocystein syntethase (SLA/LP), Phosphoseryl tRNA Kinase (PSTK) y Selenocysteine associated proteina (SECPP43). Concluímos así que el proceso de síntesis de selenoproteínas requiere de la participación de muchos genes.
Elementos SECIS
La presencia de un elemento SECIS es esencial junto con la maquinaria específica para la recodificación del UGA a una selenocisteína. Este elemento se encuentra en posición 3' con respecto a la región codificante. Cuando se realiza una modelo experimental de la estructura secundaria se pudo comprobar que existe un motivo de RNA compuesto por cuatro pares de bases consecutivos.
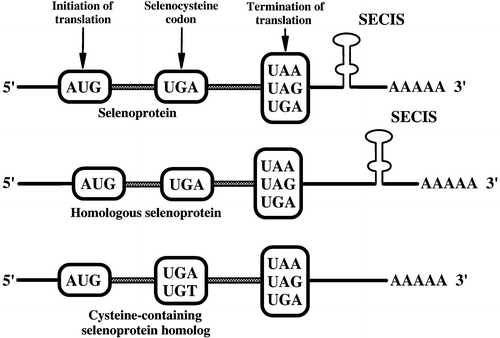
Como podemos ver en la imagen, para que sea una selenocisteína debe contener un codón UGA y un elemento SECIS en el extremos 3'. Se iniciría en la metionina y llegaría al UGA y se recodificará gracias a la presencia del elemento SECIS. Como vemos en la imagen del medio, nos encontramos como selenoproína homológa que contiene tambié el codón UGA con un elemento SECIS en 3'. Seguidamente, podemos ver que también se dan casos en la selenocisteína ha sido modificada por una cisteína y elemento SECIS desaparece; en este caso tenemos una proteína homóloga que contiene una cisteína. Finalmente, hemos de conocer la presencia de proteínas homólogas que contienen cisteína que también tienen el elemento SECIS. Éstas son selenoproteínas que evolutivamente han perdido su selenocisteína.
Nuestras Selenoproteínas
La selenoproteína R se trata de una proteína de bajo peso molecular que contiene átomos de selenio y de zinc. Forma parte de la familia de las proteínas metionina sulfóxido reductasa (MSR). Ésta cataliza estereoespecíficamente la reducción de residuos de metionina-R-sulfoxidos y pasa a ser llamada MsrB1, cuya familia, dentro de los mamíferos, también incluye las proteínas MsrA, MsrB2 y MsrB3. Éstas últimas, con una mayor localización en la mitocondria y el retículo endosplasmático, contienen en su posición catalítica el aminoácido Cys en lugar de Sec, lo que conlleva una menor actividad en estos homólogos.
SelR se distribuye ampliamente en los diferentes tejidos, aunque aparece en niveles más altos en el tejido hepático y en el tejido renal, ya que está descrito que son los tejidos más susceptibles al daño oxidativo por parte de las proteínas.
Se han descrito diferentes funciones para la SelR en los seres humanos, las cuales incluyen la protección contra la neurodegeneración, su papel en la viabilidad celular y una importante función en el daño oxidativo relacionado con el envejecimiento. Además, estudios recientes describieron el papel de MsrA en la modulación de la señalización celular de calcio/calmodulina kinasa II (CaMKII). Ésta última, contiene dos residuos de Met los cuales padecen oxidación dando lugar a la activación de esta importante proteína dentro de la ruta de señalización. Es decir, se puede decir que Sel R no sólo juega un papel en la mitigación del daño oxidativo de las proteínas en sus residuos de metionina, sino también un papel en la regulación de ciertas moléculas de señalización celular a través de la alteración del estado redox de determinados residuos de metionina.
La selenoproteína T se encuentra expresada durante el desarrollo embrionario y en la edad adulta. A nivel celular, se encuentra en el Retículo Endoplasmático fijado mediante un dominio hidrofóbico. Esta selenoproteína fue reconocida en humanos mediante un algorismo bioinformático que permite el reconocimiento de los elementos SECIS. También se ha encontrado en pez cebra y en ratón. Cabe destacar que su función es desconocida.
Aunque es posible que esta proteína se una a UDP-glucosa-UGTR, que es una proteína del retículo endoplasmático, está involucrada en la calidad del plegamiento de las proteínas.
La selenoproteína N (SelN) forma parte de la familia de la selenoproteínas, y ha sido el primer miembro que se ha visto que es responsable de desórdenes genéticos. Se trata de una glicoproteína que se encuentra en el retículo endoplasmático y se expresa en diversos tejidos (incluyendo músculo esquéletico, corazón, pulmón y placenta. Se expresa en altos niveles en el diafragma, por ello, cuando la proteína está mutada, los pacientes sufren problemas respiratorios. En definitiva, se expresa preferencialmente en células en proliferación y en tejidos fetales; y está involucrada en la formación del músculo.
Como todas las selenoproteínas, se caracteriza por la presencia de un átomo de selenio en su residuo selenocisteína.
El gen que codifica la SelN se denomina SEPN1. Mutaciones en este gen causan la miopatía relacionada con la Selenoproteína N (SEPN1-RM), que se manifiesta como Distrofia Congénita Muscular con Rigidez Espinal (RSMD1) y puede dar lugar a diversas patologías. Esta es una enfermedad autosómica recesiva.
Los protistas
El reino de los protistas está formado por un grupo heterogéneo de organismos eucariotas simples que no pueden incluirse dentro del reino de los animales, vegetales u hongos. No obstante, algunos protistas pueden parecerse o actuar igual que organismos del reino animal.
Los organismos del reino de lo protistas pueden presentar estructura unicelular, multicelular o colonial pero sin llegar a formar tejidos especializados. La mayoría son autótrofos, pero también pueden ser heterótrofos (por absorción) o una combinación de ambos. Generalmente son aerobios y pueden reproducirse de manera sexual (meiosis) o asexual (mitosis). Los procariotas son generalmente aeróbicos y se localizan principalmente en el medio acuático, no obstante, también pueden desarrollarse en ambientes terrestres húmedos.
Filogenia
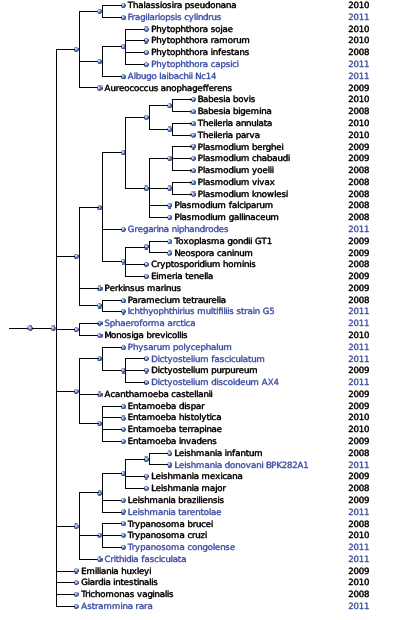
El reino protista es un grupo parafilético, ya que incluye al antepasado común de sus miembros, pero no a todos los descendientes el mismo. En la imagen inferior podemos ver el reino protista dentro del árbol filogenético.
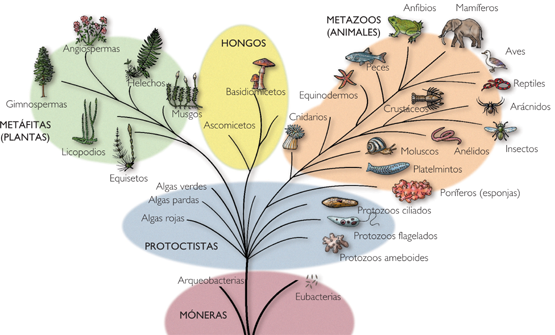
Para ver más información de los protistas, haced click aquí. En la imagen inferior se puede observar las relaciones filogenéticas de nuestros protistas con los estudiados en años anteriores.