Selenoproteínas
Una selenoproteína es cualquier proteína que incluya un residuo de selenocisteína. Las selenoproteínas existen en todas las principales formas de vida: eucariotas, bacterias y arqueas. En eucariotas, las selenoproteínas parecen ser muy comunes en el reino animal, pero no se han encontrado en el reino vegetal. Al estudiar arqueas y protistas, se observa que las selenoproteínas están presentes sólo en algunos linajes, mientras que están totalmente ausentes en otros. Estas observaciones han sido confirmadas por el análisis completo de los genoma de los organismos. (1)
Qué son:
Las seleneproteínas son un grupo de proteínas que se caracterizan por la presencia de selenio (Se) en uno de sus aminoácidos. Este corresponde al aminoácido 21, la selenocisteina (Sec). Para incorporarlo el mRNA de las selenoproteínas debe tener dos propiedades fundamentales:
- La presencia de uno o más codones UGA dentro de la región exónica que codifiquen para una selenocisteína.
- Una estructura secundaria helix-loop-helix-loop en el extremo 3'UTR.
Aunque los trabajos iniciales que descubrieron la biosíntesis y la función de las selenoproteínas se hicieron en bacterias, el interés de las mismas ha propiciado que actualmente el mayor volumen de información en el campo provenga de su estudio en eucariotas.
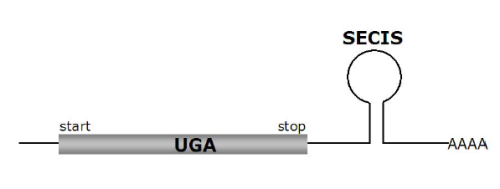
Figura 1
Biosíntesis:
La biosíntesis podría ser dividida en dos pasos:
1. Síntesis del aminoácido selenociesteína:
A diferencia del resto de aminoácidos, la Sec necesita unas estructuras propias para poder traducirse. Estudios genéticos y bioquímicos realizados en Escherichia coli identificaron tres genes esenciales para la síntesis de dicho aminoácido:
- selA: codifica para el enzima selenocisteína sintasa.
- SelC: codifica para el tRNA específico de selenocisteína (tRNASec).
- SelD: codifica para el enzima selenofostao sintetasa.
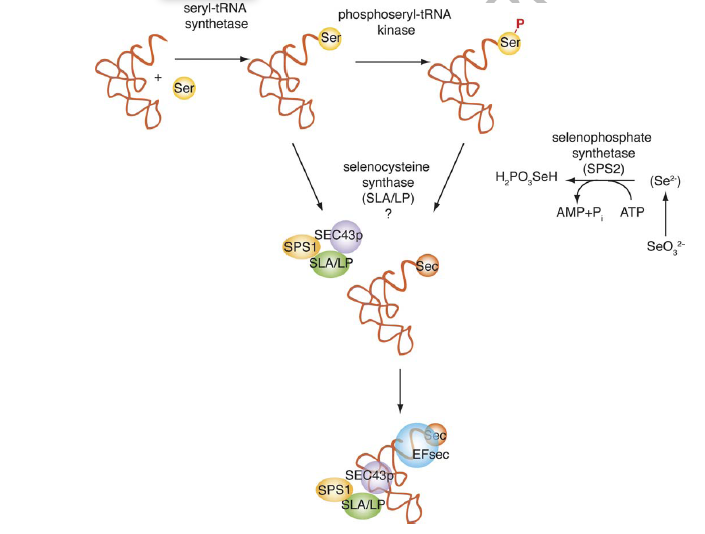
Los homólogos del gen selD en eucariotas son dos: selenofosfato sintetasa 1 (Sps1) y selenofosfato sintetasa 2 (Sps2). Sps2 es en sí misma una selenoproteína, lo que sugiere que estaría involucrada en la regulación de su propia síntesis (Guimaraes e al,.1996). Aunque la diferencia entre estas dos proteínas no está bien definida todavía, parece ser que Sps2 es considerablemente más activa que Sps1. Esta diferencia en la actividad podría deberse, en parte, al hecho de que sólo Sps2 contiene selenocisteína en su centro activo. La propuesta actual de la síntesis de Sec en eucariotas es la siguiente (Figura 2). El tRNASec es aminoacilado con una serina (ser-tRNASec) que servirá como un esqueleto de carbono para la Sec. Por otra parte,la enzima selenofosfato sintetasa es responsable de la síntesis del monoselenofosfato, el donador de Se, a partir del selenito. Para concluir, probablemente una actividad de selenocisteína sintasa es incorporar el donador de selenio al ser-tRNASec obteniendo el aminoácido Sec ya cargado sobe el tRNA: Sec-tRNASec.
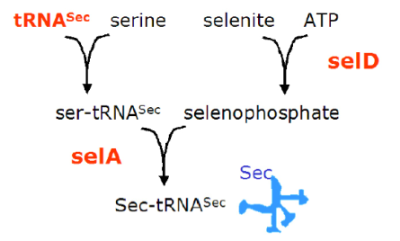
Figura 2
2.Incoporporación del aminoácido en la selenoproteína:
El aminoácido Sec es incorporado como resultado de la lectura del codón UGA. El hecho de que este codón tenga una función dual como codón stop y como codón para Sec implica que la célula ha de tener mecanismos para diferenciar entre las dos situaciones. Los elementos imprescindibles para el reconocimiento del codón UGA y para la incorporación de Sec a la cadena polipeptídica fueron identificados primeramente en procariotas, y son los siguientes:
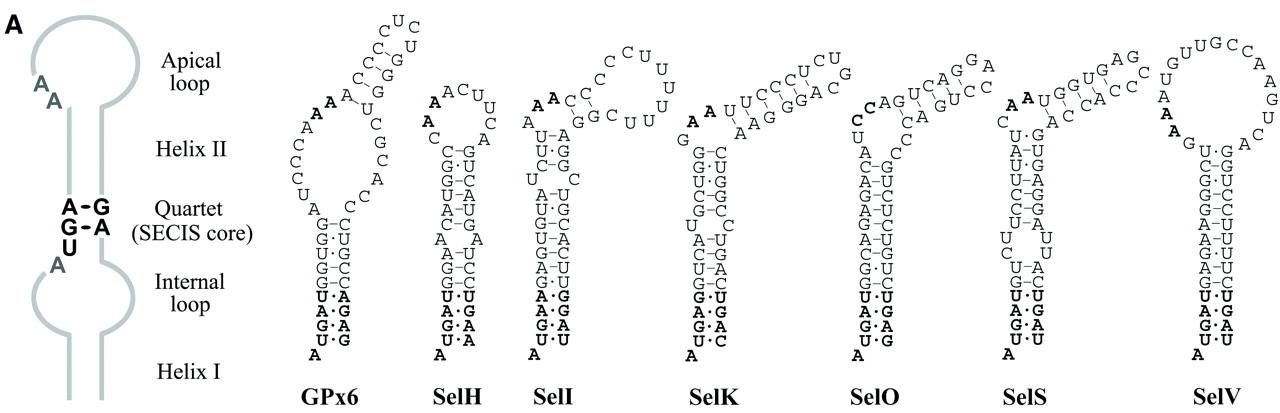
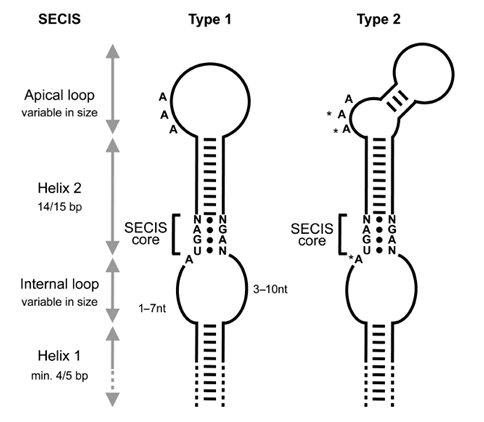
- El elemento SECIS (Sec insertion sequence) se encuentra en el extremo 3' UTR después del codón UGA.
- El gen selB codifica para el de elongación específico de Sec.
Debido al posicionamiento del SECIS en el mRNA, este necesita la proteína SBP2 (SECIS binding protein 2), que hace de nexo entre este y el ribosoma. El factor de elongación específico de Sec también interacciona con la proteína SBP2, al mismo tiempo que recluta Sec-tRNASec e incorpora el aminoácido a la cadena polipeptídica de la selenoproteína. Aunque se conoce la formación del complejo ribosoma-SBP2-EFsec-Sec tRNASec asociado al SECIS, todavía hay muchas incógnitas sobre cómo se constituye y de la mecánica de reconocimiento del codón UGA como un lugar de inserción de Sec. En última instancia se produce un plegamiento del mRNA para acercar el SECIS al lugar de inserción de Sec (figura 3). Se ha postulado que este plegamiento y el complejo proteico formado alrededor del SECIS evitan la entrada de los factores de terminación de la traducción, permitiendo la incorporación de Sec y la continuación de la transcripción.
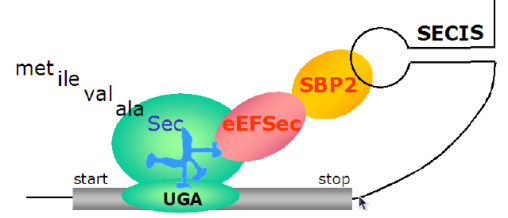
Figura 3
Identificación:
A parte de la presencia del codón UGA en pauta de lectura y el SECIS, las selenoproteínas no tienen ninguna característica específica de estructura 1aria, 2aria, 3aria o 4aria que permita la identificación de todos ellos. Es decir, la identificación de una determinada selenoproteína no sirve para identificar a otras.
Uno de los métodos que se han aplicado para identificar y caracterizar selenoproteínas se basa en las propiedades del Se y sus isótopos. La incorporación de 75Se en una proteína puede detectarse al correr un extracto proteico en un gel de policrilamida y exponerlo. Cuando se ha aislado una putativa selenoproteina y se sintetiza en presencia de 77Se, se puede realizar el análisis de la misma por técnicas más sofisticadas como diferentes tipos de espectroscopias y así obtener información estructural, mecánica y funcional sobre la selenoproteína aislada. Otra manera de identificar selenoproteínas sin la necesidad de conocer su secuencia es mediante el análisis computacional de ESTs para encontrar la presencia de elementos SECIS. Una vez se ha identificado el cDNA candidato, hay que corroborar por los métodos anteriormente descritos, que codifica para una selenoproteína.
Las técnicas mencionadas solo permiten la identificación de parte de las selenoproteínas: las expresadas a niveles detectables y las presentes en los bancos de ESTs. La secuenciación de genomas ha abierto las puertas al diseño de herramientas bioinformáticas para intentar identificar todas las selenoproteínas presentes en un determinado organismo.
Función:
El hecho de que exista una maquinaria tan compleja, y al mismo tiempo tan específica y exclusiva para la síntesis de selenoproteínas, hace pensar que su función no es trivial.
Las selenoproteínas procariotas están involucradas en procesos catabólicos y en la utilización del Se para catalizar diversas reacciones redox. Respecto a eucariotas, hasta el momento se han identificado mínimo 25 de selenoproteínas (en la figura 4 podemos ver 19 familias). De muchas todavía no se han realizado estudios que desvelen su función, pero las caracterizadas funcionalmente participan en reacciones anabólicas de oxido-reducción y funcionan como antioxidantes. Es decir, participan en el control del balance redox celular evitando los posibles efectos dañinos que una alteración del mismo puede causar. En bacterias, el papel de las selenoproteínas parece ser algo distinto (muchas selenoproteínas de bacterias participan en procesos catabólicos, por ejemplo). Por esta y otras razones, algunos autores rechazan la hipótesis de un origen único para todas las selenoproteínas.
Figura 4
La enzima glutatión peroxidasa (Gpx) fue la primera selenoproteína identificada como tal en eucariotas. Dos miembros de la familia Gpx protegen las células del daño causado por la peroxidación, reduciendo el peróxido de hidrógeno (H2O2) y los hidroperóxidos. Otro miembro de la familia, reduce los hidroperóxidos de fosfolípidos, colesterol y ésteres de colesterol, protegiendo así a las células de la peroxidación lipídica de las membranas. La tioredoxina reductasa (Trx) es una selenoproteína también importante en la detoxificación del H2O2. Tres miembros de esta familia controlan el estado redox celular reduciendo la tioredoxina y otros sustratos. Finalmente, una otra familia de proteínas oxidoreductasas, las deiodinasas, son selenoproteínas involucradas en el metabolismo de las hormonas tiroideas.
Hay que destacar que en las 19 familias de selenoproteínas identificadas, se encuentran ortólogos que tienen cisteína (Cys) en lugar de Sec. Este mismo fenómeno se da también dentro de una misma especie, por lo tanto, también se encuentran parálogos de selenoproteínas con Cys. Estructuralmente, el aminoácido Sec es idéntico al aminoácido Cys a excepción de que contiene Se en lugar de azufre. También, la Sec tiene una ventaja funcional porque su grupo selenol es más reactivo que el grupo tiol de la Cys. Estudios funcionales han mostrado que la actividad catalítica de las selenoproteínas es más grande que la que se obtiene en substituir la Sec por una Cys.