TblastN
En el TblastN s'obté un hit molt bo en que la selenocisteïna queda alineada amb un codó stop (*) de la seqüència del contig gi|126302146|gb|AAXJ01000359.1|

Clica la imatge per veure-la gran
Per veure el fitxer de l'alineament clica aquí.
Predicció del SPS2 en P. marinus
No es va poder utilitzar el programa Exonerate amb la seqüència obtinguda amb el programa Fastasubseq, de manera que es va utilitzar directament el Genewise.

Clica la imatge per veure-la gran
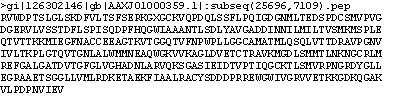
Clica la imatge per veure-la gran
Per veure el fitxer Genewise clica aquí
Alineament de la SPS2 predita
T-coffee La proteïna predita del Genewise és més curta que la seqüència extreta pel TblastN de manera que no arriba a alinear la selenocisteïna. Per això es va fer una proteïna quimèrica i es va alinear amb la proteïna humana mitjançant un T_coffee.

En l'alineament obtingut sí que es veu que la proteïna predita SPS2 conté selenocisteïna.
Blastcl3 nr Mitjançant el Blastcl3 es comprova que el procés realizat és correcte ja que es troben bons hits de la proteïna SPS2 de Perkinsus marinus amb SPS2 d'altres organismes.
Per veure el fitxer blastcl3 clica aquí
Cerca d'elements SECIS
En la cerca de SECIS s'ha obtingut un possible resultat amb un score de 12,01, el qual tenint en compte que Perkinsus marinus és un organisme allunyat filogenèticament dels altres organismes en que s'han buscat elements SECIS, és bo.
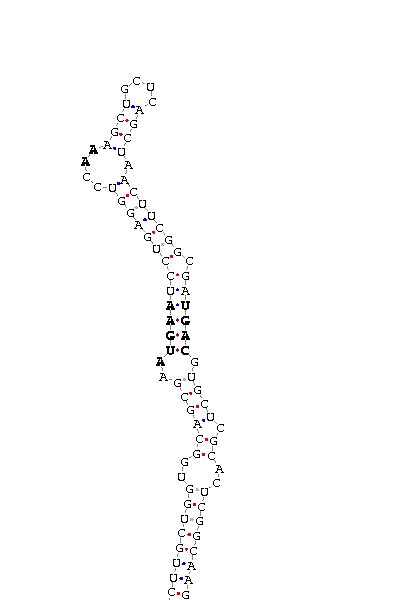
Clica la imatge per veure-la gran
Per veure la seqüència clica aquí
