Selenoproteïnes en Plasmodium vivax
Clara Conill, Estel Enreig, Albert Gil, Carlos González, Jordina Guillén
Facultat de Ciències de la Salut i de la Vida
Universitat Pompeu Fabra
SPS
La Selenophosphate Syntase (SPS) és una proteïna involucrada en la síntesi de selenoproteïnes la funció de la qual és encara desconeguda. En humans hi ha SPS1, homòloga amb T, i SPS2, selenoproteïna. Recentment, experiments han mostrat que la SPS1 està present en tots els organismes, fins i tot aquells que no tenen selenoproteïnes, cosa que fa pensar que SPS1 podria estar involucrada en altres funcions apart de la síntesi de selenoproteïnes.
RECERCA DE SPS EN Plasmodium vivax
Per començar la nostra recerca de SPS1 i SPS2 al genoma de Plasmodium vivax, vam descarregar-nos les seqüències de les proteïnes homòlogues en Homo sapiens de la base de dades de SelenoDB. Un cop obtingudes, vam fer un TBLASTN contra el nostre genoma des de la terminal. Tant en una proteïna com en l'altra vam obtenir un resultat no gaire significatiu. Però pel que havíem vist fins ara, el nostre organisme tenia selenoproteïnes, i per tant també havia de tenir la maquinària. Així, vam centrar la recerca de la SPS de Plasmodium vivax basant-nos en SPS1 d'Homo sapiens, ja que era la que ens donava un e-value més baix. A més, veiem com les dues SPS s'han alineat pràcticament a la mateixa regió del mateix contig (ctg_7052), pel que deduïm que el nostre organisme només tindrà una SPS.

|
| Figura 1. Resultats TBLASTN SPS1 |
| Descarrega't l'arxiu |

|
| Figura 2. Resultats TBLASTN SPS2 |
| Descarrega't l'arxiu |
Al TBLASTN de SPS1, els dos primers corresponen al mateix contig. Això ens va fer pensar que el gen ocupava una regió bastant gran, la qual cosa ens va fer tallar uns 4000 aminoàcids del contig per poder passar aquesta regió al GeneWise. Cal tenir en compte que el gen es trobava en el sentit invers, i per tan vam haver de córrer el programa FastaRevComp per obtenir la seqüència reverse. Ara ja podíem carregar el fragment de seqüència al GeneWise. Tot seguit s'adjunta l'output del GeneWise on es mostra la predicció del gen . Així, veiem com la predicció és d'un gen amb dos exons i dos introns.
Un cop feta la predicció, vam voler comprovar si hi havia SECIS o no, tot i que SPS1 és un proteïna de maquinària que en cap espècie és selenoproteïna. Efectivament, passant el SECISearch a la seqüència tallada (suficientment llarga com per abarcar el SECIS) no n'hi vam trobar cap.
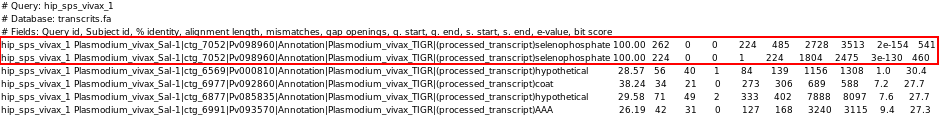
Arribats en aquest punt vam estudiar amb més deteniment la proteïna predita pel GeneWise. Primer de tot el que veiem és que no comença amb metionina, cosa que ens fa pensar que deu estar abans. Si mirem els aminoàcids previs, observem que n'hi ha un bastant més enrere, pel que pensem que deu ser aquest el codó d'iniciació. La raó perquè estigui més enrere podria ser degut a la diferència evolutiva entre Homo sapiens i Plamodium vivax. Un problema semblant ens trobem amb el codó de finalització; 64 aminoàcids més endavant trobem el primer codó stop, el que ens fa pensar que la proteïna deu acabar aquí. Tot seguit el que fem és descarregar-nos de PlasmoDB l'arxiu de tots els transcrits de Plasmodium vivax, i procedim a fer un TBLASTN des de la terminal amb la nostre proteïna predita contra la base de dades de tots els transcrits.
|
| Figura 3. Resultats TBLASTN transcrits |
| Descarrega't l'arxiu |
Així, veiem que tenim un hit molt bo que correspon al contig que ja havíem deduït, concretament al transcrit amb referència Pv098960. El primer que comprovem és que el transcrit acaba en aquell codó stop que havíem predit, però el que veiem és que el transcrit comença molt abans de la metionina que nosaltres havíem predit, així que creiem que la predicció inicial no estava del tot bé. A partir del GeneWise ens havia predit una proteïna de llargada semblant a la humana, però com ja hem dit anteriorment, la diferència evolutiva és molt gran i, per tant, creiem que el trasncrit corresponent a la SPS1 és tan llarg degut a aquesta diferència.
La nostra sorpresa més gran és que fent un alineament amb T-COFFEE entre el transcrit i el cDNA de la proteïna predita per nosaltres (amb la metionina inicial igual que la del transcrit) veiem que l'intró que havíem predit realment és codificant (veure l'alineament). Una possible hipòtesi que donem és que aquella regió no estigui tan conservada ja que no és de vital importància per la funcionalitat del gen (d'aquí que el GeneWise l'hagi predit com un intró), però que per altra banda sí que ho és pel correcte plegament, i per tant, per la proteïna en sí. Així doncs, gràcies a aquest últim BLAST, deduïm que la seqüència final consta d'un únic exó de 3516 bp i una proteïna de 1171 aminoàcids. Tot seguit es mostra la informació del gen en format .gff.
# seqname source feature start end score strand frame attributes # Plasmodium_vivax_SaI-1|ctg_7052|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome gene 400886 404398 6145 -
. gene_id 1 ; sequence SPS ; gene_orientation . Plasmodium_vivax_SaI-1|ctg_7052|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome cds 400886 404398 . -
. Plasmodium_vivax_SaI-1|ctg_7052|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome exon 400886 404398 . -
. insertions 0 ; deletions 0 Plasmodium_vivax_SaI-1|ctg_7052|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome similarity 400886 404398 6145 -
. alignment_id 1 ; Query SPS ; Align 404399 1 3513
| Figura 4.Exonerate en format .gff |
Per corroborar la nostra hipòtesi procedim a estudiar els dominis que té la nostra proteïna amb InterProScan. Així, si comparem els dominis que té la SPS1 de Homo sapiens amb els dominis de SPS de Plasmodium vivax veiem que, tot i les grans diferències, les dues contenen el domini PTHR10256, que correspon a Selenofosfat sintasa. Tot i això, en observar tantes diferències vam descarregar la seqüència de la hipotètica SPS en Plasmodium falciparum de PlasmoDB i vam comparar els dominis, veient com entre P.falciparum i P.vivax els dominis són pràcticament idèntics.
 |
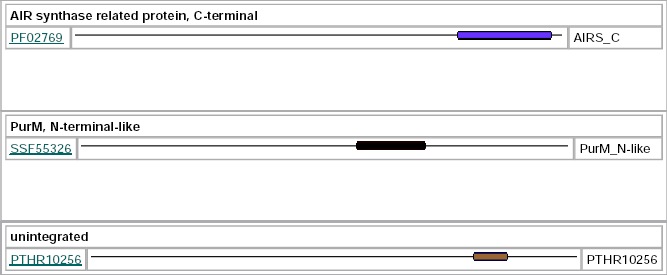  |
| Figura 5. A dalt, dominis de Homo sapiens. A baix a l'esquerre, dominis de Plasmodium vivax. A baix a la dreta, dominis de Plasmodium falciparum |
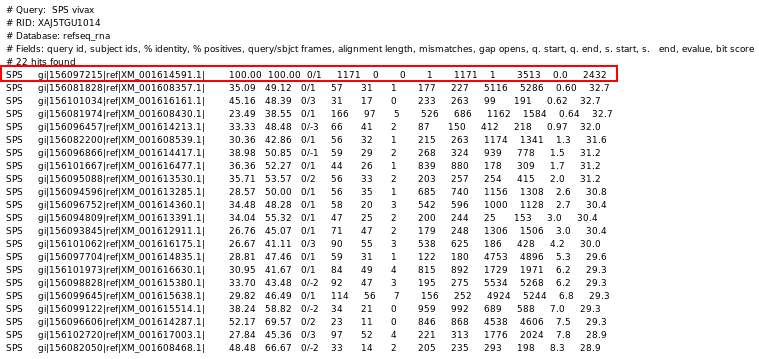
Finalment procedim a fer un TBLASTN de la nostra hipotètica proteïna contra una base de dades de mRNA de Plasmodium vivax des de NCBI per veure si realment la nostra proteïna és expressada en aquest organisme, i els resultats són òptims; obtenim un e-value de 0,0.
|
| Figura 6. Resultats TBLASTN mRNA |
| Descarrega't l'arxiu |
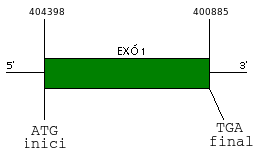
Un cop fetes les corroboracions esmentades, la nostra predicció de SPS per Plasmodium vivax és la segŁent:
|
| Figura 7. Esquema de SPS de Plasmodium vivax |