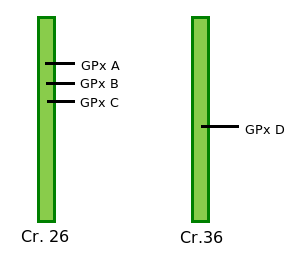
En primer lloc, vam utilitzar les 8 glutation peroxidasa d'Homo sapiens per fer un TBLASTN contra tot el genoma de Leishmania major. Vam obtenir hits a dos cromosomes diferents per totes les glutation peroxidasa, concretament a 3 regions del cromosoma 26 i en una regió del cromosoma 36. Hem reflectit aquests resultats en la taula següent:
| 1226783 | ||||
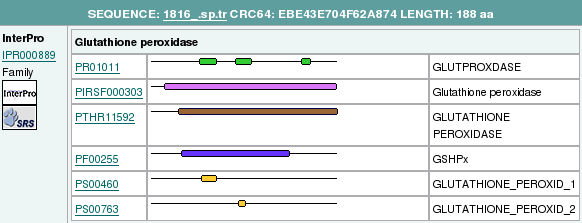
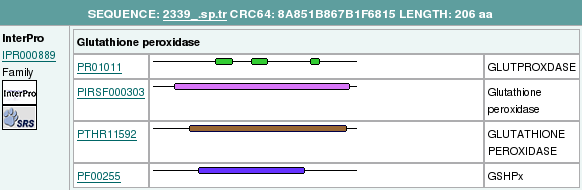
D'aquesta taula vam poder extreure que, al genoma de Leishmania hi ha 4 regions que codifiquen per glutatió peroxidasa, 3 al cromosoma 26 i 1 al 36. Per poder predir l'estructura de cada gen i predir també la proteïna de cada regió, ens vam fixar en quina GPx presentava un e-value més baix per cada una de les 4 regions diferents. És a dir:
- Per la regió ~234000-234700, utilitzarem GPx4 (e-value de 8x10-29).
- Per la regió ~236500-237000, utilitzarem GPx4 (e-value de 8x10-29).
- Per la regió ~238800-239400, utilitzarem GPx4 (e-value de 1x10-29).
- Per la regió ~1226700-1227200, utilitzarem GPx8 (e-value de 3x10-22).
La distribució, en general, seria: