






 |
 |
 |
 |
 |
 |

Malgrat
existeix un gran nombre de
molècules diferents, la gran majoria es poden agrupar en un
conjunt limitat de famílies, basant-se en la similaritat de les
seves seqüències. En el cas de les famílies
de proteïnes, s'observen algunes regions que s'han conservat
més que altres al llarg de l'evolució. Aquestes solen ser
importants per la funció de la proteïna o pel manteniment
de la seva estructura tridimensional. D'altra banda, elements
reguladors conservats del DNA desenvolupen funcions indispensables per
la vida com per exemple el reconeixement de factors de
transcripció o senyals d'splicing.
Analitzant les propietats
variables i constants de les seqüències (tant
d'aminoàcids com de nucleòtids), podem derivar una marca
que distingueixi als seus membres de la resta.
Què és un motiu conservat?
Mitjançant un alineament
múltiple de seqüències que pertanyen a la mateixa
família és possible trobar un motiu consens. El
motiu sol ser una seqüència curta expressada de manera que
quedin representades totes les possibilitats de seqüències
observades d'aquest motiu. Per exemple, el motiu
[ASV]-S-C-[NT]-T-x(2)-[LIM] representa 7200 seqüències
diferents d'aminoàcids. Si una d'aquestes és una
subseqüència d'una proteïna no caracteritzada, la nova
proteïna farà match
amb el motiu i deduirem que la
subseqüència té la funció prèviament
descrita (en aquest cas probablement seria un "lloc actiu
gliceraldehid 3-fosfat dehidrogenasa"). És a dir, la
identificació de motius conservats ens permet assignar una
funció a una proteïna o regió del DNA abans
desconeguda.
Bases de dades
Gràcies a la
investigació en aquest camp, s'han pogut
determinar un gran nombre de motius conservats que es poden consultar
en múltiples bases de dades com PROSITE i TRANSFAC.
PROSITE
recull al voltant de 1400 motius
proteics i permet escanejar una seqüència per tal de trobar
algun motiu conegut.
TRANSFAC
és una base de dades de factors de
transcipció eucariotes i els seus llocs d'unió al genoma.
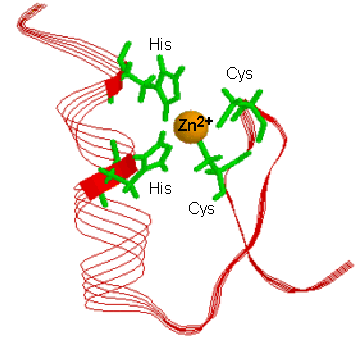
Representació dels motius: domini Zinc-finger
Per tal d'entendre millor en
què es basa la identifació
de motius conservats, exemplificarem el cas
del domini Zinc-finger. Aquest domini és una estructura
proteica d'unió als àcids nucleics inicialment
identificada al factor de transcripció TFIIIA de Xenopus i
posteriorment, trobat en múltiples proteïnes d'unió
a DNA o RNA. Es composa de 25-30 residus aminoacídics amb dos
residus de cisteïna o histidina en ambdós extrems del
domini
involucrats en la unió tetrahèdrica d'un àtom de
zinc. Es postula que interacciona amb aproximadament cinc
nucleòtids. A partir de l'alineament múltiple de
seqüències amb aquest motiu s'extrau la següent
expressió
regular: C-x(2,4)-C-x(3)-[LIVMFYWC]-x(8)-H-x(3-5)-H que s'interpretaria
com dues C separades per 2, 3 o 4 aminoàcids
qualssevol, seguides de L, I, V, M, F, Y, W o C, i separades de dues
H per 8 aminoàcids. Si tenim una proteïna amb funció
desconeguda que sospitem s'uneix al DNA, intentarem trobar
coincidències d'aquest motiu o altres per tal d'esbrinar-la.
 |
 |
|
|
Representació de l'expressió regular |
Estructura d'una regió amb domini Zinc-finger |
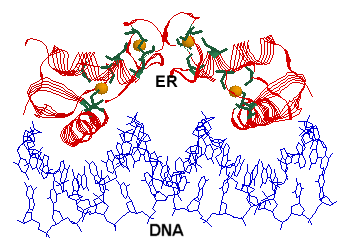
ER (receptor d'estrògens) unint-se al DNA mitjançant
un dímer amb un total de 4 motius Zinc-finger (boles taronja)
|
