La predicció dels possibles gens presents en la regió ENm003, es va fer usant 3 programes de predicció de gens diferents per, d'aquesta manera, optimitzar els resultats.
Els programes usats van ser els seguents: GENEID, GENSCAN, i FGENESH.
Cal que la predicció obtinguda estigui en format Gff, aquest format de vegades ja és proporcionat pel propi programa, tal és el cas de GENEID. Ara bé, si aixó no és així caldrà transformar-lo.
Procedirem doncs, a evaluar de manera separada l'ús del diferents programes.
PREDICCIÓ AMB GENEID
A partir de la seqüència emmascarada en format fasta (Apo.seq.masked) es fa còrrer el programa dues vegades de tal manera que s'obtinguin dos formats d'outputs ( en escollir GFF i en escollir Geneid).
Els resultats obtinguts van ser els seguents.
Donat que el format proporcionat ja era l'adequat no es va procedir a cap modificació posterior.
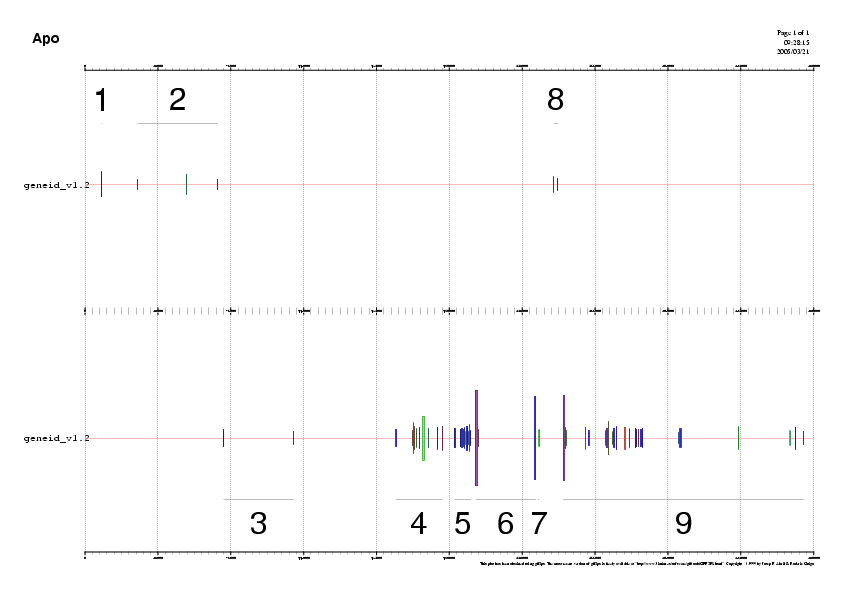
Per tal de poder visualitzar la predicció dels gens feta per geneid es va fer el següent :

L'inconvenient d'aquest programa va ser que no va permetre córrer la seqüència sencera de 500000 bp. Aquest fet va obligar a tallar dita seqüència en 5 fragments diferents: un de 100000 bp, i els altres 4 de 110000 bp, per tal de tenir un solapament de 10000 nucleòtids entre fragments que permet´s evitar possibles pèrdues d'exons.
Per tal de realitzar dita fragmentació primer calia definir un export en el shell tal com :
La comanda que va permetre dur a terme la fragmentació va ser la següent:
On el primer nombre fa referència al nucleòtid des del qual s'inicia la fragmentació, i el segon indica la longitut del fragment.
L'inici i final de cada fragment obtingut es mostra en la següenxt taula:
| 1r fragment | 0 - 100000 |
| 2n fragment | 90000 - 200000 |
| 3r fragment | 190000 - 300000 |
| 4rt fragment | 290000 - 400000 |
| 5e fragment | 390000 - 500000 |
Per tal d'obtenir la seqüència en format fasta, posteriorment a la fragmentació, es van obrir els diferents fitxers amb emacs i s'els hi va afegir "> Apo_ + tabulacio", obtenint així el format ja adequat per a procedir a la predicció de gens amb genscan.
La predicció amb GENSCAN va donar el resultat exonic per a tots 5 fragments, a més a més de proporcionar també la seqüència nucleotídica i aminoacídica de cada gen predit. En aquesta ocasió el format no es trobava en gff, així doncs calia transformar-lo, i això es va fer com es mostra a continuació.
$2 ~ /Term|Intr|Init/ {
print "Apo_1", "genscan", $2, start=($4<$5 ? $4 : $5), end=($5<$4 ? $4 : $5), $13, $3, $7,"Apo_1"$1"";
}' GENSCAN.Apo.mask1| \
sed 's/\.[0-9][0-9]$//' > GENSCAN.Apo.mask1.gff
En aquesta comanda s'inclou la informació necessària per afegir a la penúltima columna del fitxer gff el nom de la seqüència en la que es troba aquell gen determinat, ja que d'aquesta manera es podrà determinar a quina seqüència correspon cada gen i serà més fàcil enumerar els diferents gens.
Posteriorment a tenir ja la predicció en format gff cal modificar el valor del inici i final d'exó per tenir-los en valor absolut.
Per tal de modificar això es fa servir la comanda següent per a tots els fragments excepte pel primer (APO1.GENSCAN.GFF), ja que aquest ja es trobava amb les coordenades adequades.
gawk 'BEGIN{OFS="\t"}{$4=$4+90000; $5=$5+90000;print}' GENSCAN.Apo.mask2.gff > APO2.GENSCAN.GFFmodif
gawk 'BEGIN{OFS="\t"}{$4=$4+190000; $5=$5+190000;print}' GENSCAN.Apo.mask3.gff > APO3.GENSCAN.GFFmodif
gawk 'BEGIN{OFS="\t"}{$4=$4+290000; $5=$5+290000;print}' GENSCAN.Apo.mask4.gff > APO4.GENSCAN.GFFmodif
gawk 'BEGIN{OFS="\t"}{$4=$4+390000; $5=$5+390000;print}' GENSCAN.Apo.mask5.gff > APO5.GENSCAN.GFFmodif
El fet de tenir els diferents fragments en valors absoluts, permet eliminar aquells exons que es troben solapats entre fragments consecutius. Primer però, cal concatenar tots els fitxers mitjançant la següent comanda:
Posteriorment s'eliminen els exons coincidents o solapants seguint un criteri determinat que es basa en mantenir aquells exons amb major score.
Al observar els fitxers concatenats en el shell es pot concloure:
Aquest programa és capaç d'analitzar la seqüència completa, el resultat proporcionat és FGENESH,Apo.txt.
Com es pot apreciar, cal passar els resultats format gff per tal de poder-lo comparar amb les altres prediccions.
Per a fer això es fa servir una comanda similar a la usada en el cas de genscan però amb alguna modififcació ja que la frame no es proporcionada per als outputs. Cal calcular-la a partir dels inicis i finals d'exons i tenint en compte si la seqüència és formward o reverse.
Per tal de realitzar això, es fa servir un programa anomenat fgenesh2gff.awk , el qual s'executa de la manera següent:
Per tal de poder visualitzar la predicció dels gens cal fer els passos següents:
Donat que es van calcular els frames per tal de completar el gff, es va comprobar que aquests coincidissin visualitzant la predicció dels exons d'una manera més acurada, fent 4 blocs de la següent manera:
Les imatges corresponents a cada un d'aquests blocs, per comprobar que es mantinguin els marcs de lectura, es poden veure tot seguit.
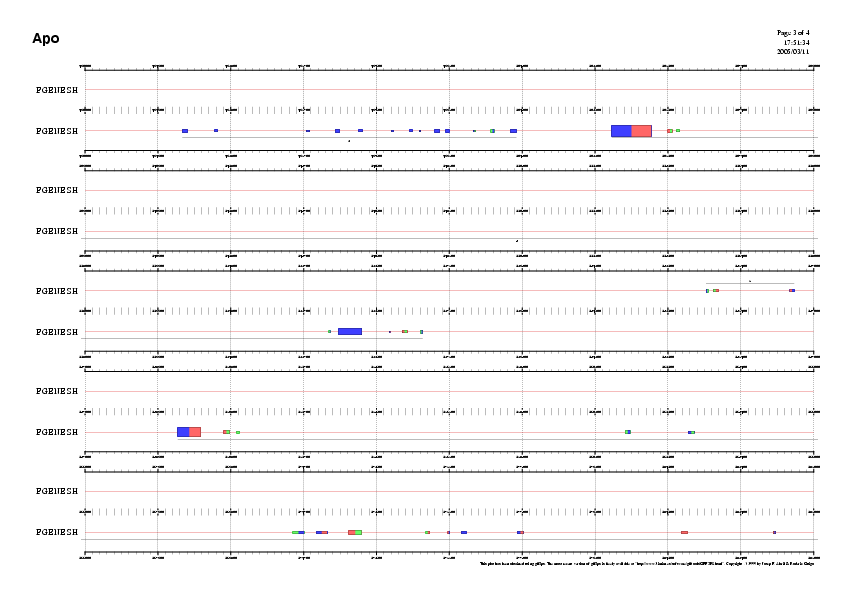
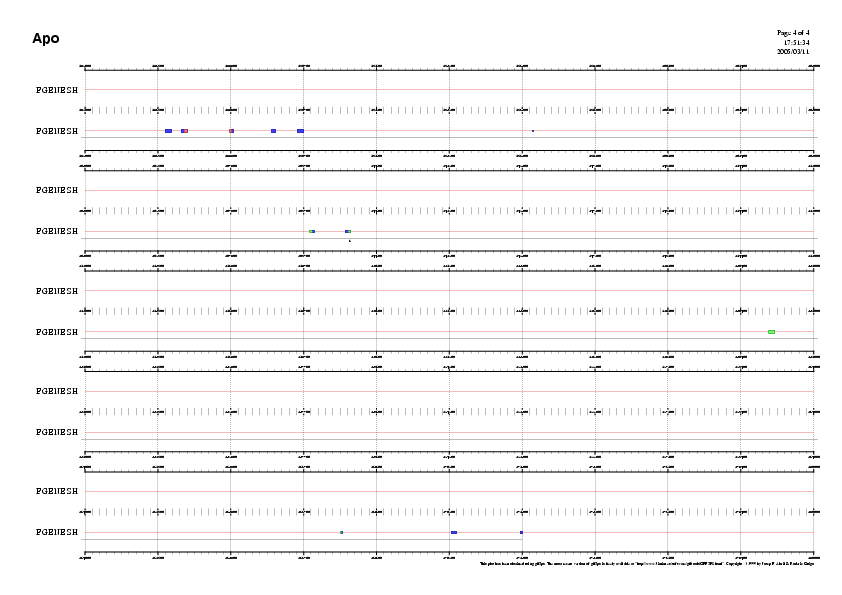
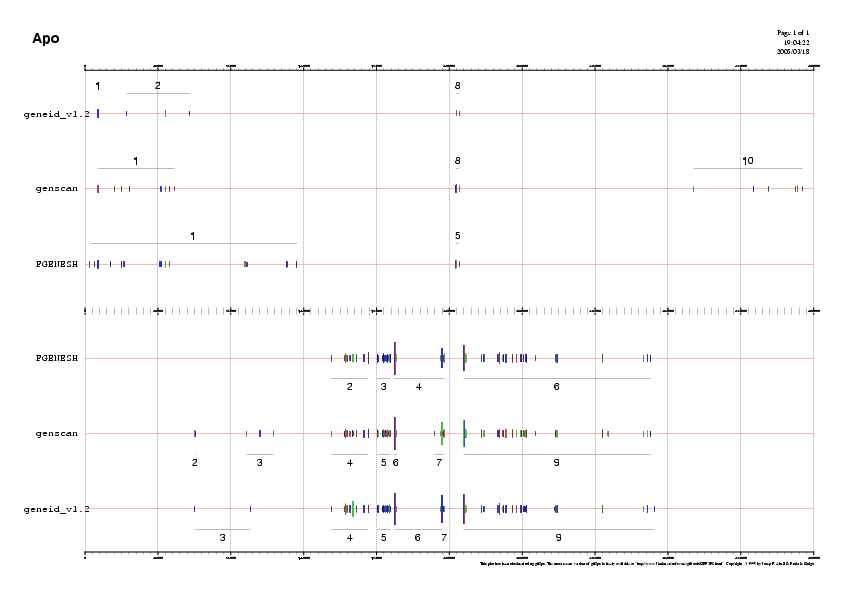
Després d'haver fet les prediccions dels gens de la regió per a tres programes diferents es pot veure en la taula que es mostra tot seguit, com cada un dóna uns resultats mínimament diferents.
| Gen | FGENESH | GENEID | GENSCAN |
|---|---|---|---|
| 1 | 3130 - 139570 (+) | 8455 - 8784 (+) | 8455 - 58950 (+) |
| 2 | 162093 - 186575(-) | 27356 - 69127 (+) | 72302 - 72894 (-) |
| 3 | 192536 - 201251 (-) | 72302 - 108786 (-) | 106398 - 124146 (-) |
| 4 | 203739 - 236802 (-) | 162093 - 186575 (-) | 162093 - 186575 (-) |
| 5 | 244194 - 246495 (+) | 192536 - 201251 (-) | 192536 - 201251 (-) |
| 6 | 249419 - 372401 (-) | 203739 - 235179 (-) | 203739 - 205462 (-) |
| 7 | - | 236266 - 236802 (-) | 229918 - 236802 (-) |
| 8 | - | 244422 - 246495 (+) | 244194 - 246495 (+) |
| 9 | - | 249419 - 374941 (-) | 249419 - 372401 (-) |
| 10 | - | - | 401014 - 472564 (+) |
Es pot veure que mentre que Fgenesh prediu només 6 gens, Geneid en prediu 9 i Genscan 10.
Tot i les petites diferències pel que fa a totalitat de gens, cal a dir que els gens predits, després de comparar tots tres resultats, són força similars en tots els casos.
Es pot comprobar perfectament després d'observar la taula, com hi han coincidències pel que fa a alguns gens predits pels diferents programes.
Per tal de poder corroborar aquesta predicció, seria convenient observar la predicció feta per tots tres programes en un mateix gràfic, aconseguit de la manera que prosegueix: