BÚSQUEDA Y TRADUCCIÓN DE ORFs EN UNA SECUENCIA GENÓMICA DE HHV-8

BÚSQUEDA Y TRADUCCIÓN DE ORFs EN UNA SECUENCIA GENÓMICA DE HHV-8 |

|
El témino Herpes viene del griego y significa latente, crónico. Alude al hecho que el virus herpes se mantiene latente después de una infección primaria, pudiendo reactivarse pasado un tiempo.
Son virus bastante grandes y complejos, con un genoma de unas 130-230 kbp que codifican para una gran variedad de enzimas involucradas en el metabolismo de los ácidos nucleicos, la síntesis de ADN y el procesamiento de proteínas. Las dianas de infección pueden variar enormemente, mientras unos infectan neuronas, otros pueden infectar linfocitos u otros tipos celulares. Aunque los diferentes tipos de Herpesvirus difieren en su secuencia genómica, tienen características similares en lo que se refiere a la estructura del virión y a su organización genómica:
 imagen 1.1
imagen 1.1
 imagen 1.2
imagen 1.2
|
|
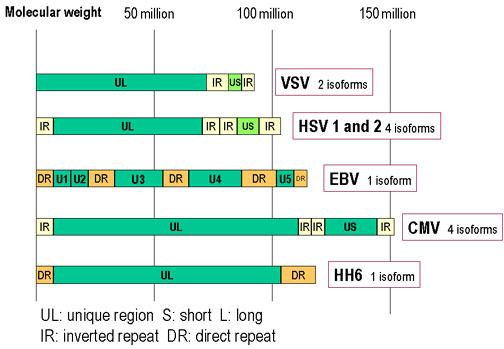 imagen 1.3
imagen 1.3
El genoma de herpesvirus tiene una región larga única (UL) y una región corta única (US) flanqueadas por repeticiones invertidas que permiten reordenamientos de estas regiones únicas. Las secuencias repepetitivas hacen variar el tamaño genómico del virus. (imagen 1.3)
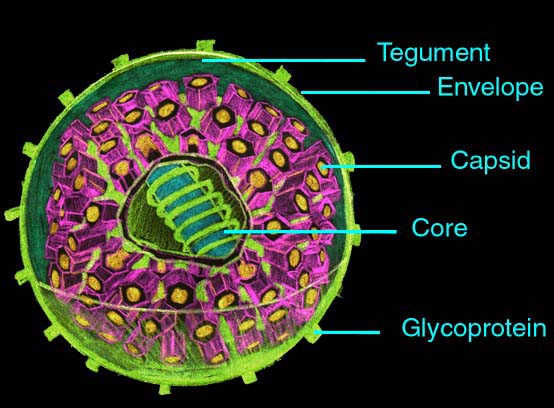
La envuelta de los herpesvirus contiene al menos 9 glicoproteínas, la mayoría de ellas necesarias para la infectividad. La interacción de estas glicoproteínas con receptores celulares da lugar a la fusión de la envuelta con la membrana celular. Tras la fusión pierde la cápside y el core entra al núcleo vía poros nucleares, donde el genoma es circularizado. La transcripción del genoma viral está regulado por ARNm producidos por la ARN-polimerasa II de la célula huésped.
Hay 8 tipo de herpesvirus humanos conocidos. Esta familia se dividide en tres subfamilias:
Dentro de la subfamilia de los Gammaherpesvirinae encontramos el herpesvirus humano 8 (HHV-8) o herpesvirus asociado al Sarcoma de Kaposi.
|
Al HHV-8, descubierto por primera vez en lesiones de Sarcoma de Kaposi, se le atribuyen propiedades tumorales. Se ha determinado su localización en células endoteliales vasculares y células fusiformes perivasculares. |
La secuencia completa de HHV-8 de aproximadamente 165 Kpb, tiene unos 80 Open Reading Frames (ORFs) organizados dentro de la región larga única (UL) y flanqueados por múltiples secuencias repetitivas con un alto contenido en C+G. La UL contiene bloques de genes conservados en la mayoría de herpesvirus, pero también encontramos genes no homólogos específicos de HHV-8 y virus relacionados.
Muchos de estos ORFs codifican para proteínas que ayudan al virus a evadir la respuesta inmunitaria de la célula huésped, previenen que su ciclo celular no se pare e interfieren en la activación de las vías apoptóticas. Todo esto les confiere características tumorales.
Hacer un programa en lenguaje Perl que permita identificar Open Reading Frames (ORFs) en secuencias de ADN. El ORF empezará siempre por metionina, acabará en un codón stop y tendrá una longitud mínima de 60 aminoácidos. El programa además tendrá que traducir la secuencia en las 6 pautas de lectura (3 forward y 3 reverse)
Analizaremos con el programa desarrollado una secuencia de ADN de HHV-8, en concreto un fragmento asociado al sarcoma de Kaposi (entrada U75698 , posiciones 73021-85260 extraída de GenBank).
Para cada ORF identificado haremos una búsqueda en bases de datos para ver si es un ORF ya identificado, cuál es la función de la proteína a la que da lugar y si hay homólogos en otros organismos.
El programa SARCORFS está escrito en lenguaje PERL y es ejecutable desde Linux. Sigue los siguientes pasos:
Tras el análisis de nuestra secuencia con el programa SARCORFS, hemos localizado 34 posibles ORFs que codifican para proteínas cuya longitud es igual o mayor a 60 aminoácidos, empiezan por metionina y terminan en un codón stop.
El análisis mediante diversas bases de datos y programas nos permitirá identificar cuál de estos ORFs potenciales, realmente lo son:
Gracias a la ejecución del programa SARCORFS con una región genómica del Herpesvirus Humano 8 asociado al Sarcoma de Kaposi, localizamos 34 posibles ORFs. Mediante BLASTP se vio que tan sólo 10 de ellos estaban ya identificados en las bases de datos y además aparecían hits con proteínas de otros organismos.
A continuación se ejecutó el CLUSTALW con cada una de las 10 proteínas predichas. Para cada uno de los alineamientos múltiples se utilizó la proteína predicha y los hits encontrados mediante BLASTP que tenían una mayor identidad a ésta.
Además se consultó en diferentes bases de datos (SWISSPROT, INTERPRO y NCBI) para conocer más sobre la función de las 10 proteínas ya identificadas.
Por último, se observó qué alineamientos del CLUSTALW entre dos proteínas tenían un score elevado y compartían la misma función. De este modo, se podría concluir que las dos proteínas tienen una probabilidad elevada de ser homólogas.
Con toda esta información podemos concluir, de cada ORF predicho ya identificado, lo siguiente:
Se trata del ORF 57 del genoma de Herpesvirus Humano 8. Es una proteína inmediata temprana (immediate early protein), muy conservada a lo largo de toda la familia de herpesvirus, cuya función es la regulación transcripcional. Tiene 442 aminoácidos y hace splicing mediante el factor SC-35, dando lugar a una proteína de 275 aminoácidos (proteína que nosotros hemos predicho con nuestro programa).
Al hacer el alineamiento múltiple de nuestro ORF predicho con los hits de mayor identidad obtenidos mediante el BLASTP, la secuencia con un mayor score (48) ha sido la proteína inmediata temprana del radinovirus 26-95 de Macaca Mulatta. Hemos comprobado que su función es la misma que la que tiene en el HHV-8 (regulación transcripcional) y que además está codificada por un gen que se llama igual (ORF 57). La traducción a proteína puede iniciarse en dos lugares diferentes del transtrito. De todo esto se puede deducir que se trata de proteínas homólogas.
El resto de hits localizados mediante BLASTP, aunque con menos score, la mayoría de ellos comparten la función de regulador transcripcional.
Forward, Pauta:1, Inicio-Final del ORF:41-1606 (Proteína 6)Se trata del ORF 50. Pertenece a la familia de los factores de transcripción tempranos o intermedio-tempranos e igual que la proteína anterior también suele realizar splicing. Es la proteína más importante en la reactivación de la fase lítica y funciona como un potente activador transcripcional de genes tempranos y tardíos del Sarcoma de Kaposi (como por ejemplo del ORF 57 y K8).
Al realizar el alineamiento múltiple, se obtuvo un score de 49 con la secuencia de Herpesvirus 17 de Cercopithecine . Es el factor transcripcional inmediato temprano Rrta, de 577 aminoácidos codificado por el ORF 50 del genoma de herpesvirus 17. El alineamiento de ambas secuencias se da entre una parte intermedia de la secuencia del ORF 50 de Herpesvirus 17 y la primera parte de la secuencia del ORF 50 de HHV-8, y por tanto este trozo de secuencia es el que se ha conservado. Con lo cual podemos decir que se trata de proteínas potencialmente homólogas.
Forward, Pauta:1, Inicio-Final del ORF:6416-8944 (Proteína 8)Se trata del ORF 56. Es una proteína de 843 aminoácidos con actividad replicativa, componente del complejo ADN helicasa-primasa.
Al realizar el alineamiento múltiple, hay dos proteínas (que también tienen como función la replicación del ADN) que poseen un score de 52, en el caso de la de rhadinovirus de Macaca Mulatta, y un score de 46, en el caso de herpesvirus 4 bovino. La longitud de dichas proteínas es de 828 y 843 aminoácidos respectivamente. Éstas también son codificadas por el ORF 56 de dichos virus. Hemos hecho un nuevo alineamiento múltiple de las tres secuencias para que se pueda ver la gran homología que hay entre ellas.
El resto de hits localizados mediante BLASTP, aunque con menos score, comparten la función replicativa.
Forward, Pauta:2, Inicio-Final del ORF:1830-2546 (Proteína 11)Es el ORF K8. Codifica para una cremallera de leucinas de 239 aminoácidos que homodimeriza y se une a regiones ricas en purinas. K8 interactua y colocaliza con el factor humano remodelador de la cromatina SNF5. Se piensa que funciona como activador transcripcional bajo condiciones específicas y su transactivación requiere su interacción con SNF5.
No ha sido posible realizar el alineamiento múltiple porque el BLASTP no encontró hits significativos que no fueran la propia proteína. Podríamos concluir por tanto que se trata de uno de los ORFs es específicos de HHV-8.
Forward, Pauta:2, Inicio-Final del ORF:2895-3485 (Proteína 12)Se trata del ORF K8.1 que codifica para una glicoproteína de la envuelta del virus. Sufre splicing en dos lugares diferentes dando lugar a otras dos proteínas: K8.1A (longer alternatively spliced glycoprotein) y K8.1B (shorter alternatively spliced glycoprotein). Es por esta razón que en el BLASTP nos identifica como hits estas dos proteínas a parte de la propia K8.1.
No ha sido posible realizar el alineamiento múltiple porque los hits localizados son la propia proteína y las proteínas resultantes de su splicing. Como en el caso anterior podemos decir que se trata de un ORF especíco de HHV-8.
Forward, Pauta:2, Inicio-Final del ORF:4647-5600 (Proteína 14)Es el ORF 54 de 318 aminoácidos. Codifica para la enzima dUTP pirofosfatasa, específica de dUTP y es crítica para la fidelidad de la replicación del ADN y su reparación. La dUTPasa hidroliza el dUTP a dUMP y pirofosfato, reduciendo así los niveles de dUTP, haciendo que el uracilo no pueda ser incorporado en el ADN, y proporcionado el dUMP para la síntesis de dTTP.
Al realizar el alineamiento múltiple se ha visto que hay dos secuencias que obtuvieron el mayor score con el ORF 54 de HHV-8: la dUTPasa de rhadinovirus de Macaca mulatta, con un score de 40, y la dUTPasa de herpesvirus 2 equino, con un score de 38. Tienen respectivamente 290 y 289 aminoácidos. El gen que las codifica es el ORF 54, al igual que en el HHV-8. Se alinean completamente con nuestra proteína, excepto en la primera parte de ésta, por lo que podemos decir que se trata de proteínas homólogas.
El resto de hits localizados comparten la misma función, de manera que también se trata de proteínas potencialmente homológas en otros organismos.
Reverse, Pauta:0, Inicio-Final del ORF:52-1398 (Proteína 16)Es el ORF K9, codifica para un factor regulador del interferon (vIRF), homólogo del celular. Gracias a esta homología, actúa como oncogen, inhibiendo la vía de señalización del interferon en la célula huésped.
Mediante el alineamiento múltiple hay una pequeña región en cuatro secuencias que obtienen el mayor score: en el vIRN-10 de Gallus gallus de 416 aminoácidos, en el vIRF de Rattus novergicus de 413 aminoácidos, en el vIRF linfoespecífico de Mus musculus de 449 aminoácidos y en el de vIRF-4 de Homo sapiens de 449 aminoácidos. Todos obtuvieron un score de 40-43. Todas ellas tienen la misma función reguladora del interferón y son homólogas con nuestra secuencia en una pequeña región conservada (ver resultados del BLASTP). Hemos realizado un nuevo alineamiento múltiple con sólo estas secuencias para ver claramente la región que se alinea entre todas ellas.
El resto de hits localizados tienen en su mayoría la misma función regulador del interferón.
Reverse, Pauta:1, Inicio-Final del ORF:5813-6493 (Proteína 26)Se trata del ORF 55 de HHV-8. Pertenece a la familia de proteínas BSRF1-like. Su función es desconocida.
En el alineamiento múltiple hemos localizado una secuencia de rhadinovirus de Macaca mulatta que tiene un score de 60, cuya función también es desconocida. Está codificada también por el ORF 55.
En general el resto de hits utilizados para realizar el alineamiento múltiple tienen scores bastante elevados (se alinean muy bien), todo y que la función de estas proteínas es también desconocida.
Reverse, Pauta:2, Inicio-Final del ORF:7596-7925 (Proteína 30)Se trata del ORF 53, de la familia de las proteínas BSRF1-like, que como la anterior tiene una función desconocida.
Al realizar el BLASTP sólo encontramos una proteína, también de función desconocida, que se alineaba con la segunda parte de nuestra proteína, posiblemente un dominio conservado. El score obtenido era de 67. Esta proteína, tiene una longitud de 104 aminoácidos y es codificada por el ORF 53 de rhadinovirus de Macaca mulatta.
Reverse, Pauta:2, Inicio-Final del ORF:8064-8456 (Proteína 31)Se trata del ORF 52 del genoma del HHV-8. Como en los dos casos anteriores, codifica para una proteína de la familia de BSRF1-like, cuya función es desconocida.
En el alineamiento múltiple todas las secuencias se alinean muy bien. Las dos que tuvieron un score más elevado fueron: la de rhadinovirus de Macaca mulatta con una longitud de 139 aminoácidos y la de herpesvirus-4 bovino con una longitud de 128 aminoácidos. Tuvieron un score de 47 y 38 respectivamente. Ambas son codificadas por el ORF 52 del genoma de sus respectivos virus, e igual que la anterior de función desconocida.