En aquest treball analitzem la seqüència AC090644 utilitzant diverses eines informàtiques. Obtenim el nostre contig de la base de dades EMBL. En primer lloc localitzem els elements repetitius i obtenim la seqüència emmascarada. Seguidament fem la predicció de gens 'ab initio' mitjançant els programes Geneid i Genscan. Per tal de validar els resultats obtinguts comparem la nostra seqüència a una base de dades amb EST's humans coneguts. Com a segona eina de validació fem servir les seqüències d'aminoàcids que ens proporcionen els programes de predicció de gens. Amb aquestes fem un BLASTP per trobar la proteïna amb més homologia a la nostra. Per tal de visualitzar aquesta homologia fem un aliniament amb Clustalw (EBI) entre la nostra proteïna predita i la proteïna més homòloga. Per tal d'obtenir més informació, extenem la seqüència i tornem a realitzar les prediccions de gens i les validacions amb les comprovacions necessàries. Finalment caracteritzem les proteïnes obtingudes amb Pfam.
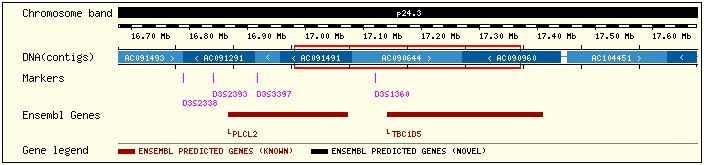
ÍndexLa nostra seqüència es troba al braç p del cromosoma 3 humà. L'hem obtinguda de la base de dades embl i l'hem transformada a format fasta (AC090644.fa) amb la comanda:
grep '^ ' AC090644.embl | sed 's/[ 0-9]//g' > AC090644.fa.
Tot seguit, per poder determinar la llargada i el contingut c+g de la seqüència, la passem a format tabulat (AC090644.tbl).
a: 0.297586
c: 0.200795
g: 0.204186
t: 0.297432
Situació de la seqüència al cromosoma 3 humà:

Per a realitzar les prediccions de gens només ens interessa tenir present aquella part del DNA que sigui codificant, que en realitat és una petita part del DNA genòmic total.
Gran part del DNA no codificant està formada per elements repetitius. Ens interessa eliminar-los per tal que no interfereixin a les nostres prediccions ja que ens conduirien a errors.
Emmascarem els elements repetitius utilitzant el programa Repeat Masker. Aquest programa llença la nostra seqüència en format fasta contra una llibreria d'elements repetitius. Així obtenim:
El tant per cent de seqüències repetitives que han sigut emmascarades és d'un 42.38% i es veuen representades en SINEs (9.31%), LINEs (17.61%), LTRs (8.58%), elements de DNA (5.99%), simple repeats (0.44%) i elements de baixa complexitat (0.47%).
A partir de l'arxiu del llistat dels elements repetitius podem visualitzar les repeticions obtingudes seguint els següents passos:
Una vegada eliminats els elements repetitius introduim la seqüència emmascarada en format fasta als programes de predicció de gens GENEID i GENSCAN els quals ens donen una primera idea dels possibles gens que podem tenir a la nostra seqüència. Cal tenir present que la seva fiabilitat és de només un 50%.
L'execució de GENEID ens proporciona un output en format gff.
GENSCAN, en canvi, no produeix un output gff. Per poder visualitzar la predicció amb el programa gff2ps_v0.98 cal tenir els resultats en format gff (genscan.gff). L'obtenim amb la següent comanda:
gawk 'BEGIN{OFS="\t"}$2 ~ /Term|Intr|Init/ {print "AC090644", "genscan", $2, start=($4<$5 ? $4 : $5), end=($5<$4 ? $4 : $5),$13,$3,$7, $1}' AC090644.genscan | sed 's/\.[0-9][0-9]$//' > AC090644.genscan.gff
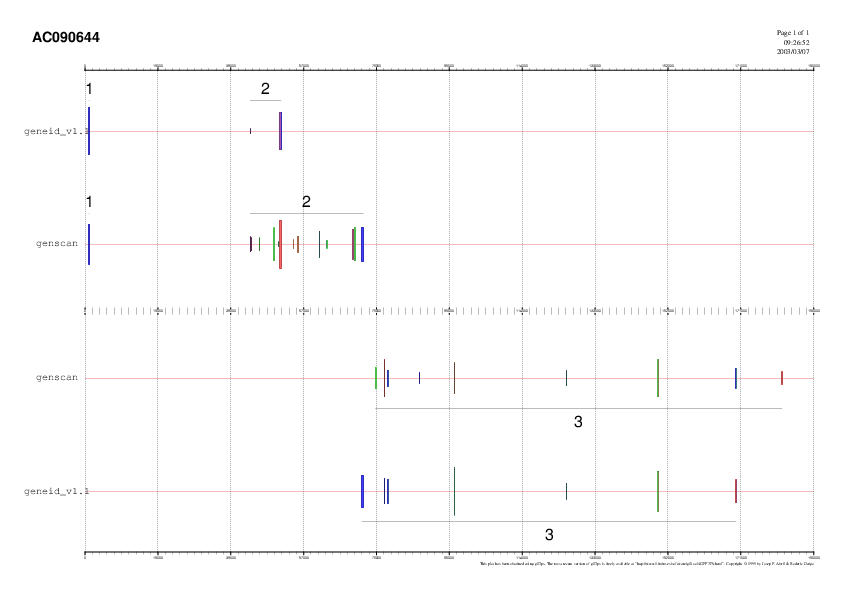
Amb GENEID obtenim tres gens: el gen 1 (un exó terminal), el gen 2 (dos exons: un inicial i un terminal) i el gen 3 (set exons: sis interns i un terminal). Amb GENSCAN visualitzem també tres gens amb algunes petites diferències: el gen 1 (un exó terminal), el gen 2 (tretze exons: un inicial, onze interns i un terminal) i el gen 3 (nou exons: un inicial, set interns i un terminal).
ÍndexEls ESTs són petites seqüències de RNAm que han estat seqüenciades una vegada s'han passat a cDNA per retrotranscripció. Per tant, podem aliniar-les amb la nostra seqüència genòmica per tal que ens ajudin a valorar la nostra predicció de gens. NCBI: Utilitzem el Megablast per obtenir el llistat de ESTs amb homologia amb la nostra seqüència i tot seguit el programa parseblast (J. Abril) per tal d'obtenir el format gff. L'execució del parseblast la realitzem amb la següent comanda:
Per a realitzar búsquedes de ESTs humans hem utilitzat dues fonts d'informació diferents: Bases de dades de NCBI i de tigr.
parseblast -Gi AC090644.est | gawk 'BEGIN{OFS="\t"}{$2=$9;$6=$7=".";print}' > AC090644.est.gff
Seguidament utilitzem el programa gff2ps_v0.98c (Abril & Guigó, 2000) per visualitzar els resultats en format postscript mitjançant ghostview
Obtenim el següent gràfic de la visualització.
De tots aquests ESTs volem considerar els que hagin patit splicing ja que només aquests seran fiables per recolzar les nostres prediccions. Aquests hsp's apareixen més d'una vegada al fitxer obtingut en format gff. Per això, per seleccionar-los utilitzem el programa awk script i l'executem amb la següent comanda:
gawk -f getsplicedhsp.awk AC090644.blast.est.gff > AC090644.blast.est.spliced.gff
Visualitzem els resultats i obtenim el següent gràfic.
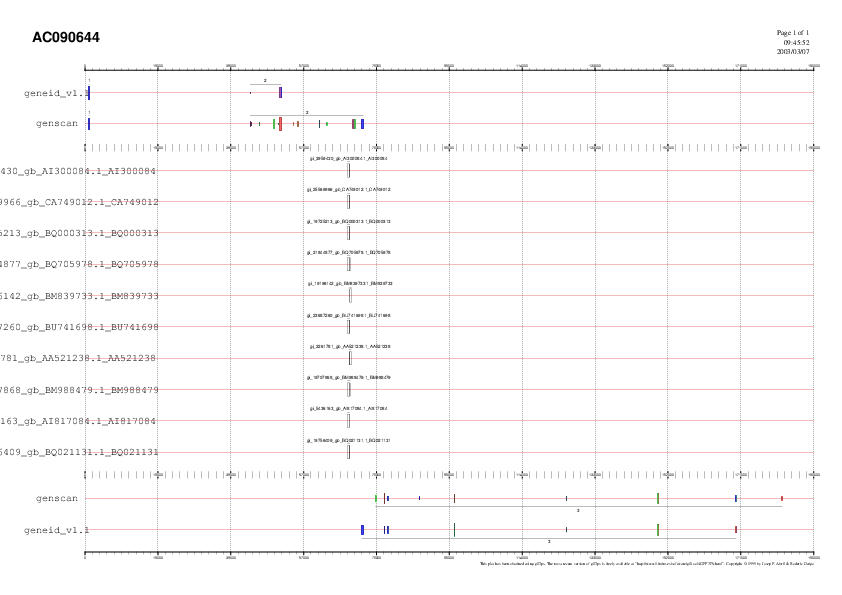
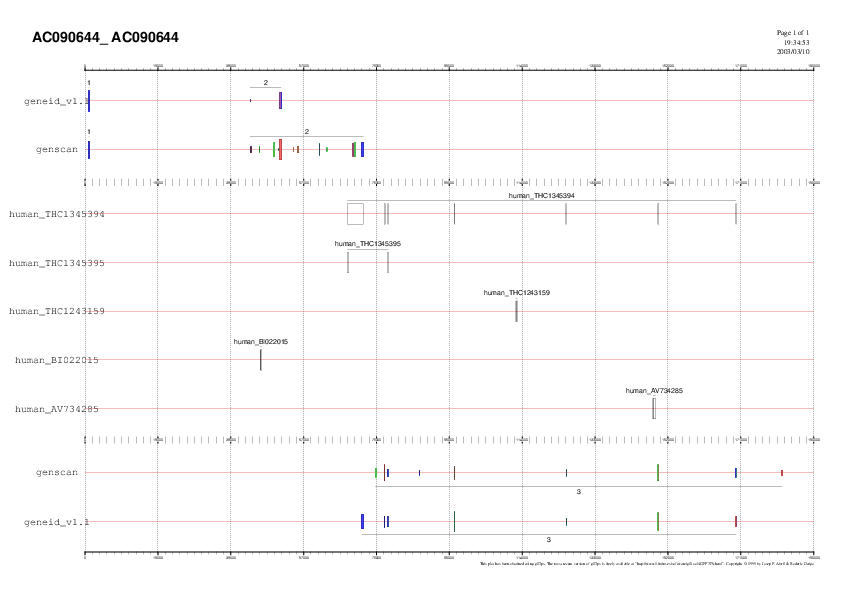
Tot seguit integrem tota la informació present fins ara. Visualitzem els resultats obtinguts als programes de predicció de gens amb els ESTs amb splicing i obtenim el següent gràfic:
TIGR: La base de dades de TIGR (The Institute For Genomic Research) conté informació d'anàlisis estructurals, funcionals i comparatius del genoma i els seus productes gènics en varis organismes.
TIGR Gene Indices ens permet realitzar un blast de la nostra seqüència contra la seva base de dades de ESTs humans. Hem obtingut nous ESTs i hem seleccionat aquells que havien patit splicing amb el mateix procés que en el punt anterior.
Tot seguit fem un nou gràfic amb els resultats de GENEID i GENSCAN i aquests nous ESTs:
| POSICIONS DELS NUCLEÒTIDS | |||
| EST HUMAN_THC1345394 | EXONS GEN 3 | ||
| Exó terminal | 68401-72499 | 72202-72495 | |
| Exó intern 1 | 78986-79068 | 78988-79067 | |
| Exó intern 2 | 77278-78282 | 78006-78167 | |
| Exó intern 3 | 96342-96447 | 96348-96447 | |
| Exó intern 4 | 125414-125613 | 125446-125609 | |
| Exó intern 5 | 149351-149663 | 149402-149658 | |
| Exó intern 6 | 169744-169832 | 169745-169830 | |
Una altra forma de validació de les nostres prediccions inicials és comprovar que les seqüències d'aminoàcids predites corresponen a proteines reals i per a fer-ho utilitzem BLASTP.
Per tal de mostrar els resultats de les homologies de forma més clara i directa hem creat la següent taula amb les proteïnes amb grau més alt d'homologia predites pels dos programes de predicció:
De les proteïnes predites per GENEID obtenim les següents homologies:
Amb GENSCAN obtenim les següents homologies:
PROTEÏNA E-VALUE
PROTEÏNA 1 GENEID Fosfolipasa C-like 2 i KIAA1092 protein 1e-28 i 2e-28
PROTEÏNA 2 GENEID Similar to over-expressed breast tumor protein 0.006
PROTEÏNA 3 GENEID Similar to a putative protein coded in C. elegans cosmid B0393 e-162
PROTEÏNA 1 GENSCAN Fosfolipasa C-like 2 i KIAA1092 protein 2e-27 i 2e-27
PROTEÏNA 2 GENSCAN Hypotetical protein (No humana) 1,7
PROTEÏNA 3 GENSCAN Similar to a putative protein coded in C. elegans cosmid B0393 e-116
Verifiquem les homologies obtingudes amb el programa d'aliniament múltiple ClustalW, introduint les proteïnes predites per GENEID i GENSCAN amb les proteïnes reals més similars obtingudes amb BLASTP. D'aquesta manera, visualitzem a quina part de la proteïna sencera correspon la nostra homologia.
Amb GENEID:
Amb GENSCAN:
Amb tota la informació que tenim fins ara predim tres gens a la seqüència AC090644. El gen 2, que no ha sigut validat per cap EST ni per BLASTP; i els gens 1 i 3 que estan incomplets. Amb la intenció d'obtenir més informació sobre aquests gens obtem per extendre el nostre contig cap a 3' i 5'. D'aquesta manera intentarem confirmar les proteïnes predites anteriorment, amb el gen sencer.
Índex
Per extendre el nostre contig utilitzem la base de dades UCSC Genome Bioinformatics que conté informació dels genomes humà, de ratolí i de rata. Amb el Genome Browser d'aquesta base de dades obtenim la seqüència AC090644 extesa.
El nostre contig està situat al cromosoma 3 i va de la posició 16868721 a la 17472962.
Per obtenir una nova predicció i validació de gens amb el contig extès realitzem:
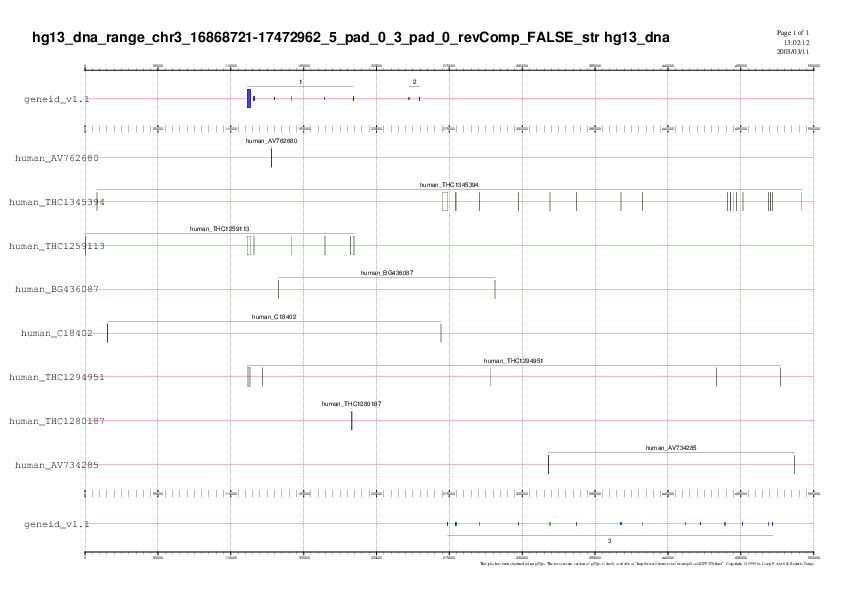
Amb la seqüència extesa hem aconseguit amplificar els gens 1 i 3; a tots dos gens apareixen nous exons interns encara que no hem pogut obtenir l'inicial en cap d'ells.
En quant als ESTs han aparegut nous ESTs recolzants i han desaparegut els menys fiables.
Veiem que el gen 2 no queda suportat. Unint aquesta informació amb els resultats obtinguts a la seqüència sense extendre (ESTs i homologia proteica nuls) afirmem que es tracta d'un gen no predit anteriorment o d'una predicció errònia.
Els nous ESTs: human_THC1259113 i human_THC1294951 poden donar evidències de recolzament pel gen 1.
En quant al gen 3 sembla estar suportat per l'EST human_THC1345394 i potser per l'EST human_THC1294951.
El gen 1 codifica per les proteïnes PLC i/o KIAA1092 amb un E-value de zero. D'aquesta manera comprovem les prediccions anteriors de GENEID i GENSCAN sense extendre i afirmem que el gen 1 ha patit splicing alternatiu o bé que les dues proteïnes pertanyen a una mateixa família i són molt similars. Per això és possible obtenir un E-value igual a zero per a les dues sense que el gen 1 codifiqui per ambdues. Aquesta segona possibilitat ha estat reforçada posteriorment al comprovar que la proteïna KIAA1092 és una fosfolipasa.
El gen 3 codifica per una proteïna similar a una proteïna putativa codificada pel cosmid B0393 de C. elegans amb un E-value de zero. Així millorem la nostra predicció anterior ja que augmenta la longitud de la correspondència.
Hem aliniat la proteïna predita pel gen 1 amb les dues més homòlogues (KIAA1092 i PLC). L'aliniament ha donat uns scores de 99 entre les dues proteïnes més homòlogues, 97 entre la PLC i la nostra predicció i 99 entre la KIAA i la nostra predicció. Observant l'aliniament veiem una molt bona correspondència malgrat que l'exó inicial no queda reflectat.
En quant a la proteïna predita pel gen 3 (Similar to a putative protein coded in C. elegans cosmid B0393) l'aliniament amb la nostra predicció ens dóna un score de 85; l'exó inicial tampoc queda reflectat.
Així doncs, comprovem que les proteïnes que havíem predit anteriorment amb la seqüència sense amplificar, són les mateixes que ha predit ara el BLASTP. Amb l'extensió augmentem el nombre d'aminoàcids que s'alinien al Clustalw.
Índex Degut a la poca fiabilitat dels programes de predicció de gens i per tal de millorar els nostres resultats, utilitzem el programa est2genome.
est2genome és un programa per aliniar EST amb seqüències de DNA genòmic. Aquest aliniament ens ajuda a millorar les prediccions dels gens per homologia de les seqüències. L'output que ens proporciona conté una llista dels introns i els exons que troba a la seqüència i la predicció de la direcció de la transcripció dels gens.
Hem realitzat un aliniament de la nostra seqüència emmascarada amb els tres ESTs obtinguts de la base de dades tigr que ens han semblat millors (human_THC1345394, human_THC1259113, human_THC1294951). Els resultats que ens ha proporcionat el programa són els següents:
Amb aquests resultats afirmem que l'EST human_THC1345394 revela que el millor aliniament es dóna entre aquest EST en revers i la seqüència genòmica, reforçant dos gens en revers.
L'EST human_THC1259113 ens mostra que el millor aliniament és entre aquest EST en forward i la seqüència genòmica reforçant el nostre gen 1 en forward.
I, ja per últim, l'EST human_THC129495 revela que el millor aliniament es dóna entre aquest EST en forward i la seqüència genòmica reforçant el gen 1 en forward.
En els resultats obtinguts, també veiem que els valors d'homologia dels introns prenen un valor de zero i els dels exons, en canvi, gairebé tots tenen un valor de 100.
Els resultats de l'est2genome tenen en compte l'ordre correcte dels ESTs de manera que augmenta la fiabilitat de la predicció de gens.
Per a realitzar la predicció de gens només ens interessa la informació dels exons (material codificant) i a més ho volem en format gff per a poder executar el geneid. Obtenim els exons en format gff amb la següent comanda:
grep Exon est2genome.THC1345394.outfile.out | gawk '{OFS="\";print"AC090644","est2genome","exon",$4,$5,$3,"-",".","THC1345394"}' > est2genome.THC1345394.outfile.out.gff
A més, cal tenir en compte que el geneid necessita tenir presents les tres possibles frames i per tant hem de construir un gff que contingui totes les possibles pautes de lectura. Això ho aconseguim amb la següent comanda:
gawk '{OFS="\"; for (frame=1;frame>4;frame=frame+1){print"AC090644","est2genome","exon",$4,$5,$3,"-",frame,"THC1345394"}}' > est2genome.THC1345394.outfile.out.geneid.gff
Així, hem obtingut:
Per a poder executar el geneid amb aquestes dades utilitzem la versió v1.1 del geneid obtinguda des del software del geneid imim server. (Cal descomprimir-lo i desempaquetar-lo).
L'execució del programa la realitzem amb les següents comandes:
Utilitzem el programa gff2ps_v0.98c (Abril and Guigó, 2000) per a visualitzar els resultats en format postcript mitjançant el ghostview.
Els resultats són els següents:
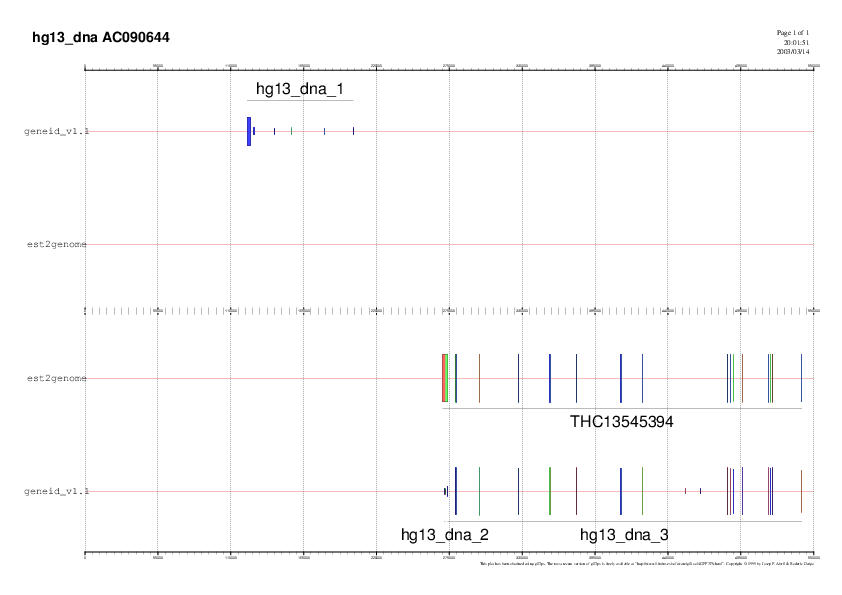
|
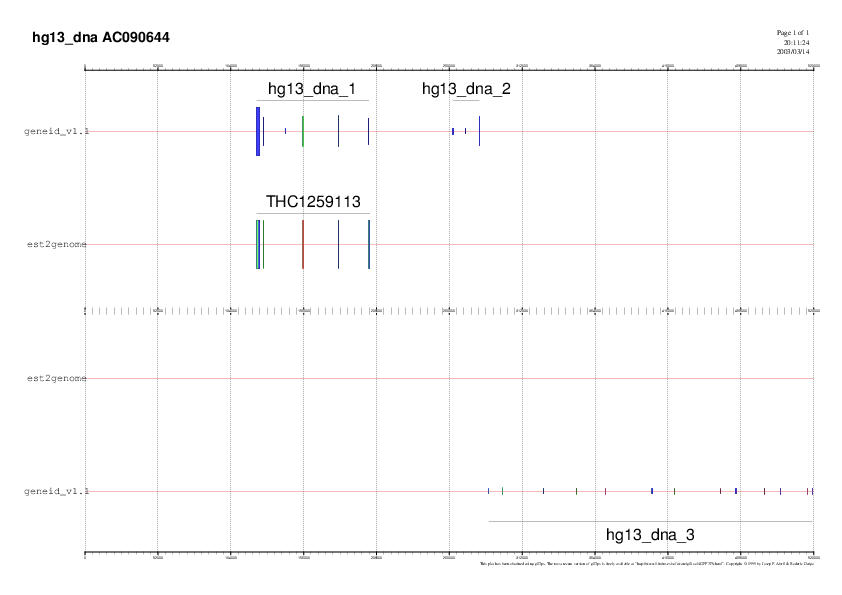
|
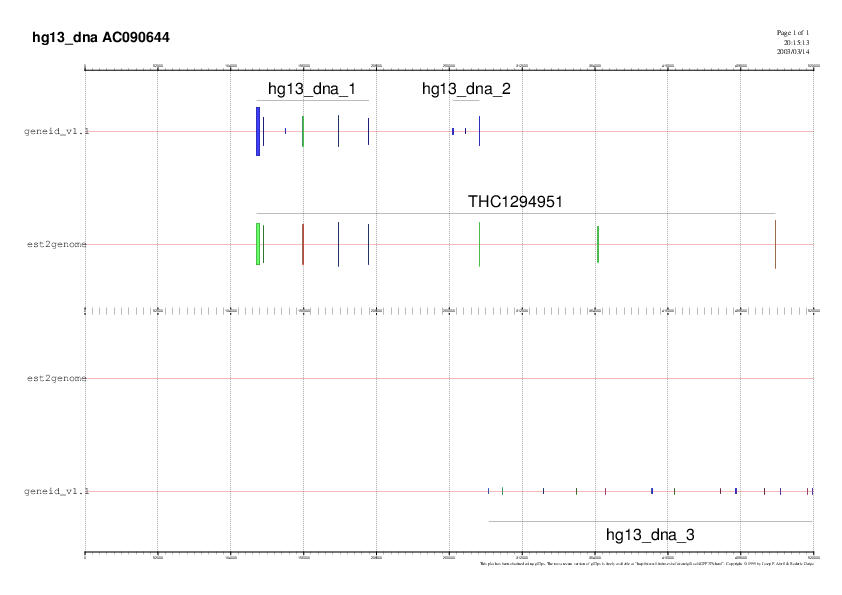
|
| human_THC1345394 | human_THC1259113 | human_THC1294951 |
Ajuntem els resultats de la figura 1 i 2 en una sola figura on queden predits els nostres gens magnificament.
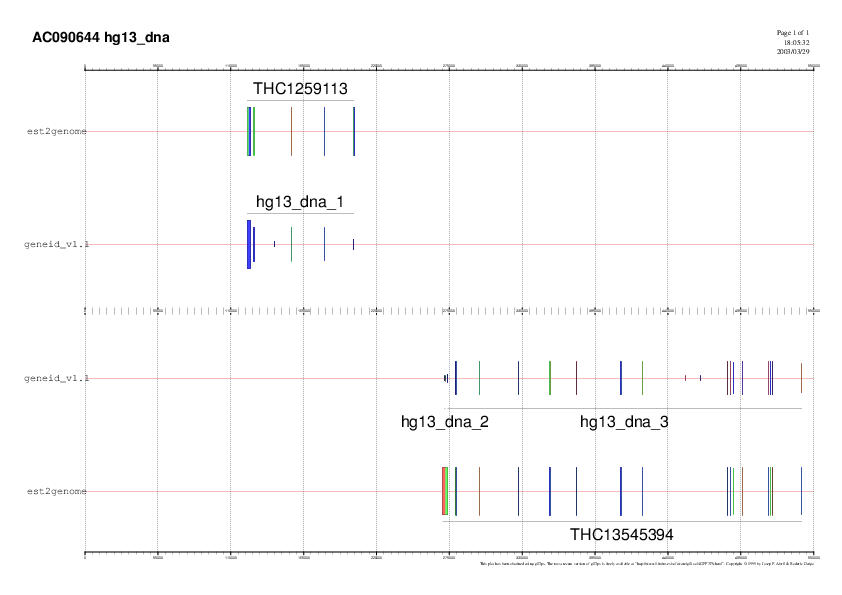
EST human_THC1345394: Aquest EST està recolzant dos gens en revers (hg13_dna_2 i hg13_dna_3). Aquest resultat en un principi ens ha sobtat ja que esperàvem trobar només el gen 3 a la cadena revers. No obstant, hem arribat a la conclusió que aquest gen apareix als resultats de l'est2genome i no als resultats anteriors ja que amb l'aliniament de la nostra seqüència genòmica amb l'EST human_THC134539 hem augmentat els scores dels exons reforçats per aquest EST.
Tot gen té als extrems 3'i 5' unes regions no codificants crucials en la regulació i expressió del gen. Aquestes regions es transcriuen però no es tradueixen; són els anomenats UTR. Aquests UTRs es poden trobar a les seqüències de ESTs
El petit nou gen (hg13_dna_2) predit es situa just a la mateixa posició nucleotídica de la regió més downstream de l'EST que presenta un alt grau d'homologia amb la nostra seqüència.
Amb el que hem dit i veient que el gen 2 no codifica per a cap proteïna concluim que aquest gen ha estat predit a partir de la seqüència d'un UTR. Així doncs, no és un gen real.
EST human_THC1259113: Ens assegura que el gen 1 està suportat.
EST human_THC1294951: Tot i que sembla recolzar el gen 1 com havíem suposat anteriorment, els resultats obtinguts amb est2genome no encaixen amb els del geneid. Ha aparegut un nou gen (hg13_dna2) a la cadena forward; aquest gen no és real ja que queda totalment refutat tant amb l' EST human_THC1259113 (homologia molt alta) com amb els resultats obtinguts amb els dos ESTs comentats anteriorment. Mentres que les prediccions dels dos primers ESTs encaixen perfectament en una sola teoria, aquest tercer EST ens suposaria soroll, amb el que obtem descartar la seva informació a les prediccions finals.
Per tant, resumint, validem l'existència d'un únic gen a la cadena forward que es correspon amb el gen 1 i un únic gen a la cadena revers que es correspon amb el gen 3.
Per tal de caracteritzar les proteïnes predites pels nostres dos gens hem utilitzat la base de dades Pfam. Proteïna predita pel gen 1
Proteïna predita pel gen 2 En aquest treball, després d'utilitzar diferents eines bioinforàtiques, hem analitzat de la forma més fiable possible la seqüència AC090644 del cromosoma 3 humà.
Andrea Edo Guimerà andrea.edo01@campus.upf.es Agraïments al Grup de Recerca d'Informàtica i Biomèdica de IMIM/UPF, i en especial a en Genís Parra i a en Josep Abril per la seva gran ajuda i paciència. Gràcies!
Servidors Programes
Pfam conté informació sobre dominis conservats en famílies de proteïnes i la seva representació com a matrius de pesos (perfils). Llancem la seqüència de la nostra proteïna predita contra aquestes matrius per identificar similaritat a dominis coneguts. Si els dominis no són de funció coneguda segurament no els trobarem.
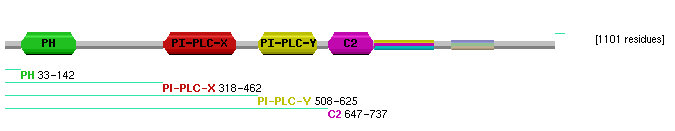
Hem llançat la seqüència d'aminoàcids de la proteïna predita pel gen 1 i hem vist que presenta quatre dominis. El domini PH, el PI-PLC-X, el PI-PLC-Y i el C2.
El domini PH, format per aproximadament uns 100 residus, és present en moltes proteïnes de funció relacionada amb la senyalització intracel.lular o com a constituents del citoesquelet. Presenta un E-value de 9.1e-08 està situat dels residus 33 al 142.
El domini PH-PLC-X s'associa amb el domini PH-PLC-Y per a formar una única unitat estructural, l'enzim de la fosfolipasa C específica de fosfatidilinositol (enzim eucariota intracel.lular). Aquest enzim té un paper important en els processos de transmissió de senyals i és regulat de forma reversible per fosforilació i unió de proteïnes reguladores. Els dominis PI-PLC-X i PI-PLC-Y tenen un valor de E-value de 9.9e-69 i 3.4e-48 respectivament i estàn situats dels residus 318 al 462 i del 508 a 625 respectivament.
El domini C2, d'uns 116 residus d'aminoàcids, està relacionat en unions de fosfolípids depenents de calci, tot i que es suposa que ha de tenir altres possibles funcions ja que s'ha observat que també està relacionat amb proteïnes que no uneixen calci. Té un E-value de 5.5e-24 està situat dels residus 647 a 737.
En quant a la seqüència d'aminoàcids de la proteïna predita pel gen 2 hem vist que presenta un sol domini de coincidència, el domini TBC. Aquest domini fou identificat en llevat i actua com a activador de GTPases.
La coincidència va del residu 47 al 415 i el valor del seu E-value és de 1.5e-09.
Conclusions
La contínua dualitat entre acceptació/refutació dels resultats obtinguts al llarg del treball, ens ha conduït a la predicció final.
En aquesta, concluim que el nostre contig (amb la pertinent amplificació) conté dos gens.
El primer gen es troba situat a la cadena forward i està format per sis exons, cinc interns i un terminal; codifica per una fosfolipasa. El segon gen està a la cadena revers i conté tretze exons, dotze interns i un terminal; codifica per una proteïna activadora de GTPases.
Gràcies a les validacions per homologia de proteïnes i la utilització de est2genome hem pogut refutar l'existència del gen 2. Aquest, havia estat predit 'ab initio' amb GENEID i GENSCAN encara que ja mostrava poca fiabilitat als programes de predicció de gens.
Autores i agraïments
Montserrat Ferrer Batallé montserrat.ferrer01@campus.upf.es
Software i bibliografia electrònica
embl
Repeat Masker
Megablast
GENEID
GENSCAN
TIGR
BLASTP
ClustalW
EST2GENOME
Pfam
gff2ps_v0.98
awk script
Parseblast (J. Abril)
Ghostview
