Introducció
Selenoproteïnes
Les selenoproteïnes són aquelles proteïnes que incorporen una selenocisteïna en la seqüència del seu gen i solen tenir funció com a proteïna redox. Aquestes selenocisteïnes tenen una estructura molt semblant a les cisteïnes: la diferència és que contenen un àtom de seleni en comptes d"un radical sulfur. El seleni és un element molt important per als éssers vius i la seva manca pot produir malalties. Alhora, però, el seu excés pot ser molt tòxic. En el genoma d'humans en trobem unes 25.
Selenocisteïna
La selenocisteïna és considerada l'aminoàcid número 21 i sovint s'abrevia com a SeC o U. Es troba molt extesa entre bacteris, arquees i eucariotes, únicament amb l'excepció del regne vegetal, on és absent. El codó que codifica per les selenocisteïnes és el UGA. Aquest normalment funciona com a codó Stop, però en el cas de les selenoproteïnes forma l'aminoàcid selenocisteïna. A l'extrem 3' d'aquests gens trobem una estructura característica anomenada SECIS (seleno cystein insertion sequence).
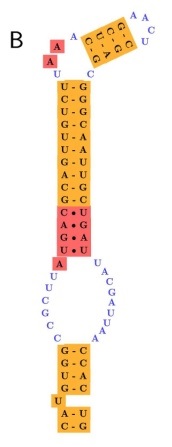
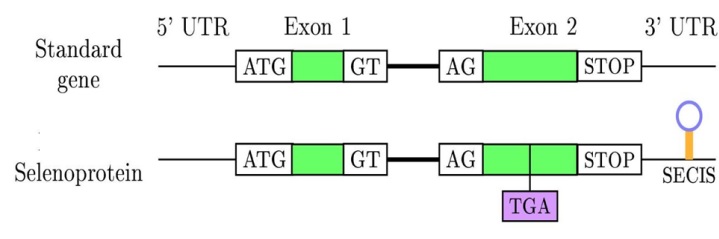
Codificació de l'aminoàcid
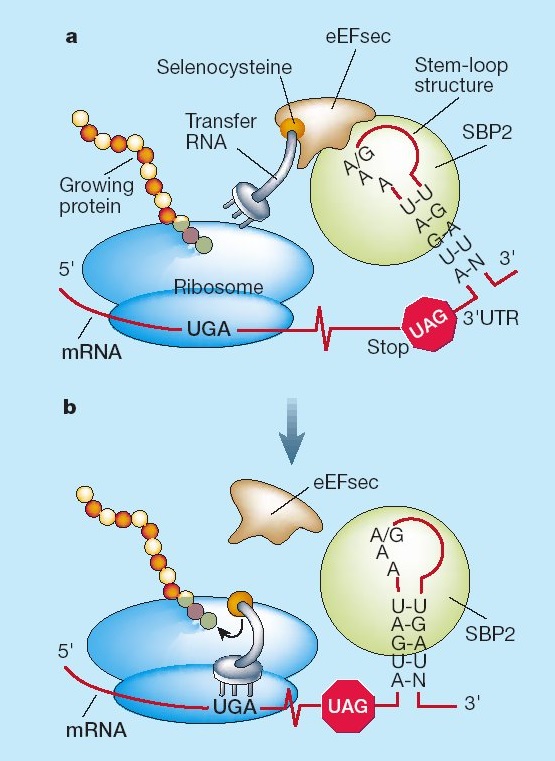
El mecanisme per el qual el codó és recodificat està mediat per la seqüència SECIS.
Quan els ribosomes troben un triplet UGA continuen la traducció fins el següent codó stop. L'estructura SECIS permet la unió de SBP2 (Secis binding protein 2) que, alhora, recluta un tRNA que és específic per la selenocisteïna i continua la traducció fins el següent codó stop. Després és necessari un factor d'elongació eEFSec per tal de sintetitzar la proteïna.
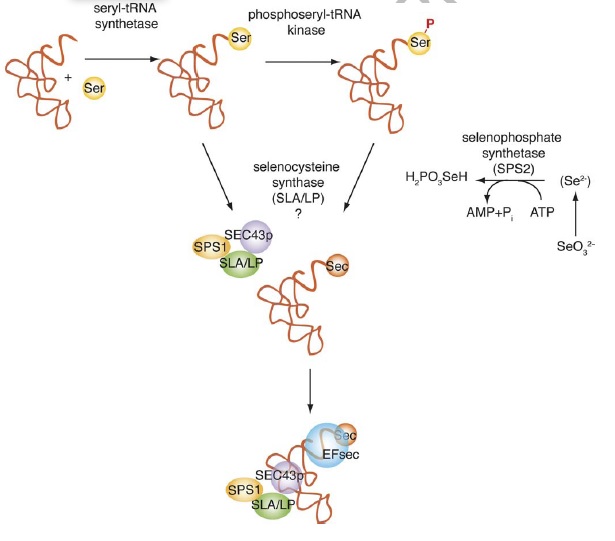
Síntesi de la selenocisteïna
A part de les proteïnes que participen en la síntesi de selenoproteïnes, n'hi ha que participen en la síntesi de l'aminoàcid. La selenocisteïna és l'únic aminoàcid que es sintetitza in situ al seu tRNA (la resta són incorporats després). De fet, el tRNA de la selenocisteïna té inicialment una serina i aleshores un selenofosfat és incorporat per la selenocisteïna sintasa, la qual catalitza la conversió de serina a selenocisteina mitjançant la incorporació del selenofosfat.
L'altra proteïna important és la selenofosfat sintetasa (SPS2) que catalitza la reacció d'òxid de seleni a selenofosfat. Hi ha unes 10 proteïnes de maquinària ceŀlular necessàries perquè un genoma pugui fer selenoproteïnes.
Predicció de selenoproteïnes
Les selenoproteïnes normalment no estan ben predites al genoma perquè en la seva anotació automàtica s'assumeix que UGA és un codó stop. Això suposa un problema.
Una de les primeres aproximacions a la predicció de selenoproteïnes va ser la busca de gens de Drosophila melanogaster que continguessin tant l'element SECIS com el codó UGA.
Ara bé, els elements SECIS, tot i tenir una estructura molt similar (amb loops i hèlix), tenen una baixa conservació de seqüència (gairebé només es conserven dos aminoàcids en el loop i unes 6 posicions a l'hèlix) i això va suposar un problema. S'ha de tenir en compte que si la seqüència total és d'aproximadament 100 nucleòtids, tenir només 7 o 8 nucleòtids conservats no permet identificar aquests elements correctament.
Va sorgir la idea de buscar estructures similars, mitjançant programes capaços de comparar patrons com PatScan. Aquests programes buscarien en el genoma una regió amb hèlix, loop intern, una altra hèlix i un loop apical. Plegarien un conjunt de seqüències computacionalment i veurien si s'ajusta a aquesta estructura.
Tot i així, aquests mètodes eren massa poc específics i la quantitat de SECIS potencials trobats era molt gran. Per tant, el següent pas va ser utilitzar el programa Geneid, que buscava tots els exons interromputs per un UGA. Eliminava la norma que considerava UGA un codó de terminació del programa.
Per trobar les selenoproteïnes s'havia de barrejar, doncs, la informació de PatScan (que predeia els elements SECIS) i de Geneid (que permetia trobar els exons interromputs per un codó UGA). D'aquesta manera, es considerarien selenoproteïnes aquelles regions del genoma amb un element SECIS en l'extrem 3' precedit per un codó UGA, el qual s'hauria de trobar en una regió codificant per proteïna.
En la imatge següent, l'element encerclat en vermell seria un bon candidat per ser una selenoproteïna, ja que compleix les dues condicions: té un element SECIS en l'extrem 3' (en taronja) i està precedit per un codó UGA (en vermell) que es troba en una regió codificant per proteïna (en blau).
D'altra banda, l'aproximació que es va seguir per trobar les selenoproteïnes en humans va ser diferent. Com que el moment en què es va portar a terme el projecte de la seqüenciació del genoma humà també es va fer per altres organismes com el ratolí, es van comparar els elements SECIS de les selenoproteïnes que ja es coneixien en humans amb les de ratolí. Es va observar que hi havia una gran conservació entre elements SECIS de ratolins i humans (més del 95%). Per tant, una bona aproximació seria predir tots els elements SECIS a humans i a ratolí (dos espècies properes filogenèticament) i quedar-se amb els que tenen més d'un 95% de conservació.
Això, però, no es podia aplicar en genomes com el de Fugu: al no tenir cap espècie propera de la qual es conegués la seqüència del genoma, aquesta aproximació no era vàlida. En aquest cas, el que es va fer va ser predir tots els gens interromputs per UGA en humans i Fugu i fer l'alineament d'aquests gens, perquè s'havien adonat que en els alineaments de selenoproteïnes hi havia una gran conservació, que continuava després del codó UGA (no passa si aquest funciona com a codó stop). Per tant, després de fer l'alineament dels possibles candidats dels dos genomes, es predeia com a selenoproteïna aquelles seqüències conservades després del codó UGA.
Selenoproteïnes en eucariotes
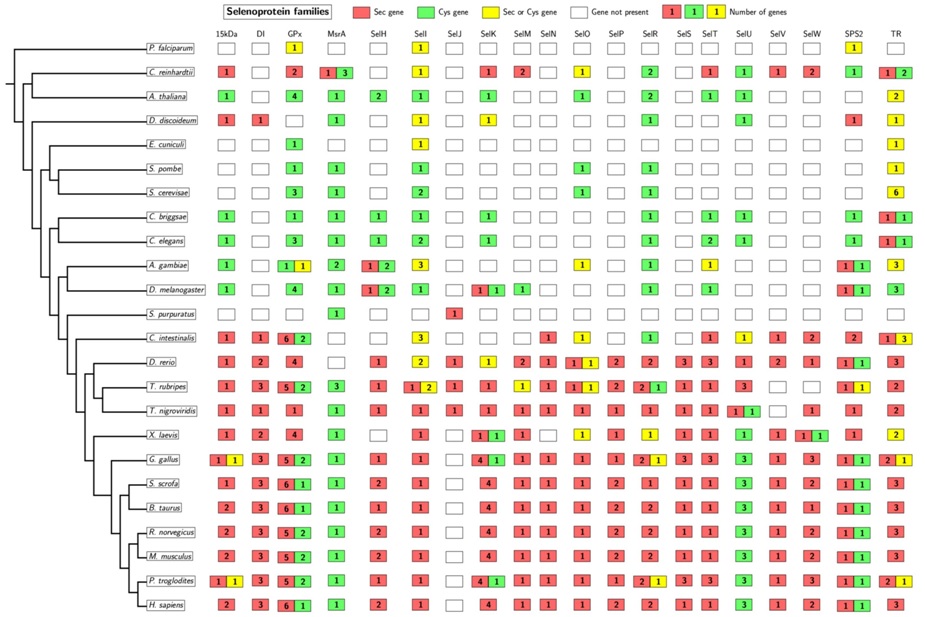
En aquest gràfic observem com el nombre de selenoproteïnes augmenta a mesura que augmenta la complexitat de l'organisme. També és important destacar la presència d'homòlegs en cisteïna, és a dir, proteïnes semblants a les selenoproteïnes però que enlloc d'una selenocisteïna presenten una cisteïna en la mateixa posició. En aquesta imatge, en cas que sigui una selenoproteïna ho veiem marcat de color vermell i en cas que sigui un homòleg en cisteïna, de color verd. Si no està pintat de cap color vol dir que aquell gen no es troba present en aquell organisme.
En la següent imatge observem quines selenoproteïnes trobem en els vertebrats. Veiem com han aparegut duplicacions (en verd), deleccions (en gris) i canvis de selenocisteïnes per cisteïnes. Aquest arbre ens permet fer una predicció de quines selenoproteïnes esperem trobar en un genoma en concret.
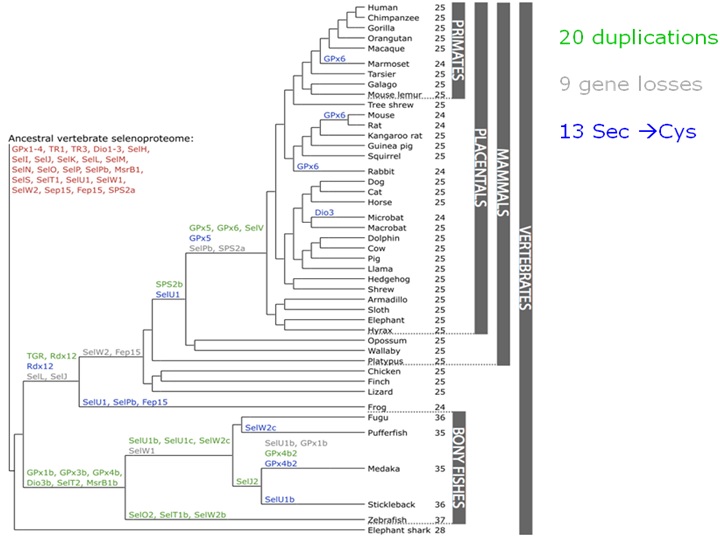
Segons aquesta filogènia, en humans esperaríem trobar: GPx1-4, TR1, TR3, DI1-3, SelH, SelI, SelK, SelM, SelN, SelO, SelU1, SelV, SelP, MsrB1, SelS, SelT1, SelW1, Sep15, SPS2b, TGR i Rdx12. Tambè homòlegs en cisteïna com SelU1, GPx5 i GPx6.
Les selenoproteïnes conegudes en humans (trobades a la base de dades SelenoDB) són: Sel15, GPx1, GPx2, GPx3, GPx4, GPx5, GPx6, GPx7, GPx8, DI1, DI2, DI3, MsrB1, SPS, SelH, SelI, SelK, SelM, SelN, SelO, SelP, SelR1, SelR2, SelR3, SelS, SelT, SelV, SelW i TR.
Les proteïnes de la maquinària de seleni són: eEFSec, MsrA, PSTK, SBP2, SecS, i SECp43.
Les proteïnes de la maquinària de seleni (trobades a la base de dades SelenoDB) són: eEFSec, MsrA, PSTK, SBP2, SecS, i SECp43.
Chrysochloris asiatica : el talp daurat del Cap
El Chrysochloris asiatica és anomenat comunament talp daurat del Cap. És un petit mamífer insectívor sense cua que té un pelatge curt, tupit i llustrós de color oliva, marró o gris segons la incidència de la llum. La seva longitud és d'entre 9 i 14cm. És una espècie solitària que no es comunica amb els individus de la seva mateixa espècie a no ser que s'hagi de reproduir o demanar socors.

Aquest talp és endèmic d'una petita regió de Sud-àfrica; es troben des de la regió més del sud de Western Cape fins la regió nord del Northern Cape. Viuen en túnels subterranis en una gran varietat de sòls, que van des de sorrencs fins a sòls humits barrejats amb argila. Els talps actualment es troben dins la llista d'espècies amenaçades a causa de la destrucció del seu hàbitat. Es troben amb més freqüència en zones urbanes que a terres rurals.
Com que viuen a la foscor, la taxa de renovació d'aigua, la taxa metabìlica basal i la freqüència cardíaca són molt baixes. Són sensibles a les fluctuacions de temperatura ambient a causa de la seva relació superfície-volum gran, petita quantitat de greix corporal i intercanvi de calor ràpid amb el medi ambient; en conseqüència, són actius durant el matí i la nit, que són les parts del dia més fresques.
Els talps daurats del Cap tenen comunicació acústica i química: només tenen desenvolupats els sentits de l'oïda i l'olfacte. Són capaços de percebre la vibració produïda per preses, depredadors i altres pertorbacions de les ones sísmiques.
L'aparellament en aquests animals no està gaire estudiat, però s'hipotetitza que es dóna a través de comunicació de llarga distància via ones sísmiques. La seva reproducció es dóna durant tot l'any, però es veu afavorida durant els mesos d'estiu, degut a la major quantitat preses que els aporta major quantitat d'energia. Solen tenir dues cries per ventrada.
Classificació taxonòmica del talp daurat del Cap
- Regne: Animalia
- Filum: Chordata
- Classe: Mammalia
- Ordre: Afrosoricida
- Família: Chrysochloridae
- Gènere: Chrysochloris
- Subgènere: Chrysochloris
- Espècie: C. asiatica
Selenoproteïnes del talp daurat del Cap
El talp daurat del Cap no apareix a la imatge de selenoproteïnes en vertebrats perquè aquestes encara no han estat descrites en aquest animal. És per això que per predir quines selenoproteïnes esperem trobar en el nostre genoma, hem de conèixer la filogènia del nostre animal.
Com hem vist anteriorment, el talp daurat del Cap és un mamífer que es troba dins l'ordre dels Afrosoricida. En aquest ordre trobem tant el talp daurat com el tenrec (Echinops telfairi). Per aquesta raó, el tenrec pot ser considerat el millor animal de referència per predir quines selenoproteïnes té el talp.
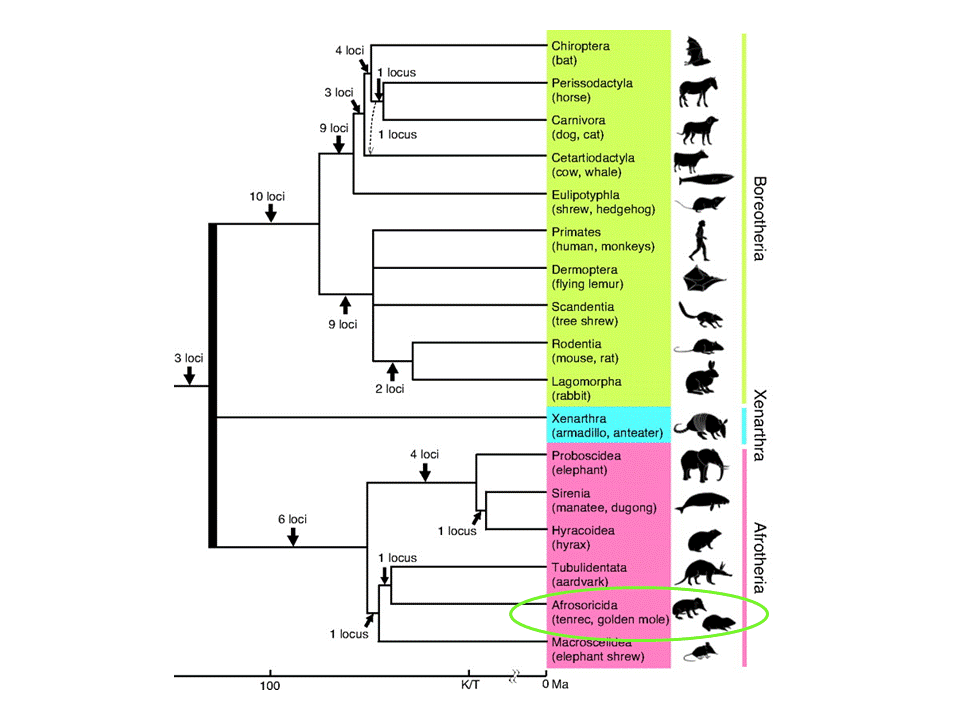
Per tant, seguint la filogènia de les selenoproteïnes podem veure que E. telfairi té proteïnes semblants a les humanes i que, per tant, passarà el mateix amb Chrysochloris asiatica. Si ens fixem amb les selenoproteïnes i proteïnes de la maquinària del seleni que es coneix en el tenrec, podem predir que en el talp daurat del Cap trobarem:
- - Les selenoproteïnes: Sel15, GPx1, GPx2, GPx3, GPx4, GPx5, GPx6, GPx7, GPx8, DI1, DI2, DI3, SPS, SelH, SelI, SelK, SelM, SelN, SelO, SelP, SelR1, SelR2, SelR3, SelS, SelT, SelV, SelW i TR.
- - Homòlegs en cisteïna: GPx5 i SelU1.
- - Proteïnes de la maquinària del seleni: eEFSec, MsrA, PSTK, SBP2, SecS i SECp43.