Conclusiones
Una vez analizados los genomas de los protistas anotados durante los años 2008, 2009, 2010 y 2011 podemos concluir que la selenoproteína dsbA está más conservada en protistas que la selenoproteína I. Dsba se encuentra presente en los protistas principalmente como homóloga en cisteína u otros aminoácidos, pero en algunos casos también hemos encontrado protistas que presentan la selenoproteína.
A continuación mostramos los resultados obtenidos para cada una de las familas de selenoproteínas que hemos analizado divididos por años.
2011
| dsbA C.intestinalis | dsbA P.ingrahamii | dsbA A.gypseum | SelI H.sapiens | SelI M.musculus | SelI P.infestans | |
|---|---|---|---|---|---|---|
| Homólogos | P.tricornutum |
|
|
|
|
|
|
|
|
|
|
|
|
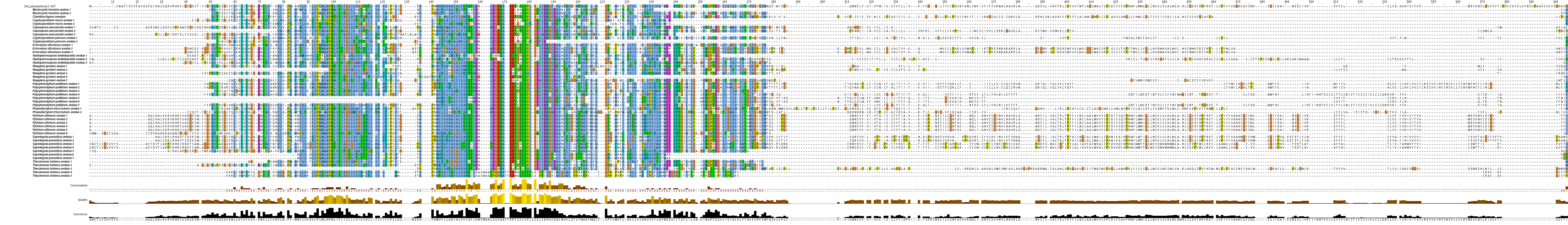
Para todos los protistas de 2011 hemos hecho un alineamiento múltiple de las distintas queries contra todos los hits obtenidos en el tBLASTn con los que hemos conseguido llegar al Tcoffee. Para poder ver los alineamientos y lo que podemos concluir de ellos haz click aquí.
dsbA
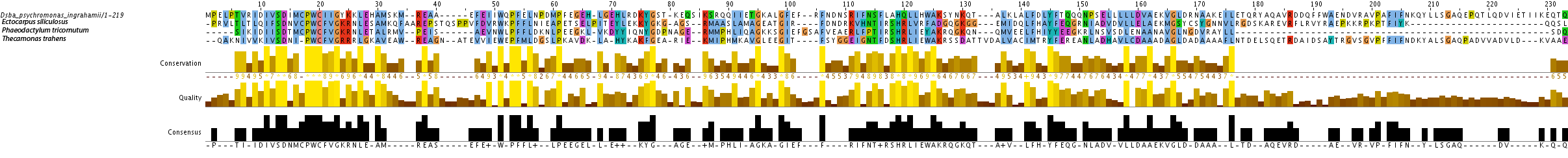
En los genomas de protistas de 2011 hemos encontrado 3 organismos que presentan una proteína homóloga en cisteína. Estos organismos son Ectocarpus siliculosus (1) (2), Naegleria gruberi (1) y Phaeodactylum tricornutum (1) (2) (3) . Usando la query de Ciona intestinalis facilitada por los profesores de la asignatura sólo obtuvimos un homólogo en cisteína, los otros dos resultados lo obtuvimos con las queries que contenían una C en vez de una U.
Thecamonas trahens presenta una selenoproteína (1) (2): la U de la query alinea con un gap, las regiones flanqueantes al gap están conservadas y en el exonerate el codón correspondiente al gap es un TGA. Al analizar este protista manualmente, con un fastasubseq de 7000 nucleótidos, obtuvimos un gap de varios aminoácidos en la región que contiene la selenocisteína, mientras que con el fastasubseq automatizado de 50000 nucleótidos obtuvimos un único gap alineado con la U de la query y las regiones de antes y después conservadas.
En cuanto a los elementos SECIS, hemos encontrado SECIS en T.trahens con un score de 2.68 usando el patrón loose.
SelI
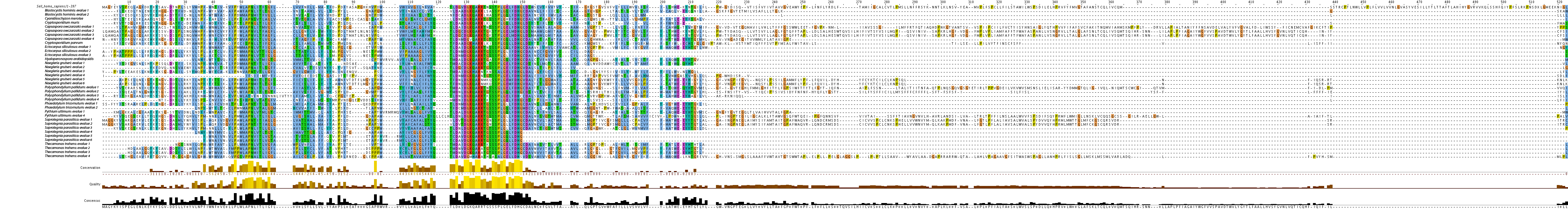
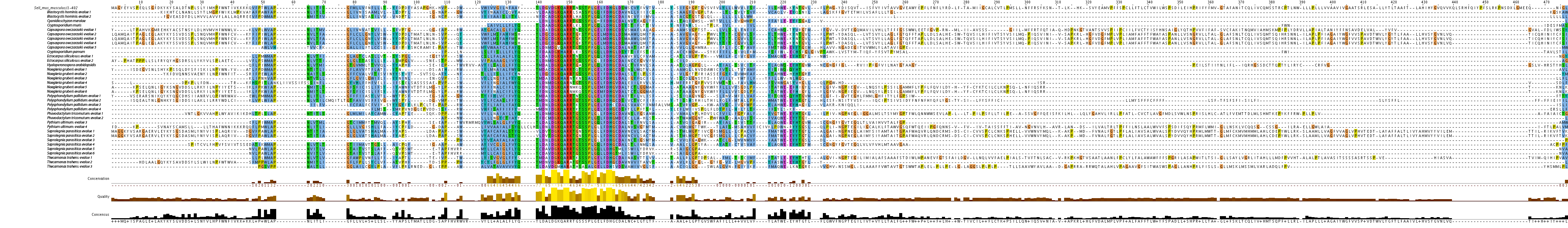
En el análisis de SelI en los genomas de protistas de 2011 no obtuvimos ningún resultado positivo. En todos los organismos obtuvimos una región que alinea muy bien con la query pero, al hacer el BLASTp en la base de datos del NCBI, vimos que corresponde a una región que presenta una gran homología con otra enzima que no es una selenoproteína, la CDP-alcohol phosphatidyltransferase. La región conservada en los protistas corresponde al inicio de la selenoproteína I mientras que la región de la query que contiene la selenocisteína no alinea muy bien ni en el tBLASTn ni en el Tcoffee.
Al hacer el BLASTp de Naegleria gruberi contra la base de datos del NCBI obtuvimos un hit con una SelI de Carzapora owczarzaki. Sin embargo, en estos organismos nosotros no obtuvimos ninguna selenoproteína pero en comparación con los Tcoffees de los demás organismos, el Tcoffee de Carzapora owczarzaki era el mejor con diferencia ya que presentaba un gap muy pequeño en la zona de la U (1). En el Tcoffee manual de Naegleria gruberi obtuvimos un gap pequeño en la región que contiene la U pero también hay muchos gaps en otras zonas del alineamiento de la proteína con la query siendo la región más conservada la que corresponde al dominio de homología existente entre la SelI y la CDP-alcohol phosphatidyltransferase. Además, los translates de C.owczarzaki presentan todos numerosos codones de STOP por lo que los Tcoffee que obtuvimos no son buenos.
En lo referente a elementos SECIS, hemos encontrado SECIS en C.owczarzaki con un score de 8.32 usando un patrón default y en N.gruberi con un score de 9.78 usando un patrón default.
Creemos que los protistas Carzapora owczarzaki y Naegleria gruberi pueden presentar selenoproteínas en su genoma aunque no podemos concluirlo con total seguridad debido a que en ningún caso hemos conseguido alinear la U de la query.
2010
| dsbA C.intestinalis | dsbA P.ingrahamii | dsbA A.gypseum | SelI H.sapiens | SelI M.musculus | SelI P.infestans | |
|---|---|---|---|---|---|---|
|
--- | --- | M.brevicollis | --- | --- | --- |
| Selenoproteínas | --- | --- | --- | --- | --- | --- |
dsbA
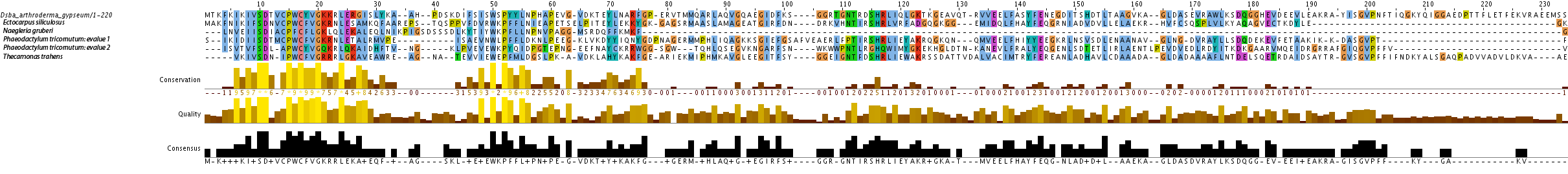
Hemos encontrado un homólogo con C para dsbA en el protista M.brevicollis (1). En T.pseudonana obtuvimos un alineamiento con otro aminoácido, en dos de las queries la U (o C en el caso de las queries homólogas) estaba alineada con una I (1) y en otro caso con una A (2). Éste último protista presenta un homólogo en isoleucina y un homólogo en alanina.
Dsba no había sido estudiada en años anteriores por lo que no podemos comparar nuestros resultados con los de nuestros compañeros.
SelI
En SelI hemos obtenido los mismos resultados que habían obtenido compañeros de otros años, ningún protista presenta esta selenoproteína. Los hits significativos para SelI obtenidos en el alineamiento con el tBLASTn, no alinean la región que contiene la U. Al hacer el BLASTp recíproco en la base de datos del NCBI, los hits con mejores E-values corresponden a la enzima CDP-alcohol phosphatidyltransferase, una enzima que carece de U pero que presenta una gran homología con la selenoproteína I.
2009
| dsbA C.intestinalis | dsbA P.ingrahamii | dsbA A.gypseum | SelI H.sapiens | SelI M.musculus | SelI P.infestans | |
|---|---|---|---|---|---|---|
|
--- | A.anophagefferens | --- | --- | --- | --- |
| Selenoproteínas | E.huxleyi | E.huxleyi | E.huxleyi | --- | --- | --- |
dsbA
Hemos encontrado una selenoproteína en el protista E.huxleyi que presenta la U alineada con un gap (1). En el protista A.anophagefferens obtuvimos un alineamiento de la U con una R en la query de Psychromonas ingrahamii (1) y con las otras queries obtuvimos un gap de 10 nucleótidos. Probablemente este protista presenta un homólogo de dsbA con arginina aunque quizá usando otras queries se obtendrían resultados más concluyentes.
En cuanto a los elementos SECIS, hemos encontrado SECIS en E.huxleyi con un score de 20.32 usando el patrón loose.
SelI
Usando la query de Phytophora infestans sacada de un trabajo del 2008, en el Tcoffee de L.mexicana obtuvimos un gap de 10 nucelótidos mientras que con las otras queries sólo alineaba la región que presenta homología con la enzima CDP-alcohol phosphatidyltransferase. Este resultado nos hace pensar que sería interesante estudiar si este protista presenta SelI usando otra query más ya que puede que nuestros compañeros hubiesen cometido algún error y nosotros estemos partiendo de una query inapropiada.
2008
| dsbA C.intestinalis | dsbA P.ingrahamii | dsbA A.gypseum | SelI H.sapiens | SelI M.musculus | SelI P.infestans | |
|---|---|---|---|---|---|---|
|
--- | --- | --- | --- | --- | --- |
| Selenoproteínas | --- | --- | --- | --- | --- | ¿P.infestans? |
dsbA
Para los protistas de 2008 no hemos obtenido ningún resultado con el tBLASTn. Esto probablemente es debido a que los E-values eran superiores a 0'001 y el programa automatizado sólo considera aquellos hits que tienen un E-value inferior a 0'001.
SelI
En el protista Phytophora infestans, usando la query de Mus musculus obtuvimos un gap de 10 nucleótidos. Al usar la query de Phytophora infestans obtenida de un trabajo de nuestros compañeros de años anteriores obtuvimos un alineamiento en el Tcoffee con un score de 99 y en el que la X correspondiente a la selenocisteína alinea con un gap (1). El problema es que la query usada es del mismo organismo que estamos analizando.
Basándonos en los resultados de nuestros compañeros y considerando que la query de Phytophora infestans es correcta, podemos decir que Phytophora infestans contiene una selenoproteína. Sin embargo, al buscar elementos SECIS no encontramos nada, ni siquiera usando el patrón loose de modo que sospechamos que a lo mejor este protista no presenta selenoproteína y que el resultado de nuestros compañeros es erróneo.
Maquinaria de los protistas de 2011
La maquinaria sólo la hemos analizado para los protistas de nuestro año.
Tal y como está anotado en el apartado de resultados, todos los protistas presentan maquinaria aunque no siempre tienen todas las proteínas. H.arabidopsidis tan solo presenta secp43 y eEFsec y A.taiwanensis solo tiene eEFsec.
En cuanto a la PSTK debemos señalar que no está presente en muchos de los protistas analizados. Esta proteína está involucrada en la fosforilación del tRNAsec y sólo la hemos encontrado en los protistas A.taiwanensis, H.arabidopsidis y S.parasitica en los que, sorprendentemente, no hemos encontrado PSTK.
Alineamientos múltiples
En este apartado mostramos los alineamientos múltiples de las distintas queries usadas con los protistas de 2011 en los que hemos obtenido selenoproteínas o homólogos. Para obtener los alineamientos realizados mediante multiTCOFFEE.sh hemos usado el programa Jalview, que permite visualizar los alineamientos de manera intuitiva, con información adicional.
dsbA
Con estos alineamientos múltiples de las distintas queries de dsbA, podemos ver que todos los protistas de 2011 para los que hemos obtenido Tcoffee alinean gran parte de la proteína.
SelI
Con los alineamientos múltiples de las distintas queries de SelI podemos ver que los protistas de 2011 para los que hemos obtenido Tcoffee tan solo alinean el inicio la proteína y no la región en la que se encuentra la U (20 últimos nucleótidos de las queries). Esta región inicial es la que presenta homología con la enzima CDP-alcohol phosphatidiltransferase. Con esto remarcamos que los protistas carecen de SelI aunque conservan la CDP-alcohol phosphatidiltransferase.