Selenoproteïnes en Plasmodium vivax
Clara Conill, Estel Enreig, Albert Gil, Carlos González, Jordina Guillén
Facultat de Ciències de la Salut i de la Vida
Universitat Pompeu Fabra
Aquesta és una proteïna associada a tRNA Sec. Va ser aïllada per immunoprecipitació en mostres de pacients amb hepatitis crònica d'origen autoimmune. Recentment, s'ha relacionat amb SECp43 i s'ha proposat que aquestes dues proteïnes juguen un paper molt important en el procés general de biosíntesi i incorporació de selenocisteïnes.
RECERCA DE SLA/LP EN Plasmodium vivax
Amb la seqüència aminoacídica de SLA/LP de Drosophila melanogaster realitzàrem un TBLASTN contra el genoma de Plasmodium vivax:
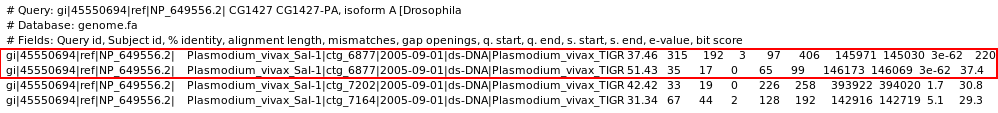 |
| Figura 1. Resultats TBLASTN |
| Descarrega't l'arxiu |
Els resultats obtinguts del TBLASTN ens serviren per a confirmar que en el genoma del nostre organisme existeixen seqüències homòlogues a aquesta proteïna. A més a més, ens van indicar a quina posició del genoma es troba aquesta homologia significativa. Així, vam saber que en el ctg_6877 trobem aquesta seqüència homòloga.
A continuació realitzàrem un GeneWise amb la proteïna SLA/LP de Drosophila melanogaster contra el fragment del ctg on el TBLASTN ens va predir el gen que codifica per a la proteïna homòloga a la de Drosophila melanogaster. El programa ens va predir l'estructura del gen contingut en aquesta seqüència nucleotídica, així com la seqüència aminoacídica de la proteïna a la que donaria lloc. (resultats aquí)
Per tal de comprovar que la proteïna predita pel GeneWise existeix veritablement en el genoma de Plasmodium vivax vàrem fer un TBLASTN de la predicció (malgrat no es tracti de la proteïna sencera) contra totes les seqüències de mRNA de la base de dades d'aquest organisme. D'aquesta manera, esperàvem veure uns alineaments pràcticament perfectes entre la nostra predicció i algun transcrit del genoma de Plasmodium vivax.
Seqüència predita pel GeneWise:
HNEHSHKCMQLRVIRRGNGLPLLRHATLPPPLLSILNYGRVPNEGLNEITIMSILHQISS QNLCNSEKNVKIGERENRIYSALVRNKYIGFGHGIGRSGNLDDVQPKSAGNSVLAKATTS FVKDLIKSFGIKGCEDVYILPYATGMCLSTCILYTKKEREKSEYVIVSRIDHKTCYKCID FCALKYLVVDMVYRDEELHTNLSEIEKLIQTYGEKICCVMSVTSSYAPRNSDDIVKIGHM CRRYNIPHIINNAFGLQCNYLCKEIQKCFETKGRVDFVVQSCDKNFLVPVNGGIVFSSDK KKMKELKKHYPGRTPVHAYLDLFITLLELGKRKILNLRKEREENFAWLKNKVSTLCSKYN LSLIKASKNKISMAINLNELYKIYHVENPRSITLLGSLLFYRNVTGHRVICSPLLIRNGG VDPNGVDTTEQGEPKPNGVSFPNPRNS
Seqüència aminoacídica del mRNA:
MNTPGVDVRNGKAVQGDICSNRYSLISKQTLNQKENTLWNILNYGRVPNEGLNEITIMSILHQI SSQNLCNSEKNVKIGERENRIYSALVRNKYIGFGHGIGRSGNLDDVQPKSAGNSVLAKATTSFV KDLIKSFGIKGCEDVYILPYATGMCLSTCILYTKKEREKSEYVIVSRIDHKTCYKCIDFCALKY LVVDMVYRDEELHTNLSEIEKLIQTYGEKICCVMSVTSSYAPRNSDDIVKIGHMCRRYNIPHII NNAFGLQCNYLCKEIQKCFETKGRVDFVVQSCDKNFLVPVNGGIVFSSDKKKMKELKKHYPGRT PVHAYLDLFITLLELGKRKILNLRKEREENFAWLKNKVSTLCSKYNLSLIKASKNKISMAINLN ELYKIYHVENPRSITLLGSLLFYRNVTGHRVICSPLLIRNGGVDPNGVDTTEQGEPKPNGVSFP NPRNSSEDARPTHSPNNSNQGGVKETSQTHLVKNENATHAAILGKGLTIGNHTFEHFGCSYDLY PFSYIAFSCVIGIEREELQSFVEKLDDAIGCFIRRFGRRASPVERSYAPL
Prenent la seqüència del mRNA completa amb la qual vam obtenir un alineament pràcticament perfecte, poguérem obtenir la seqüència real de la nostra proteïna. Com es pot observar, no vam poder predir la totalitat de la proteïna. Però amb l'ajuda del programa Exonerate deduírem de manera completa l'estructura del nostre gen a la seqüència genòmica.
Com a resum de l'estructura del gen:
# seqname source feature start end score strand frame attributes # Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome gene 144592 146786 2880 - .
gene_id 1 ; sequence ; gene_orientation + Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome cds 146700 146786 . - . Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome exon 146700 146786 . - .
insertions 0 ; deletions 0 Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome splice5 146698 146699 . - .
intron_id 1 ; splice_site "GT" Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome intron 146523 146699 . - .
intron_id 1 Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome splice3 146523 146524 . - .
intron_id 0 ; splice_site "AG" Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome cds 146491 146522 . - . Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome exon 146491 146522 . - .
insertions 0 ; deletions 0 Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome splice5 146489 146490 . - .
intron_id 2 ; splice_site "GT" Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome intron 146370 146490 . - .
intron_id 2 Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome splice3 146370 146371 . - .
intron_id 1 ; splice_site "AG" Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome cds 146283 146369 . - . Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome exon 146283 146369 . - .
insertions 0 ; deletions 0 Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome splice5 146281 146282 . - .
intron_id 3 ; splice_site "GT" Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome intron 146178 146282 . - .
intron_id 3 Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome splice3 146178 146179 . - .
intron_id 2 ; splice_site "AG" Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome cds 146076 146177 . - . Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome exon 146076 146177 . - .
insertions 0 ; deletions 0 Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome splice5 146074 146075 . - .
intron_id 4 ; splice_site "GT" Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome intron 145970 146075 . - .
intron_id 4 Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome splice3 145970 145971 . - .
intron_id 3 ; splice_site "AG" Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome cds 144592 145969 . - . Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome exon 144592 145969 . -
. insertions 0 ; deletions 0 Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome similarity 144592 146786 2880
| Figura 2. Resultats Exonerate (.gff) |
Per últim, per tal de determinar si aquesta proteïna conserva la seva funcionalitat, vam utilitzar el programa InterProScan, el qual localitza motius característics funcionals que es poden trobar a la seqüència aminoacídica de la nostra proteína.
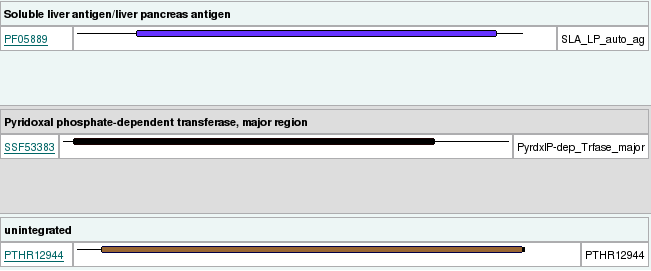 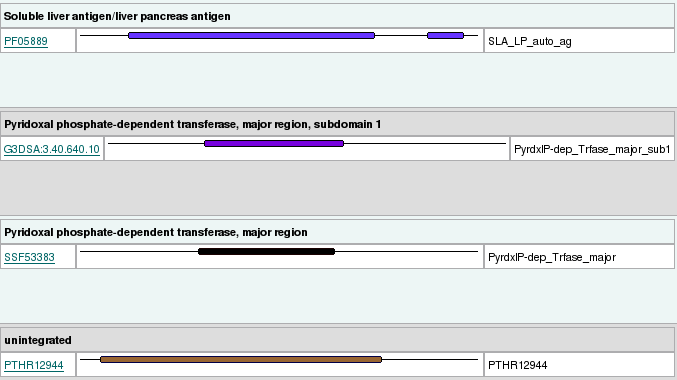 |
| Figura 3. Dominis SLA/LP en D. melanogaster (esquerra) i en P.vivax (dreta) |
Observem que la proteïna estudiada conserva la funcionalitat respecte a la seva homòloga en Drosophila melanogaster, la qual cosa és concloent de que hem aconseguit trobar veritablement la proteïna homòloga a SLA/LP funcional al nostre organisme.
Per resumir tota aquesta informació aquí podem veure un esquema de la SLA/LP de P.vivax:
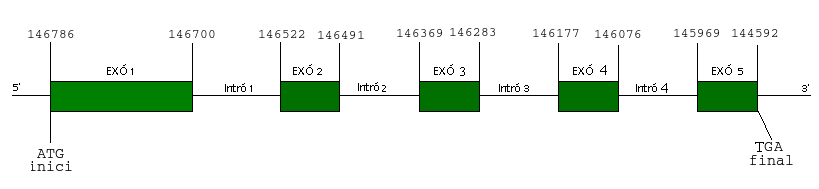 |
| Figura 4. Esquema SLA/LP |