
Daniela Arias Lázaro (ladaniela@gmail.com) y Ana Nadal Sard (aina.aineta@gmail.com)
Facultad de Ciencias de la Salud y de la Vida. Universidad Pompeu Fabra

En este trabajo presentamos la caracterización de la estructura exónica, homología, función y expresión de la proteína de fusión FIP1L1/PDGFRA. Esta proteína se forma por la fusión de FIP1L1, factor involucrado en el procesamiento del mRNA, y PDGFRA, receptor alfa de factores de crecimiento derivados de plaquetas. Ambos genes se encuentran en el cromosoma 4q12 y será la delección de alrededor de 800pb la que posibilite su fusión. Se ha demostrado que en pacientes con síndrome de hipereosinofilia idiopática se produce la fusión de estas dos proteínas, y que ello conlleva la activación constitutiva de una tirosin-kinasa que transforma células hematopoyéticas. Esta patología se caracteriza por la sobreexpresión de eosinófilos en la médula ósea, infiltración y daño tisular.
El estudio genómico de la proteína de fusión FIP1L1/PDGFRA se ha realizado a partir de la caracterización de los dos genes involucrados, por separado. A continuación se presentan los resultados obtenidos en el estudio de cada uno de estos genes.
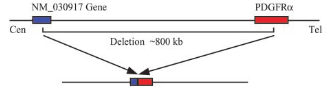
Figura 1. Formació de la proteína de fusión FIP1L1/PDGFRA por la delección de 800pb.
Respecto al gen FIP1L1 hemos encontrado que existen 4 transcritos diferentes, que codifican para 4 isoformas. En la figura 1, podemos observar la estructura genómica de los transcritos del gen FIP1L1 y, a continuación presentamos una tabla en la que describimos cada uno de estos transcritos.
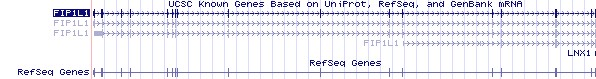
Figura 2. Estructura genómica de los transcritos del gen FIP1L1.
| TRANSCRITO 1 | TRANSCRITO 2 | TRANSCRITO 3 | TRANSCRITO 4 | |
| POSICIÓN (chr4) | 53938620-54020597 | 53938620-54020540 | 53938727-54020540 | 53988990-54021785 |
| CADENA | + | + | + | + |
| TAMAÑO GENÓMICO | 81.978 | 81.921 | 81.814 | 32.796 |
| TAMAÑO mRNA | 2.154 | 1.914 | 3.201 | 2.173 |
| EXONES | 18 | 15 | 15 | 7 |
| EXONES CODIFICANTES | 18 | 15 | 15 | 4 |
| 5' UTR | 143 | 143 | 1328 | 217 |
| 3' UTR | 224 | 167 | 167 | 1333 |
| TAMAÑO PROTEÍNA | 594 | 117 | 117 | 208 |
Tabla 1. Descripción de los transcritos del gen FIP1L1.
El estudio del splicing alternativo se encuentra en el siguiente enlance: ISOFORMAS FIP1L1
Tras realizar un análisis detallado exón por exón y realizar varios alineamientos podemos concluir que la isoforma 1(NM030917) es la escogida como referencia. Observamos que en la isoforma 2 (AL136910) desaparecen los exones 2, 9 y 11; y que en la isoforma 3 (BC024016) desaparecen los exones 2, 3 y 10. En los dos casos se mantienen las pautas de lectura.
Con respecto a la isoforma 4 (AK090938) , desaparecen los primeros exones. Los exones 1 y 2 no son codificantes, por tanto, no repercuten en la pauta de lectura. El inicio del exón 3 coincide con el exón 15 de la isoforma 1, pero deja de coincidir la parte codificante. Los exones 4, 5 y 6 corresponden respectivamente a los exones 16, 17 y 18 de las isoforma 1. Incluso siendo la isoforma 4 la más diferente; en las partes codificantes, igual que en las otras dos isoformas, se mantienen las respectivas pautas de lectura.
Respecto al gen PDGFRA hemos encontrado que existen 2 transcritos diferentes, que codifican para 2 isoformas. En la figura 2, podemos observar la estructura genómica de los transcritos del gen PDGFRA y, a continuación, describimos en forma de tabla cada uno de estos transcritos.

Figura 3. Estructura genómica de los transcritos del gen PDGFRA.
| TRANSCRITO 1 | TRANSCRITO 2 | |
| POSICIÓN (chr4) | 54790204-54859168 | 54790235-54842902 |
| CADENA | + | + |
| TAMAÑO GENÓMICO | 68.965 | 52.668 |
| TAMAÑO mRNA | 6.405 | 3.032 |
| EXONES | 23 | 18 |
| EXONES CODIFICANTES | 22 | 15 |
| 5' UTR | 148 | 117 |
| 3' UTR | 2.972 | 642 |
| TAMAÑO PROTEÍNA | 1.089 | 743 |
Tabla 2. Descripción de los transcritos del gen FIP1L1.
El estudio del splicing alternativo se encuentra en el siguiente enlance: ISOFORMAS PDGFRA
En el caso de la proteína PDGFRA, la isoforma 1 es la de referencia, y en la isoforma 2 desaparecen los últimos exones (del 18 al 23), esenciales para la fusión con FIP1L1. Se mantienen las pautas de lectura pero no se conserva la posibilidad de fusionarse.
En la siguiente enlace presentamos una tabla con los ortólogos predichos por la base de datos Ensembl. En color gris se muestran aquellos ortólogos que presentan una homología más alta. Esta selección, las imágenes correspondientes y los porcentages de homología (Target) se muestran en la tabla 3.
ORTÓLOGOS de FIP1L1
| NOMBRE | IMAGEN | NOMBRE | IMAGEN |
| Pan troglodytes %ID 100 |
 |
Macaca mulatta %ID 99 |
 |
| Bos taurus %ID 98 |
 |
Rattus norvegicus %ID 95 |
 |
| Mus musculus %ID 95 |
 |
Canis familiaris %ID 92 |
 |
| Monodelphis domestica %ID 90 |
 |
En la siguiente enlace presentamos una tabla con los ortólogos del gen PDGFRA. En color gris se muestran aquellos ortólogos que presentan una homología más alta. Esta selección, las imágenes correspondientes y los porcentages de homología (Target) se muestran en la tabla 4.
En este caso no hemos utilizado la base de datos Ensembl, sino que hemos realizado un blastp a partir de la base NCBI. Las razones que justifican los diferentes métodos de obtención de ortólogos se presentan en el apartado de métodos.
ORTÓLOGOS de PDGFRA
| NOMBRE | IMAGEN | NOMBRE | IMAGEN |
| Mus musculus %ID 93 |
 |
Macaca mulatta %ID 99 |
 |
| Bos taurus %ID 95 |
 |
Rattus norvegicus %ID 94 |
 |
| Canis familiaris %ID 97 |
 |
Hemos encontrado diferentes patrones de expresión para el gen FIP1L1 y para la PDGFRA. Ambos son presentados a continuación.
| TEJIDO | EXPRESIÓN | TEJIDO | EXPRESIÓN |
| Endotelio | ELEVADA | Mieloide | ELEVADA |
| Células dendríticas | ELEVADA | Células T: CD4+ y CD8+ | ELEVADA |
| Células NK | ELEVADA | Linfoblastos | ELEVADA |
| Linfoma Burkitts Daudi | ELEVADA | Leucemia linfoblástica | ELEVADA |
| Cerebelo | DISMINUÍDA | Médula oblongata | DISMINUÍDA |
| Ganglio cervical superior | DISMINUÍDA | Apéndice | DISMINUÍDA |
| Glándula adrenal | DISMINUÍDA | Glándula salival | DISMINUÍDA |
| Hígado | DISMINUÍDA | Riñón | DISMINUÍDA |
Tabla 5. Perfil de expresión del gen PDGFRA en GNF Expression Atlas 2 Data from U133A and GNF1H Chips.
En el siguiente enlace se muestran los microarrays, a partir de los cuales se ha elaborado la anterior tabla: microarrays de la expresión de FIP1L1
| TEJIDO | EXPRESIÓN | TEJIDO | EXPRESIÓN |
| Adipocitos | ELEVADA | Músculo liso | ELEVADA |
| Ovario | ELEVADA | Células T: CD4+ y CD8+ | DISMINUÍDA |
| Células NK | DISMINUÍDA | Linfoblastos | DISMINUÍDA |
| Monocitos | DISMINUÍDA | Células dendríticas | DISMINUÍDA |
| Células B | DISMINUÍDA | Ganglio cervical superior | DISMINUÍDA |
| Adenocarcinoma colorrectal | DISMINUÍDA | Leucemia mielógena crCónica | DISMINUÍDA |
Tabla 6. Perfil de expresión del gen PDGFRA en GNF Expression Atlas 2 Data from U133A and GNF1H Chips.
En el siguiente enlace se muestran los microarrays, a partir de los cuales se ha elaborado la anterior tabla: microarrays de la expresión de PDGFRA
A continuación, presentamos en este enlace los GOs asociados a cada uno de los genes.
Para la caracterización de las regiones promotoras se han utilizados dos métodos diferentes. En primer lugar presentamos los resultados obtenidos a partir de la base de datos PROMO y posteriormente, presentamos nuestro programa Perl, junto con los resultados que hemos obtenido. En los siguientes enlaces se presentan las listas de factores de transcripción seleccionados, así como la información que nos ha permitido determinar que eran los que presentaban una mayor probabilidad de unirse con la región promotora de FIP1L1 y PDGFRA, respectivamente.
Factores de transcripción para FIP1L1
Factores de transcripción para PDGFRA
Por otro lado, hemos desarrollado un programa en Perl con el objetivo de determinar que factores de transcipción se unen a cada una de las secuencias promotoras. Para ello, determinaremos que factores tienen menor probabilidad de rechazar erróneamente la hipótesis inicial de que el factor no se une.
Tras ejecutar el programa con la secuencia promotora de PDGFRA, obtenemos los resultados que se presentan en el siguiente enalce:
De estos resultados podemos extraer que el mejor p-value lo presenta el factor de transcripción AP-1 [T00029], con un valor de 0,07. Al ser el valor más pequeño nos indica que tiene menos probabilidad de unirse al azar a la secuencia promotora de nuestro gen.
En el siguiente enlace presentamos una tabla con los factores de transcripción que presentan menores p-value.
FACTORES DE TRANSCRIPCIÓN PDGFRA
Al ejecutar el programa con la secuencia promotora de FIP1L1, obtenemos los resultados que se presentan en el siguiente enalce:
De estos resultados podemos extraer que el mejor p-value lo presenta el factor de transcripción HNF-4 [T02758], con un valor de 0,16. Al ser el valor más pequeño nos indica que tiene menos probabilidad de unirse al azar a la secuencia promotora de nuestro gen.
En el siguiente enlace presentamos una tabla con los factores de transcripción que presentan menores p-value.
FACTORES DE TRANSCRIPCIÓN FIP1L1.
El gen PDGFRA codifica para un receptor tirosin-kinasa. Pertenece a la gran familia de los factores de crecimiento derivados de plaquetas. Estos factores de crecimiento son mitógenos de células con origen mesenquimal. La unión del factor de crecimiento al receptor determina la estructura del receptor,homo o heterodímero, compuesto por los polipéptidos PDGFR alpha y beta.
A partir de ratones heterocigotos se vió que la forma alpha de PDGFR era particularmente importante para el desarrollo de los riñones. En cambio, estudios realizados en ratones, demuestran que la homocigosidad de este gen es letal.
Respecto a FIP1L1, sabemos que esta involucrado en poliadenilación. En levadura, la proteína Fip1 (de unos 40 amino ácidos de longitud) es un componente del factor de poliadenilación pre-mRNA, que interacciona de forma directa con la polimerasa poly(A). A partir de aquí se vió que era necesario para el procesamiento; poliadenilación del precursor del pre-mRNA. Forma parte del complejo CPSF (Cleavage and polyadenilation specifity factor), encargado de reconocer y procesar el extremo 3'.
Nuestra proteína de fusión fue descubierta a través de estudio de varios pacientes que presentaban el síndrome hipereosinofílico idiopático (HES). Se demostró que este síndrome estaba causado por una deleción en el cromosoma 4q12, que resulta en la fusión de PDGFRA y un gen vecino, FIP1L1. El gen PDGFRA-FIP1L1, es una tirosina kinasa constitutivamente fosforilada y activa, capaz de transformar células hematopoyéticas.
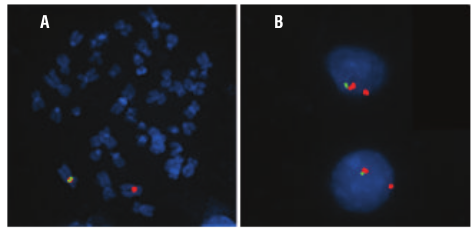
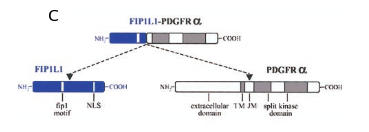
Figura 4. (A-B) FISH de doble color con los clones RP11-3H2O (verde) y RP11-120K16 (rojo). La señal verde/roja indica un cromosoma 4, normal, mientras que la señal únicamente roja muestra la delección del otro cromosoma 4, que provoca la fusión de los dos genes. (C) Esquema de la fusión de la proteína FIP1L1/PDGFRA.
El síndrome hipereosinofílico es una enfermedad hematológica muy rara que se caracteriza por una sobreproducción de eosinófilos en el hueso medular, eosinofília, y daño de órganos. Se trata de una enfermedad mieloproliferativa con una etiologia por ahora desconocida. En estudios clínicos de HES, se han constatado diferentes variantes de la proteína de fusión y la continua disrupción del dominio juxtamembrana autoinhibitorio (JM) en PDGFRA, que parece ser esencial para la correcta activación de la proteína.
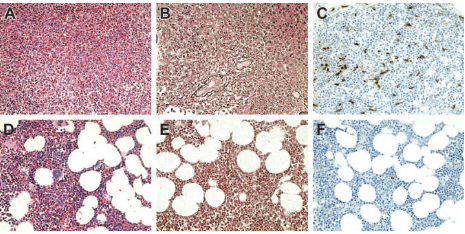
Figura 5. Tinción histoló... Las imagenes de la parte superior muestran hueso medular enfermo, mientras que las de la región inferior muestran hueso medular tratado. Observamos al compararlas una gran diferencia en cuanto a volumen de eosinófilos.
Respecto a la prevalencia, se ha visto que es un síndrome más común en hombres que mujeres, y suele presentarse entre los 20 y los 50 años de edad. El diagnóstico más frecuente se basa en la búsqueda de anormalidades en los cariotipos y en ensayos de inactivación del cromosoma X, y como terapia prometedora tenemos; Imatinib. Este fármaco es un antitumoral capaz de inhibir de forma aberrante las tirosin-kinasas constitutivamente activas. Esta terapia podría ser eficaz para algunos pacientes con HES pero se necesitan muchos estudios previos.
Con el fin de saber a que proteína corresponde la secuencia que se nos ha adjudicado, realizamos un blastp en el que se alinea nuestra secuencia contra todas las proteínas de la base de datos del NCBI. De los resultados obtenidos con un BLOOM 80, hemos seleccionado aquella proteína que presenta un 100% de identidad:
Derived growth factor receptor alpha fusion protein; Rhe-PDGFRA [Homo sapiens]; Length=849; Score = 1853 bits (4287), Expect = 0.0, Method: Composition-based stats.Identities = 849/849 (100%), Positives = 849/849 (100%), Gaps = 0/849 (0%)
En el siguiente enlace presentamos el blastp.
Como podemos ver se trata de una proteína de fusión, que está formada por un total de 878 aminoácidos. De estos, 340 corresponen a la proteína FIP1L1, y 509 aminoácidos a la PDGFRA.Ésta se forma a partir de la delección de unas 800pb y la posterior fusión de dos proteínas FIP1L1 y PDGFRA. Ambas proteínas se encuentran en el cromosoma 4q12.
En la siguientes tabla presentamos las isoformas que permiten la fusión de las dos proteínas.
| FIP1L1: Isoforma 1 (594aa) | PDGFRA: Isoforma 1 (1089aa) |
MSAGEVERLVSELSGGTGGDEEEEWLYGGPWDVHVHSDLAKDLDENEVERPEEENASANP PSGIEDETAENGVPKPKVTETEDDSDSDSDDDEDDVHVTIGDIKTGAPQYGSYGTAPVNL NIKTGGRVYGTTGTKVKGVDLDAPGSINGVPLLEVDLDSFEDKPWRKPGADLSDYFNYGF NEDTWKAYCEKQKRIRMGLEVIPVTSTTNKITAEDCTMEVTPGAEIQDGRFNLFKVQQGR TGNSEKETALPSTKAEFTSPPSLFKTGLPPSRNSTSSQSQTSTASRKANSSVGKWQDRYG RAESPDLRRLPGAIDVIGQTITISRVEGRRRANENSNIQVLSERSATEVDNNFSKPPPFF PPGAPPTHLPPPPFLPPPPTVSTAPPLIPPPGFPPPPGAPPPSLIPTIESGHSSGYDSRS ARAFPYGNVAFPHLPGSAPSWPSLVDTSKQWDYYARREKDRDRERDRDRERDRDRDRERE RTRERERERDHSPTPSVFNSDEERYRYREYAERGYERHRASREKEERHRERRHREKEETR HKSSRSNSRRRHESEEGDSHRRHKHKKSKRSKEGKEAGSEPAPEQESTEATPAE |
MGTSHPAFLVLGCLLTGLSLILCQLSLPSILPNENEKVVQLNSSFSLRCFGESEVSWQYP MSEEESSDVEIRNEENNSGLFVTVLEVSSASAAHTGLYTCYYNHTQTEENELEGRHIYIY VPDPDVAFVPLGMTDYLVIVEDDDSAIIPCRTTDPETPVTLHNSEGVVPASYDSRQGFNG TFTVGPYICEATVKGKKFQTIPFNVYALKATSELDLEMEALKTVYKSGETIVVTCAVFNN EVVDLQWTYPGEVKGKGITMLEEIKVPSIKLVYTLTVPEATVKDSGDYECAARQATREVK EMKKVTISVHEKGFIEIKPTFSQLEAVNLHEVKHFVVEVRAYPPPRISWLKNNLTLIENL TEITTDVEKIQEIRYRSKLKLIRAKEEDSGHYTIVAQNEDAVKSYTFELLTQVPSSILDL VDDHHGSTGGQTVRCTAEGTPLPDIEWMICKDIKKCNNETSWTILANNVSNIITEIHSRD RSTVEGRVTFAKVEETIAVRCLAKNLLGAENRELKLVAPTLRSELTVAAAVLVLLVIVII SLIVLVVIWKQKPRYEIRWRVIESISPDGHEYIYVDPMQLPYDSRWEFPRDGLVLGRVLG SGAFGKVVEGTAYGLSRSQPVMKVAVKMLKPTARSSEKQALMSELKIMTHLGPHLNIVNL LGACTKSGPIYIITEYCFYGDLVNYLHKNRDSFLSHHPEKPKKELDIFGLNPADESTRSY VILSFENNGDYMDMKQADTTQYVPMLERKEVSKYSDIQRSLYDRPASYKKKSMLDSEVKN LLSDDNSEGLTLLDLLSFTYQVARGMEFLASKNCVHRDLAARNVLLAQGKIVKICDFGLA RDIMHDSNYVSKGSTFLPVKWMAPESIFDNLYTTLSDVWSYGILLWEIFSLGGTPYPGMM VDSTFYNKIKSGYRMAKPDHATSEVYEIMVKCWNSEPEKRPSFYHLSEIVENLLPGQYKK SYEKIHLDFLKSDHPAVARMRVDSDNAYIGVTYKNEEDKLKDWEGGLDEQRLSADSGYII PLPDIDPVPEEEDLGKRNRHSSQTSEESAIETGSSSSTFIKREDETIEDIDMMDDIGIDS SDLVEDSFL |
Posteriormente, hemos analizado la estructura genómica de las dos proteínas por separado. Para ello, hemos recurrido a la base de datos UCSC Genome Browser. Ésta nos indicaba el número de exones, codificantes o no codificantes, y la información necesaria para analizar las diferentes isoformas de cada proteína (ver Estructura genómica).
A continuación, hemos analizado la homología que presenta cada uno de los genes con otras especies. Para ello, hemos utilizado la base de datos Ensembl. Introduciendo el gen FIP1L1 obtuvimos una lista de predicción de ortólogos con los correspondientes porcentages de identidad query y target (ver ORTÓLOGOS de FIP1L1). En cambio, para el gen de la PDGFRA no encontramos en Ensemblinformación sobre los ortólogos (Hace 15 días sí que estaban. El hecho que hayan desaparecido nos permite suponer que estas predicciones no eran correctas). Por ello, hemos realizado un blastp de la PDGFRA contra todas las proteínas (de todas las especies) del NCBI. A partir de los resultados obtenidos, hemos seleccionado aquellas secuencias que presentan un e-valor más bajo y que el porcentaje de homología es superior. (ver ORTÓLOGOS de PDGFRA ).
Para el análisis de la expresión de los genes, hemos seleccionado la base de datos UCSC Genome Browser. En esta, al introducir cada uno de los genes obtenemos los resultados de diferentes microarrays. El análisis de estos se muestra en las tablas 5 y 6, respectivamente, en las que hemos indicado que tejidos presentan una expresión del gen superior o inferior a la basal. (ver Estudio de la expresión)
Para el estudio de la región promotora y de los factores de transcripción que la regulan hemos seguido dos líneas de trabajo. Por un lado, a partir de la base de datos UCSCGenome Browser, hemos obtenido la secuencia promotora de cada uno de los genes ( FIP1L1 y PDGFRA). Esta secuencia la hemos introducido en el PROMO, y seleccionando un valor máximo de disparidad de 10 hemos obtenido los posibles factores de transcripción que se podrían unir a cada una de las secuencias promotoras. Posteriormente, hemos extraído la información de cada uno de estos factores y con la ayuda de procesadores de texto y Excel hemos ordenado según su valor de RE equally y query. El criterio de selección utilizado es a menor RE equally y menor disparidad, mayor probabilidad de unión a la secuencia promotora. De esta forma, hemos podido seleccionar los más significativos: 13 de 411 factores de transcripción para PDGFRA y, 8 de 337 para FIP1L1(ver Factores de transcripción para FIP1L1 y Factores de transcripción para PDGFRA)
Por otro lado, hemos desarrollado un programa en Perl con el objetivo de determinar que factores de transcipción tienen menor probabilidad de rechazar erróneamente la hipótesis inicial de que el factor no se une. A este programa se le dan dos ficheros: el primero, contiene matrices de factores de transcripción y el segundo, contiene una secuencia, en formato FASTA, de la región promotora del gen.
Para ello, hemos desarrollado un algorismo en el cual; primero hemos introducido las matrices de forma que se almacenen en memoria como un hash de vectores. Luego hemos transformado cada matriz de ocurrencias en una matriz de pesos, de forma que obtenemos una matriz dónde cada nucleótido tiene asociado un peso en cada posición diferente del motivo. Para cada factor de transcripción hemos calculado la puntuación (score) que proporciona en cada posición de unión del FT posible dentro de la secuencia.
Finalmente, para cada FT, hemos estimado la frecuencia con la que se observa al azar la puntuación máxima anterior. Se trata de una aproximación a la probabilidad de rechazar erróneamente la hipótesis inicial de que el factor no se une (p-value).
Partimos sabiendo que valores pequeños son indicativos de que la unión que identificamos es difícil de observar al azar, y por tanto parece razonable pensar que este FT se une efectivamente a la región promotora. (ver Estudio de la región promotora).
El algorismo lo podemos encontrar en el siguiente enlace: PROGRAMA
Para el estudio de la función del gen, nos hemos basado principalmente en información del NCBI, puesto que encontramos numeros artículos sobre la proteína de fusión FIP1L1/PDGFRA, función y patologías asociadas. (ver Referencias).
Tras el estudio genómico computacional de nuestra proteína hemos obtenido las siguientes conclusiones.
Nuestra secuencia corresponde a una proteína de fusión: FIP1L1/PDGFRA. Esta proteína se forma tras la delección intersticial de 800pb del cromosoma 4 y la posterior unión de una región del gen FIP1L1 y otra del gen PDGFRA. Ambas regiones corresponden a la parte final de cada gen. En cuanto a la funión de estos genes, decir que FIP1L1 está relacionado con el procesamiento del mRNA, mientras que PDGFRA es un receptor tirosin kinasa de factores de crecimiento derivados de plaquetas.
El resultado de la fusión de estos genes conlleva la activación constitutiva de una tirosin kinasa, que es responsable de la transformación de células hematopoyéticas. Esta alteración da lugar a una enfermedad poco frecuente, conocida como síndrome hipereosinófilo idiopático.
Respecto a la estructura genómica, cabe destacar que ambos genes tienen diferentes transcritos, resultado del splicing alternativo, que codificarán para diferentes isoformas. La FIP1L1 presenta 4 isoformas y la PDGFRA presenta 2 isoformas. Además, debemos señalar que tan solo la isoforma 1 de la PDGFRA será capaz de dar lugar a la proteína de fusión, ya que la isoforma 2 ha perdido los exones involucrados en la fusión.
Del estudio de homología, hemos podido concluir que ambos genes presentan una gran conservación en aquellas especies evolutivamente más cercanas al humano. Este hecho nos permite confirmar que se trata de genes de gran importancia, en los que la selección natural ha ejercido su presión.
En cuanto al estudio de la expresión, hemos observado diferencias. Mientras que la expresión del gen FIP1L1 es elevada en células del sistema inmune y algunos cánceres, la del gen PDGFRA es elevada en tejido adiposo, ovarios y músculo liso.
Finalmente, respecto al estudio de las regiones promotoras cabe decir que al no estar regulados el uno por el otro, cada uno de los genes tendrá sus propios factores de transcripción. A partir de los diferentes criterios de selección, tanto con el programa en Perl como con la base de datos PROMO, podemos concluir que los resultados obtenidos son significativos, es decir, que los factores de transcripción no se unirán al azar.
Páginas web
Clustalw: http://www.ebi.ac.uk/clustalw/
NCBI: http://www.ncbi.nih.gov/
Ensembl: http://www.ensembl.org/
Biomart: http://www.ensembl.org/biomart/martview/be6035946b855fcf976e76171b44e275/
UCSC: http://genome.ucsc.edu/
Uniprot: http://www.ebi.uniprot.org/index.shtml/