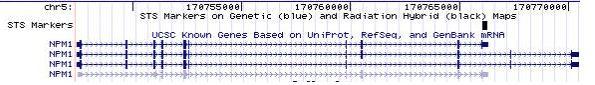
Figura 1. Estructura genòmica dels transcrits del gen NPM1.
Human nucleophosmin-anaplastic lymphoma kinase fusion protein
Cristina Gómez Castellà (cristina.gomez02@campus.upf.edu) i Anna González Tendero (ana.gonzalez04@campus.upf.edu)
Facultat de Ciències de la Salut i de la Vida
Universitat Pompeu Fabra
Quan es produeix una translocació t(2;5)(p23;q35), es produeix la
fusió de porcions dels gens codificants per la proteïna NPM provinent
del cromosoma 5 i la proteïna ALK provinent del cromosoma 2; de forma
que el resultat és una proteïna de fusió causant d'una malaltia
anomenada Non-Hodgkin's limfoma.
La proteïna de fusió conté la seqüència aminoterminal de NPM unida al domini catalític de ALK. NPM es una fosfoproteïna nucleolar, ALK és un receptor protein-tirosin-quinasa
transmembrana.
S'ha observat que 1/3 d'aquests limfomes presenten aquesta
alteració t(2;5)(p23;q35); per tant la translocació d'aquests
protoncogens cel·lulars podria contribuir a la formació de limfomes.
Els limfomes en els que hi és present la translocació, involucren nodes
limfàtics, pell, pulmons, teixits tous, ossos, tracte gastrointestinal
i emergeixen predominantment dels limfòcits T activats. Les cèl·lules
malignes resultants d'aquest procés tumoral expressen el receptor
d'interleucina (IL-2), l'antigen de CD30 i un receptor per un lligand
relacionat amb els factors de necrosis tumorals.
Mitjançant experiments de FISH, PCR, Northern blot i d'altres s'ha
determinat quin fragment del gen de fusió prové del gen codificant per
NPM i quin fragment del gen prové del gen codificant per ALK.
Realitzant experiments de Northern blot amb una sonda per l'extrem 5'
de NPM, es va veure que línies cel·lulars positives per la translocació t(2;5) expresen els transcrits normals i a més un transcrit anormal de
2'4kb, mentre que les línies cel·lulars negatives per t(2;5) només
expressen els transcrits normals. En canvi, amb una sonda per l'extrem
3' de NPM, només es detectaven els transcrits normals en totes les
línies cel·lulars. D'altra banda, amb una sonda per l'extrem 3' del
cDNA fusionat, sí es detectava el transcrit anormal de 2'4kb. Es va
veure que la seqüència 5' dels transcrits anormals de 2'4kb era
idèntica a la seqüència de NPM, però comença a variar a partir de la Valina en posició 117. La seqüència 3' d'aquest codó estava reemplaçadaper 1223 nucleòtids, resultant en una ORF de 1575 nucleòtids.
La seqüència 3' del cDNA del gen de fusió és idèntica a la seqüència 3' d'ALK, que correspon només al seu domini catalític. El transcrit de 2'4kb que dóna lloc a la proteïna quimèrica prové del cromosoma 5 translocat, no hi ha evidència de que el transcrit quimèric s'hagi pogut generar del cromosoma 2.
Es postula que en la proteïna de fusió NPM/ALK, NPM constitueix
un promotor actiu per dirigir l'expressió del domini catalític d'ALK en
les cèl·lules limfoides que presenten la translocació t(2;5). Aquest
fet podria ser crucial pel que fa al paper oncogènic de NPM, ja que el
promotor d'ALK normalment es troba silent en aquestes cèl·lules.
El paper oncogènic de la proteïna ALK podria trobar-se en el fet
de que la majoria d'oncogens tirosina quinasa, com l'ALK, quan pateixen
mutacions o fusions gèniques, porten a una activitat catalítica
constitutiva. Segons això, en la proteïna de fusió NPM/ALK, es podria
predir que l'ALK truncada es disregula i fosforila substrats
intracel·lulars donant lloc a l'activació de la transformació maligna.
En la realització d'aquest treball hem cercat la informació en les
bases de dades disponibles que aporten dades sobre gens coneguts. Hem
estudiat la proteïna de fusió NPM/ALK, formada per parts de dos gens
(NPM i ALK) fusionats a causa d'una translocació entre els
cromosomes 2 i 5.
Com que en les bases de dades no trobem la informació de la
proteïna fusionada, hem estudiat els gens NPM i ALK de forma separada.
A partir de la seqüència proteica donada, hem determinat quina proteïna i quin gen era realitzant un BLASTp.
Primerament ens hem basat en la base de dades de Ensembl per
caracteritzar l'estructura genòmica dels dos gens. Per tal de
complementar i confirmar la informació, hem cercat en altres bases de
dades com UCSC, RefSeq, NCBI, Biomart. Hem utilitzat varies bases de
dades degut a que la informació d'una base de dades a una altra pot
variar.
Per tal d'estudiar l'homologia dels gens en altres espècies, per
un dels gens, Ensembl ens proporcionava una llista d'espècies en les
que hi trobem gens ortòlegs. Per l'altre gen, no ens proporcionava cap
ortòleg, per aquest motiu hem utilitzat les altres bases de dades abans
anomenades. A més a més, per confirmar els ortòlegs i trobar més
possibles ortòlegs, hem realitzat Blast. Amb aquesta eina hem complementat la llista.
Amb l'objectiu d'obtenir més dades sobre l'homogia dels gens en
altres espècies, hem realitzat un alineament entre totes le seqüències
amb el clustalw, amb el qual també hem obtingut un arbre filogenètic.
Altres tipus d'informació com l'expressió, la funció, malalties,
etc, ha estat extreta de les bases de dades abans esmentades i d'altres
com Gene Ontology, OMIM, VisiGene, Pubmed, InterPro.
Finalment hem volgut caracteritzar la regió promotora del gen.
Aquest estudi l'hem fet mitjançant el servidor web del programa PROMO i
mitjançant un programa en Perl que hem desenvolupat nosaltres mateixes.
Seqüència proteica:
MEDSMDMDMSPLRPQNYLFGCELKADKDYHFKVDNDENEHQLSLRTVSLGAGAKDELHIVEAEAMNYEGS PIKVTLATLKMSVQPTVSLGGFEITPPVVLRLKCGSGPVHISGQHLVAVEEDAESEDEEEEDVKLLSISG KRSAPGGGSKVPQKKVKLAADEDDDDDDEEDDDEDDDDDDFDDEEAEEKAPVKKSIRDTPAKNAQKSNQN GKDSKPSSTPRSKGQESFKKQEKTPKTPKGPSSVEDIKAKMQASIEKGGSLPKVEAKFINYVKNCFRMTD QEAIQDLWQWRKSL
Es troba al cromosoma 5q35 (170,746,725-170,770,492).
Associats a aquest gen hi trobem tres transcrits:

Figura 1. Estructura genòmica dels transcrits del gen NPM1.
TCTATACGAGATACTCCAGCCAAAAATGCACAAAAGTCAAATCA GAATGGAAAAGACTCAAAACCATCATCAACACCAAGATCAAAA
SIRDTPAKNAQKSNQNGKDSKPSSTPRSK
Les diferents isoformes són obtingudes degut al procés de splicing
alternatiu. Les diferències entre elles afecten porcions codificants,
tot i això, la pauta de lectura es manté, de manera que les proteïnes
obtingudes tenen la mateixa seqüència d'aminoàcids, excepte les
variacions de llargada de la proteïna degut a la traducció o no d'algun
dels exons.
| Espècie | % Identitat | Espècie | % Identitat |
 Pan troglodytes Pan troglodytes |
100% |  Pongo pygmaeus Pongo pygmaeus |
99% |
 Macaca mulatta Macaca mulatta |
99% |  Canis familiaris Canis familiaris |
98% |
 Bos taurus Bos taurus |
95% |  Mus musculus Mus musculus |
94% |
 Rattus norvegicus Rattus norvegicus |
94% |  Monodelphis domestica Monodelphis domestica |
87% |
La següent taula ens mostra els teixits en els que s'expressa el gen
NPM1. El color negre mostra que no hi ha expressió en aquests teixits;
el color verd indica poca expressió i el color vermell denota molta
expressió.
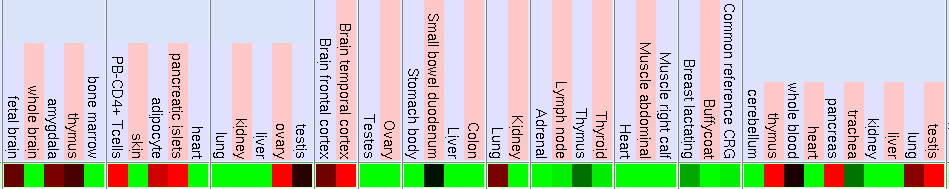
Figura 2. Expressió del gen NPM1.
En aquesta imatge
podem veure la distribució nucleolar de NPM1. La imatge correspon a
un embrió preimplantacional de ratolí visualitzat mitjançant
microsopia immunoelectrònica. S'han utilitzat anticossos anti-NPM1.
Mitjançant la base de dades de Gene Ontology, hem estudiat la funció del gen. La següent taula mostra les anotacions de GO associades a aquest gen, les qual es troben dividides en tres grups: component cel·lular, procés biològic i funció molecular. Els dos últims són els que ens seran més útils per estudiar la funció del gen:
| Gene Ontology | Funció | |
| GO:0003676 | Nucleic acid binding | |
| GO:0003713 | Coactivador de la transcripció | |
| GO:0003723 | RNA binding | |
| GO:0005515 | Protein binding | |
| Funció Molecular | GO:0030957 | Tat protein binding |
| GO:0042803 | Activitat homodimeritzadora | |
| GO:0046982 | Activitat heterodimeritzadora | |
| GO:0051082 | Unfolded protein binding | |
| GO:0006886 | Transport intracel·lular | |
| GO:0006913 | Transport nucleocitoplasmàtic | |
| GO:0006916 | Anti-apoptosi | |
| Procés Biològic | GO:0006950 | Resposta a estrés |
| GO:0007165 | Transducció de senyals | |
| GO:0008285 | Regulació negativa de la proliferació cel·lular | |
| GO:0042255 | Ensemblatge de ribosomes | |
| GO:0005634 | Nucli | |
| Component cel·lular | GO:0005730 | Nucleol |
| GO:0005737 | Citoplasma | |
| GO:0005813 | Centrosoma |
NPM1 és un fosfoproteïna no ribosomal que es troba concentrada a la regió granular del nuclèol. Es troba de forma més abundant en cèl·lules tumorals que en cèl·lules normals. Està involucrada en l'ensamblatge de partícules prerribosomals, de les subunitats petites i de les grans. S'uneix amb gran afinitat a àcids nucleics de cadena única, exercint una activitat que desestabilitza l'hèlix de RNA, i a més es troba en associació amb les ribonucleoproteïnes prerribosomals nucleolars més madures. La transcripció i la traducció de NPM estàn regulades pel cicle cel·lular, assolint els nivells més alts just abans de l'entrada en fase S i amb una davallada abans de l'inici de la fase G2.
NPM ha estat identificada com a substrat de CDK2/ciclina E en la duplicació del centrosoma; s'associa amb els centrosomes no duplicats i es dissocia d'ells mitjançant la fosforilació per part de CDK2/ciclina E.
Pel que fa a la seva genètica molecular, es sap que NPM1 regula la via supressora de tumors ARF/p53, així com també que translocacions cromosòmiques que l'involucren provoquen una deslocalització citoplasmàtica, associada a malalties.
MGAIGLLWLLPLLLSTAAVGSGMGTGQRAGSPAAGPPLQPREPLSYSRLQRKSLAVDFVVPSLFRVYARD LLLPPSSSELKAGRPEARGSLALDCAPLLRLLGPAPGVSWTAGSPAPAEARTLSRVLKGGSVRKLRRAKQ LVLELGEEAILEGCVGPPGEAAVGLLQFNLSELFSWWIRQGEGRLRIRLMPEKKASEVGREGRLSAAIRA SQPRLLFQIFGTGHSSLESPTNMPSPSPDYFTWNLTWIMKDSFPFLSHRSRYGLECSFDFPCELEYSPPL HDLRNQSWSWRRIPSEEASQMDLLDGPGAERSKEMPRGSFLLLNTSADSKHTILSPWMRSSSEHCTLAVS VHRHLQPSGRYIAQLLPHNEAAREILLMPTPGKHGWTVLQGRIGRPDNPFRVALEYISSGNRSLSAVDFF ALKNCSEGTSPGSKMALQSSFTCWNGTVLQLGQACDFHQDCAQGEDESQMCRKLPVGFYCNFEDGFCGWT QGTLSPHTPQWQVRTLKDARFQDHQDHALLLSTTDVPASESATVTSATFPAPIKSSPCELRMSWLIRGVL RGNVSLVLVENKTGKEQGRMVWHVAAYEGLSLWQWMVLPLLDVSDRFWLQMVAWWGQGSRAIVAFDNISI SLDCYLTISGEDKILQNTAPKSRNLFERNPNKELKPGENSPRQTPIFDPTVHWLFTTCGASGPHGPTQAQ CNNAYQNSNLSVEVGSEGPLKGIQIWKVPATDTYSISGYGAAGGKGGKNTMMRSHGVSVLGIFNLEKDDM LYILVGQQGEDACPSTNQLIQKVCIGENNVIEEEIRVNRSVHEWAGGGGGGGGATYVFKMKDGVPVPLII AAGGGGRAYGAKTDTFHPERLENNSSVLGLNGNSGAAGGGGGWNDNTSLLWAGKSLQEGATGGHSCPQAM KKWGWETRGGFGGGGGGCSSGGGGGGYIGGNAASNNDPEMDGEDGVSFISPLGILYTPALKVMEGHGEVN IKHYLNCSHCEVDECHMDPESHKVICFCDHGTVLAEDGVSCIVSPTPEPHLPLSLILSVVTSALVAALVL AFSGIMIVYRRKHQELQAMQMELQSPEYKLSKLRTSTIMTDYNPNYCFAGKTSSISDLKEVPRKNITLIR GLGHGAFGEVYEGQVSGMPNDPSPLQVAVKTLPEVCSEQDELDFLMEALIISKFNHQNIVRCIGVSLQSL PRFILLELMAGGDLKSFLRETRPRPSQPSSLAMLDLLHVARDIACGCQYLEENHFIHRDIAARNCLLTCP GPGRVAKIGDFGMARDIYRASYYRKGGCAMLPVKWMPPEAFMEGIFTSKTDTWSFGVLLWEIFSLGYMPY PSKSNQEVLEFVTSGGRMDPPKNCPGPVYRIMTQCWQHQPEDRPNFAIILERIEYCTQDPDVINTALPIE YGPLVEEEEKVPVRPKDPEGVPPLLVSQQAKREEERSPAAPPPLPTTSSGKAAKKPTAAEISVRVPRGPA VEGGHVNMAFSQSNPPSELHKVHGSRNKPTSLWNPTYGSWFTEKPTKKNNPIAKKEPHDRGNLGLEGSCT VPPNVATGRLPGASLLLEPSSLTANMKEVPLFRLRHFPCGNVNYGYQQQGLPLEAATAPGAGHYEDTILK SKNSMNQPGP
Es troba al cromosoma 2p23 (29,269,594-29,997,029). Associats a aquest gen hi trobem dos transcrits1:

Figura 3. Estructura genòmica dels transcrits del gen ALK.
Si comparem els dos transcrits anteriors podem veure que pesenten el mateix nombre de parells de bases i residus en la proteïna traduïda. La diferencia entre ells es troba en el nombre d'exons.
Tal i com es pot veure en les imatges següents, les quals corresponen a una part de la seqüència dels exons i introns d'aquests dos transcrits, el transcrit 1 presenta un exó més que el transcrit 2; el transcrit 1 presenta un exó de cinc nucleòtids (exó 9) que no apareix en el transcrit 2.
Si ens fixem en les longituds dels exons i els introns dels dos transcrits podem veure que l'exó 8 del primer transcirt té una llargada de 96 nucleòtids, en canvi aquest mateix exó en el segon transcrit té una llargada de 101 nucleòtids.
Si observem els introns, podem veure que en el transcrit 1 l'intró 8-9 té una longitud de 8883 nucleòtids i l'intró 9-10 és de 12363 nucleòtids. En el cas del transcrit 2, l'intró 8-9 té una longitud de 21246 nucleòtids.
Vist això, ens adonem que hi ha una diferència de 5 nucleòtids en l'exó 8 dels transcrits que es correspon amb els 5 nucleòtids de l'exó 9 del transcrit 1 i que la suma de la longitud dels dos introns del primer transcrit es correspon amb la llargada de l'intró 8-9 del segon transcrit.
En fer l'alineament dels dos transcrits veiem que difereixen en un sol nucleòtid, canvia una citosina per una guanina.
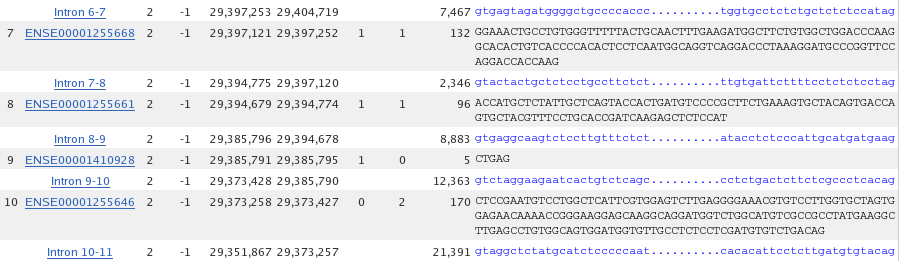
Figura 4. Seqüència parcial del transcrit 1.
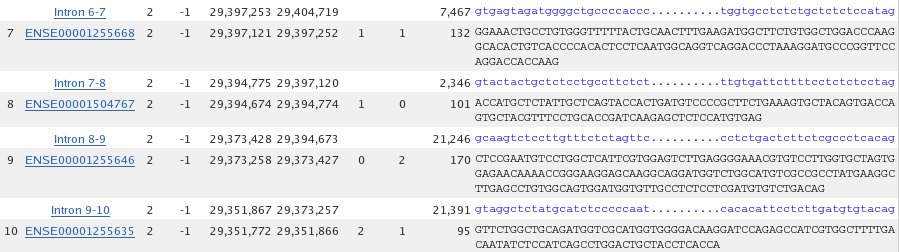
Figura 5. Seqüència parcial del transcrit 2.
Hem trobat els ortòlegs del gen ALK en diferents espècies. La següent
taula mostra les espècies i el % d'identitat amb cada una d'elles. Hem utilitzat un criteri propi per tal de decidir quines espècies considerem que presenten un gen ortòleg. Aquest criteri és agafar només aquelles espècies que presente un percentatge d'identitat superior al 60 %.
| Espècie | % Identitat | Espècie | % Identitat |
 Bos taurus Bos taurus |
77 % |  Canis familiaris Canis familiaris |
70 % |
 Dasypus novemcinctus Dasypus novemcinctus |
68 % |  Echinops telfairi Echinops telfairi |
65 % |
 Felis catus Felis catus |
83 % |  Loxodonta africana Loxodonta africana |
66 % |
 Macaca mulatta Macaca mulatta |
97 % |  Monodelphis domestica Monodelphis domestica |
62 % |
 Mus musculus Mus musculus |
87 % |  Pan troglodytes Pan troglodytes |
87 % |
 Rattus norvegicus Rattus norvegicus |
88 % |  Tupaia belangeri Tupaia belangeri |
74 % |
A partir de l'alineament hem construït un arbre filogenètic en el que observem l'evolució d'aquest gen.
En aquesta taula podem veure l'expressió de la proteïna en diferents
teixits. El color negre mostra que no hi ha expressió en els teixits;
el color verd indica poca expressió i el vermell denota molta
expressió.
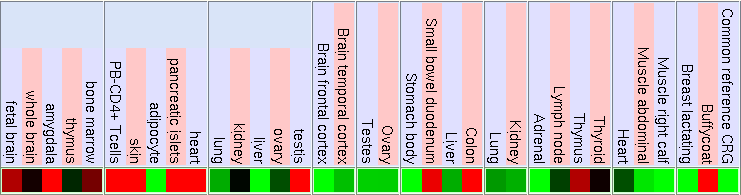
Figura 6. Expressió del gen ALK.
Mitjançant la base de dades de Gene Ontology, hem estudiat la funció del gen. La següent taula mostra les anotacions de GO associades a aquest gen, les qual es troben dividides en tres grups: component cel·lular, procés biològic i funció molecular. Els dos últims són els que ens seran més útils per estudiar la funció del gen:
| Gene Ontology | Funció | |
| GO:0000166 | Unió a nucleòtids | |
| GO:0004672 | Activitat protein-quinasa | |
| GO:0004713 | Activitat protein-tirosin-quinasa | |
| GO:0004714 | Receptor transmembrana amb activitat protein-tirosin-quinasa | |
| Funció Molecular | GO:0004716 | Receptor senyalitzador de proteïna amb activitat tirosin-quinasa |
| GO:0004872 | Receptor | |
| GO:0005524 | ATP binding | |
| GO:0016301 | Activitat quinasa | |
| GO:0016740 | Activitat transferasa | |
| GO:0006468 | Activitat quinasa | |
| Procés Biològic | GO:0007169 | Receptor transmembrana |
| GO:0007399 | Desenvolupament del sistema nerviós | |
| GO:0007420 | Desenvolupament cerebral | |
| Component cel·lular | GO:0016020 | Membrana |
El gen ALK codifica per un receptor transmembrana tirosin-quinasa, la seva expressió fisiològica en mamífers es troba molt limitada a regions específiques del sistema nerviós central i perifèric. La proteïna ALK està involucrada en oncogènesis, tant de tumors hematopoiètics com no hematopoiètics, ja que fusions gèniques d'ALK són presents en tumors miofibroblèstics i limfomes. L'expressió disregulada d'ALK està relacionada amb tumors no limfoides, com sarcomes, neuroblastomes i gliomes.
La regió C-terminal conté el domini catalític, i la regió N-terminal conté dominis de dimerització.
Hem extret la regió promotora del gen 1kb upstream i 100bp downstream del lloc de començament de la transcripció. A partir de la regió promotora, hem determinat un conjunt de factors de transcripció que podrien unir-se a aquesta regió. Com que la proteïna és una proteïna quimèrica que prové d'un gen fusionat producte d'una translocació, hem seleccionat la regió promotora del gen que es troba en primera posició, és a dir, el NPM1.
Per tal de seleccionar el conjunt de factors que s'uneixen a la regió promotora, ho hem fet de dues formes: d'una banda mitjançant un programa en Perl que hem desenvolupat nosaltres mateixes, d'altra banda, utilitzant el servidor web del programa PROMO.
Programa en Perl:
Aquest programa ha estat desenvolupat per tal de que a partir d'una sèrie de matrius donades, que han estat extretes de les matrius utilitzades pel programa PROMO i a partir d'una seqüència promotora en format fasta, mitjançant una sèrie de procediments, ens proporcioni el p-value de cada factor de transcripció. El p-value és la probabilitat de que el factor de transcripció s'uneixi a la seqüència promotora per atzar, per tant, com més baix sigui aquest valor, ens indica que la probabilitat de que el factor de transcripció s'uneixi a la seqüència promotora, és alta, és a dir, que la probabilitat de que s'uneixi per atzar és baixa.
Programa PROMO :
Aquest programa ens mostra un llistat de factors de transcripció que es poden unir a la seqüència promotora del gen d'interès. En introduir la seqüència, per defecte ens dóna els factors de transcripció que presenten com a màxim un 15% de dissimilaritat. Per tal d'acotar la llista de factors de transcripció, hem posat el màxim de dissimilaritat a 3%. D'aquesta manera, obtenim aquest llistat. Ens fixem en aquells que presenten un RE query inferior a 0'09 (marcats en color verd), ja que com més baix sigui el valor, ens indica que la probabilitat de que el factor de transcripció s'uneixi i que no sigui degut a l'atzar, és alta. A més, els valors de dissimilaritat són també baixos en aquests factors de transcripció seleccionats.
El programa PROMO utilitza moltes matrius per un factor de transcripció. Si comparem els resultats obtinguts amb el nostre programa en Perl i els obtinguts amb el programa PROMO, no coincideixen. La raó és el fet de que PROMO utilitza moltes matrius per un factor de transcripció, i en el programa en Perl només utilitzem una matriu. Segurament les matrius que nosaltres hem utilitzat i les utilitzades per PROMO són diferents. A part d'això, el procés per determinar els factors de transcripció que s'uneixen a la regió promotora és molt més complex que el que hem realitzat mitjançant el programa en Perl.
Veiem, però, que hi ha alguns factors de transcripció que sí coincideixen: RXR-alpha [T01345] i YY1 [T00915], aquest últim amb un valor RE query molt alt en PROMO. Tot i això, els llocs d'unió no són els mateixos.