Abstract |
|
|
Resultats
Els resultats que s'expliquen a continuació són els obtinguts seguint els procediments detallats més endavant (veure apartat de Mètodes).
1. Estructura genòmica
Després de fer un BLASTP1
(proteïna-proteïna) de la nostra seqüència contra tot el genoma humà,
s'ha pogut determinar que la seqüència proteica donada inicialment
correspon a la proteïna de fusió PML-RAR, ja que és la que
ha presentat un major score, concretament de 1395, i un E-value (probabilitat d'error) de 0.
A continuació es mostra la seqüència inicial que se'ns va proporcionar i a partir de la qual s'ha desenvolupat tot l'estudi:
MEPAPARSPRPQQDPARPQEPTMPPPETPSEGRQPSPSPSPTERAPASEEEFQF LRCQQCQAEAKCPKLLPCLHTLCSGCLEASGMQCPICQAPWPLGADTPALDNVF FESLQRRLSVYRQIVDAQAVCTRCKESADFWCFECEQLLCAKCFEAHQWFLKHE ARPLAELRNQSVREFLDGTRKTNNIFCSNPNHRTPTLTSIYCRGCSKPLCCSCA LLDSSHSELKCDISAEIQQRQEELDAMTQALQEQDSAFGAVHAQMHAAVGQLGR ARAETEELIRERVRQVVAHVRAQERELLEAVDARYQRDYEEMASRLGRLDAVLQ RIRTGSALVQRMKCYASDQEVLDMHGFLRQALCRLRQEEPQSLQAAVRTDGFDE FKVRLQDLSSCITQGKAIETQSSSSEEIVPSPPSPPPLPRIYKPCFVCQDKSSG YHYGVSACEGCKGFFRRSIQKNMVYTCHRDKNCIINKVTRNRCQYCRLQKCFEV GMSKESVRNDRNKKKKEVPKPECSESYTLTPEVGELIEKVRKAHQETFPALCQL GKYTTNNSSEQRVSLDIDLWDKFSELSTKCIIKTVEFAKQLPGFTTLTIADQIT LLKAACLDILILRICTRYTPEQDTMTFSDGLTLNRTQMHNAGFGPLTDLVFAFA NQLLPLEMDDAETGLLSAICLICGDRQDLEQPDRVDMLQEPLLEALKVYVRKRR PSRPHMFPKMLMKITDLRSISAKGAERVITLKMEIPGSMPPLIQEMLENSEGLD TLSGQPGGGGRDGGGLAPPPGSCSPSLSPSSNRSSPATHSP
Figura 1. Seqüència inicial
A partir de la informació continguda dins la base de dades de NCBI2,
s'ha pogut determinar que la proteïna PML-RAR (Accession: M73779) té
una longitud de 3036 parells de bases, dels quals 2394 codifiquen per a
797 aminoàcids. La proteïna PML-RAR està constituïda per 10 exons.
Per tal d'obtenir informació més detallada de l'estructura genòmica de la proteïna de fusió, es va intentar buscar en les bases de dades genòmiques (NCBI, Ensembl4 i UCSC5), però al tractar-se d'una proteïna generada per una translocació, no es pot trobar informació d'un gen únic. Tot i així, degut a la seva accessibilitat i fàcil interpretació (veure apartat de Mètodes), la base de dades escollida per centrar l'estudi ha estat Ensembl.
Per aquest fet, s'ha fet un BLASTN (nucleòtid-nucleòtid) de la seqüència de PML-RAR contra tot el genoma d'Homo sapiens però ara des d'Ensembl.
Coneixent l'origen de la proteïna de fusió, per analitzar el resultat
obtingut només s'han tingut en compte les coincidències en seqüències
peptídiques dels cromosomes 15 i 17. Posteriorment, s'han ordenant les
dades segons el valor de score i s'ha observat que la seqüència que
presenta una major semblança amb el CDS de PML-RAR és la proteïna RARA.
La taula següent ens mostra els resultats:
| Seqüència proteïca | Cromosoma | Score | E-value | %ID |
| ENSP00000316769 | Cromosoma 17 | 2587 | 3,5-e225 | 84,55 |
En canvi, en analitzar les coincidències
trobades amb la seqüència dins del cromosoma 15, entre aquestes no s'hi
ha inclòs la seqüència de PML. Això és degut a que la proteïna de fusió
només conté 3 exons provinents de PML i, per tant, al tractar-se d'una
porció de seqüència tan curta, en fer el BLAST el %identitat i el score
no donen valors significatius.
En l'esquema que es mostra a continuació
s'observa la composició de la proteïna de fusió, que és dels 3 primers
exons de PML i els 7 últims provinents de la proteïna RARA:
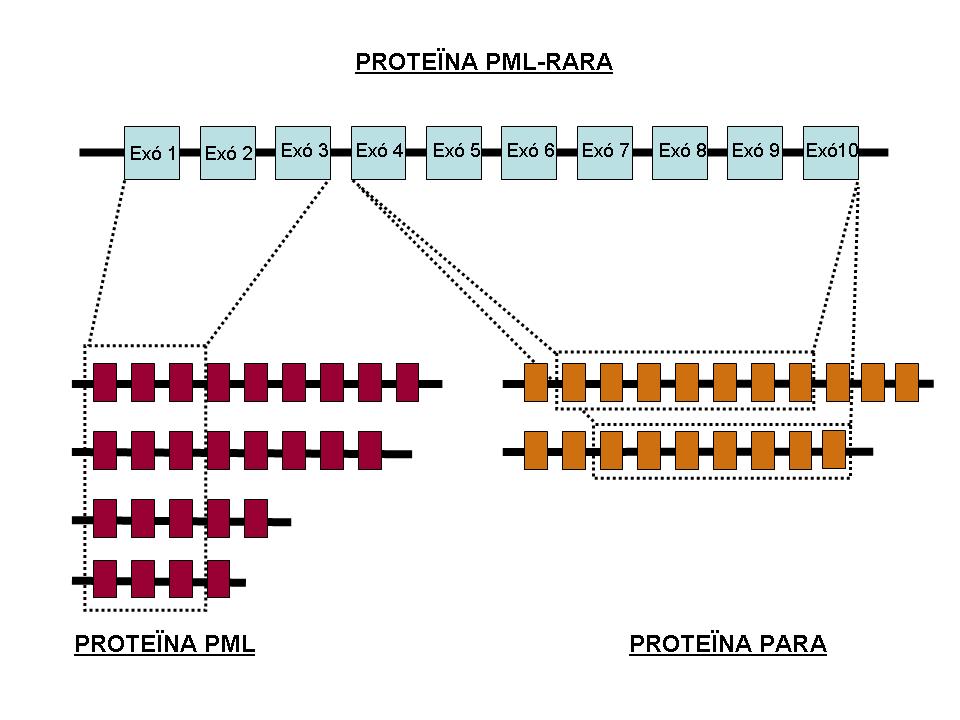
Figura 2. Distribució exònica de la proteïna PML-RAR
Tot seguit, es detallen les característiques
genòmiques de les seqüències que codifiquen per les proteïnes PML i RARA,
que donen lloc a la proteïna de fusió per translocació.
a) PML (Ensembl Gene ID: ENSG00000140464)
El gen que codifica per la proteïna PML (Acute
Promyelocytic Leukemia Inductor) té una llargada de 53137 parells de
bases i es troba situat en el cromosoma 15, concretament en la regió
compresa entre les bases 72.074.067 i 72.127.204 (q24.1, en el braç
gran del cromosoma).

Figura 3. Ubiació de la proteïna PML dins el cromosoma 15
Aquesta proteïna conté un total de 9 exons, la
codificació dels quals varia en funció dels diferents transcrits.
Segons la base de dades d'Ensembl, la seqüència de PML dóna lloc a 4
transcrits diferents.
A continuació se'n detallen les característiques principals:
| Ensembl ID | Llargada total (nt) | Llarda proteïna (aa) | Exons codificants | Exons no codificants |
| ENST0000268058 | 5.591 nt | 882 aa | 9 (1r i últim pacials) | 142/269 (1r) + 2848/3590 (últim) |
| ENST0000268059 | 2.630 nt | 829 aa | 8 (1r parcial) | 142/269 (1r) |
| ENST0000354026 | 2.486 nt | 781 aa | 7 (1r parcial) | 142/269 (1r) |
| ENST0000359928 | 1448 nt | 435 aa | 5 (1r parcial) | 142/269 (1r) |
Taula 1. Transcrits de la proteïna PML (font: Ensebml)
Cal tenir en compte que, tot i la diferència en
quant als nombre d'exons codificants, aquest fet no afecta a la
proteïna de fusió objecte de l'estudi. Això és degut a que són només
els 3 primers exons de PML els implicats en la proteïna de fusió
PML-RAR, els quals s'ha vist que són idèntics. També s'ha realitzat un l'alineament dels 4 transcrits amb el programa ClustalW3, en el que
es pot veure com, de fet, els quatre primers exons són iguals pels 4
transcrits (1254 primers nucleòtids).
D'altra banda, destacar que la diferència en els
exons restants tampoc és massa rellevant perquè tots els transcrits
donen lloc a la mateixa proteïna (PML). A continuació es mostra un
esquema del diferents transcrits que s'han anat originant per splicing
alternatiu.
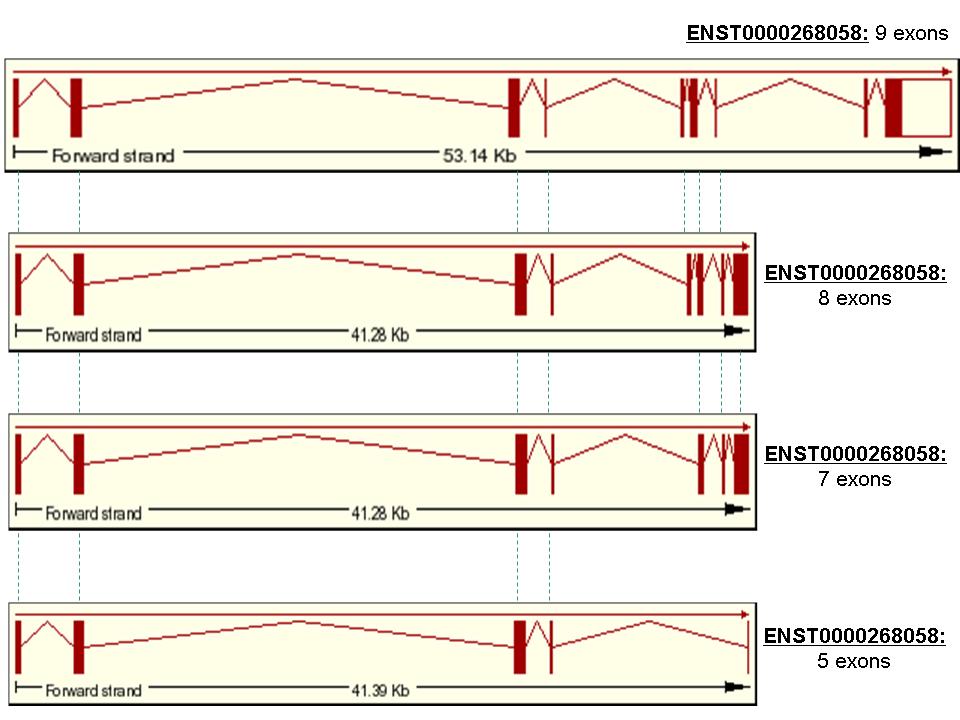
Figura 4. Esquema dels transcrits de la proteïna PML originats per splicing alternatiu
Tal i com es pot veure en la figura 4, tots els
transcrits tenen els 4 primers exons en comú. Més detalladament i
comparant els transcrits, en base al primer i més complert; tant a
partir de l'alineament i com de l'anàlisi de les seqüències exòniques,
es pot concloure:
- El segon transcrit (ENST0000268059), respecte el primer, presenta els 7 primers exons iguals, però ha patit una pèrdua per splicing alternatiu de l'exó 8. Pel que a l'exó 9, els dos transcrits només comparteixen alguns parells de bases.
- El tercer transcrit (ENST0000354026), presenta els 4 primers exons iguals, però ha patit una pèrdua de l'exó 5 del primer transcrit, per splicing. Els exons 6 i 7 del primer transcrits, es correspon perfectament amb els exons 5 i 6 d'aquest tercer. Finalment, com en el cas del segon transcrit, l'últim exó només presenta alguna residus en comú amb l'exó 9 del primer transcrit. D'altra banda, destacar que l'exó 8 del segon transcrit i el 7 d'aquest tercer són exactament iguals.
- El quart transcrit (ENST0000359928), presenta els 4 primers exons en com amb la resta de transcrits. A més a més, conté 54 parells de bases que no coincideixen amb cap residu dels altres transcrits. Per tant, per splicing alternatiu el quart transcrit dóna lloc a una seqüència més curta que la resta de transcrits, però que codifica per la mateixa proteïna.
En aquest punt cal destacar la diferència
d'informació que trobem continguda en les diferents bases de dades, ja
que en buscar informació referent a les diferents isoformes de la
proteïna hem obtingut dades diferents.
Per a Ensembl, no s'ha pogut saber si els 4
transcrits donen lloc a isoformes diferents, ja que no s'especifica
aquesta informació. El que si es pot confirmar és que tots ells
codifiquen per la mateixa proteïna. Des d'Ensembl s'ha pogut accedir a
la base de dades de UniProt6, en la qual apareixen les 5 isoformes de la
proteïna PML. Les isoformes són:
| Nom isoforma | Identificador (UniProt) |
| PML-1 | P29590-1 |
| PML-2 | P29590-2 |
| PML-3 | P29590-3 |
| PML-3B | P29590-4 |
| PML-X | P29590-5 |
Taula 2. Isoformes proteïPML (font: UniProt)
D'altra banda, s'ha realitzat un alineament amb ClustalW i s'ha pogut veure que aquestes isoformes tenen en comú els 6 primers exons (1656 primers nucleòtids), i que per la resta de la seqüència divergeixen força entre ells. També s'ha realitzat un alineament dels les seqüències dels 4 transcrits amb les seqüències de les 4 isoformes, per tal d'intentar trobar correlacions que permetin saber quins transcrits donen lloc a les diferents isoformes.
De l'alineament es pot dir que:
- La primera isoforma ve donada pel primer transcrit, que es considera la forma més habitual en la que es troba la proteïna PML.
- La isoforma 2 pels primers 570 nucleòtids és igual que els tres primers transcrits. Tot i així, aquesta isoforma té 14 residus més, que difereixen molt de tots els altres transcrits, fent-se difícil la identificació del seu origen. Només l'últim nucleòtid, en posició 583, que és una serina és comú amb els tres primers transcrits.
- La isoforma 3 podria molt probablement venir donada pel transcrit 2, al qual és quasi idèntic en quant a seqüència a excepció de 5 residus (584-588), corresponents a l'exó 8, que desapareixen en la isoforma. També s'assembla molt al transcrit 3, tot i que aquest difereix del transcrit 2 en què per splicing alternatiu ha patit una pèrdua de l'exó 5, tal i com ja s'ha comentat anteriorment.
- La quarta isoforma té els primers 552 nucleòtids en comú amb els tres primers transcrits, és a dir, fins a l'exó 6. Després té 8 residus addicionals d'origen desconegut, en quant al transcrit. Destacar que aquesta és la isforma més curta amb només 560 residus.
- La isoforma 5 és idèntica a la resta d'isoformes fins al residu 570 o final de l'exó (excepte la isoforma 4) i després és igual a nivell de seqüència al transcrit 1 fins al final de l'exó 8 (residu 620), finalment posseeix 13 residus diferents.
Cal tenir en compte que la seqüència de totes les isoformes és més llarga que el quart transcrit, el qual és bastant més curt que la resta. Tot i així, totes les isoformes són idèntiques fins al residu 552 (final de l'exó 6) i totes donen lloc a la mateixa proteïna. Per tant, podem concloure que tot i les diferències, sembla que els residus extres són tots codificants i que no es produeix una modificació de la pauta de lectura que afecti a la funcionalitat de la proteïna.
b) RARA (Ensembl Gene ID: ENSG00000131759)
La proteïna RARA (Retinoic Acid Receptor Alpha) té
una llargada de 48448 parells de bases i es troba situada al cromosoma
17, concretament a la regió compresa entre les bases 35.718.972 i
35.767420 (q12, en el braç gran del cromosoma).

Figura 6. Ubiació de la proteïna RARA dins el cromosoma 17
Aquesta proteïna conté un total de 16 exons, la
codificació dels quals varia en funció dels diferents transcrits.
Segons la base de dades d'Ensembl, la seqüència de la proteïna RARA
dóna lloc a 2 transcrits diferents. El primer dels dos transcrits
(ENST0000254066) presenta un total de 9 exons i el segon transcrit
(ENST0000319149) un total d'11 exons. Les característiques principals d'aquests dos transcrits es detallen en la taula següent:
| Ensembl ID | Llargada total (nt) | Llarda proteïna (aa) | Exons codificants | Exons no codificants |
| ENST0000254066 | 3.260 nt | 462 aa | 6 exons sencers codificants (del 3r al 8è) i 2 exons parcialment codificants (el 2n i el 9è) | 1r exó (93 nt) + 362/540 (2n) + 218/1634 (9è) |
| ENST0000319149 | 3.395 nt | 633 aa | 89 exons sencers codificants i 2 exons parcialment codificants (el 1r i el 11è) | 615/778(1r) + 612/1490(11è) |
Taula 3. Transcrits de la proteïna RARA (font: Ensebml)
Si analitzem els valors de la taula, podem veure
diferències entre els dos transcrits en el nombre d'exons codificants.
Malgrat aquest fet, aquesta diferència no afecta a la formació de la
proteïna de fusió PML-RAR; doncs les seqüències dels exons de la
proteïna de fusió provinents de RARA, són iguals en els
dos transcrits. En el primer (ENST0000254066) corresponen als exons del
2 al 10 i en el segon (ENST0000319149) del 3 al 11. Per a corroborar
aquest fet s'ha realitzat un alineament amb ClustalW. (link)
A continuació es mostra un esquema del diferents transcrits que s'han anat originant per splicing alternatiu.
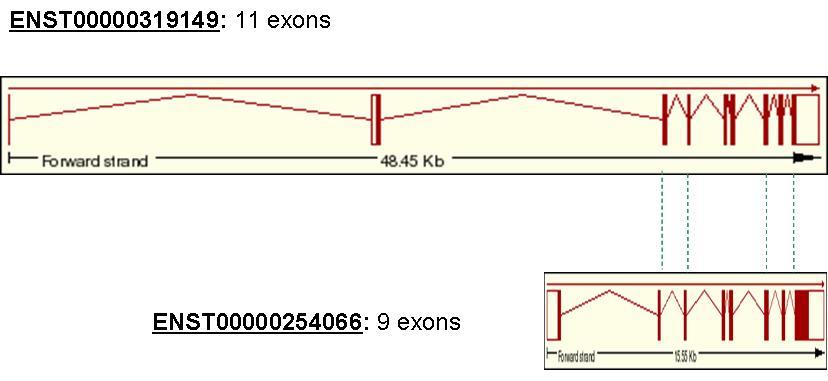
Figura 7. Esquema dels transcrits de la proteïna RARA originats per splicing alternatiu
Com es pot veure en la figura anterior, els 2
transcrits presenten 4 exons en comú (concretament els exons 3,4 7 i 8
del transcrit ENST0000254066), que es corresponen amb els exons 2, 3 8
i 9 del transcrit ENST0000319149). Cal destacar però que malgrat els
altres exons no coincideixin exactament en número de parells de bases,
els nucleòtids sí que són iguals en els dos transcrits tot i que formen
part d'exons diferents. Aquest fet és important per tal d'entendre que
ambdós transcrits poden acabar formant la proteïna de fusió PML-RARA, i
que és per acció de l'splicing alternatiu que aquests dos transcrits
presenten les diferències explicades anteriorment.
Més detalladament i comparant els transcrits, es pot concloure que:
El primer transcrit (ENST0000254066) presenta 6 dels 9 exons codificants, i el primer exó sencer i part del segon i del novè no codificants. Per altra banda, el segon transcrit (ENST0000319149) presenta 9 exons codificants dels 11 que conformen el gen de la proteïna, i 2 exons parcialment no codificants (el primer i l'últim). Cal destacar que, el primer dels exons codificants del transcrit ENST0000319149, és a dir, el segon exó d'aquest es correspon amb el tercer exó del transcrit ENST0000254066.
Després d'analitzar els diferents transcrits de la proteïna RARA, analitzarem les isoformes d'aquesta proteïna. Per a Ensembl, no s'ha pogut saber si els 2 transcrits donen lloc a isoformes diferents, ja que no s'especifica aquesta informació, però es pot confirmar que tots ells codifiquen per la mateixa proteïna. Des d'Ensembl s'ha pogut accedir a la base de dades de UniProt, en la qual apareixen 2 isoformes de la proteïna RARA. S'han obtingut les seqüències aminoacídiques de les isoformes i s'ha pogut comprovar que la primera isoforma de 462 aminoàcids coincideix amb el transcrit de 9 exons (ENST00000254066). Per tal de corrobarar aquest fet, s'ha realitzat un alineament de ClustalW i sembla ser que només una coincideix amb el primer transcrit.
Per altra banda, s'ha procedit a realitzar el
mateix anàlisi amb l'altra isoforma de 457 aminoàcids amb el transcrit de 11 exons (ENST0000319149) però en aquest cas, el resultat de l'alineament amb ClustalW obtingut
no ha estat l'esperat.
En aquest alineament es pot veure que la seqüència aminoacídica
d'aquesta isoforma no coincideix exactament amb la seqüència del segon
transcrit, doncs difereixen en alguns aminoàcids i, a més a més, aquesta
seqüència de la isoforma és més curta que la del transcrit. Per aquest
fet, s'ha decidit realitzar un BLASTP en la base de dades de l'Ensembl
amb les seqüències aminoacídiques d'aquesta isoforma i del transcrit
ENST0000319149. D'aquesta manera s'ha pogut determinar que la seqüència
del genoma humà més semblant a la isoforma és, amb un %ID de 88,44 i
amb un score de 2567 la seqüència del transcrit ENST0000319149.
Segons aquests resultats, la seqüència de la
primera isoforma (de 462 aminoàcids) coincideix exactament amb la
seqüència del primer transcrit (ENST00000254066) i, per tant, el
nombre d'exons és el mateix. En aquest cas doncs, i degut a aquesta
coincidència, es manté la pauta de lectura en aquesta isoforma.
Per altra banda, pel que fa a la segona isoforma
(de 457 aminoàcids), la seqüència d'aquesta, tal i com ja s'ha
explicat, no és exactament igual a la seqüència del transcrit. Les
diferències en alguns dels aminoàcids dels exons 4, 5, 6 i 7 del
transcrit ENST0000319149 de 11 exons amb la seqüència de la isoforma,
juntament amb el fet que la seqüència de la isoforma és més curta, han
permès concloure que en aquest cas sí que s'altera la pauta de lectura.
Finalment, s'ha pogut concloure, basant-se en
aquests resultats que, per a la proteïna RARA existeix una isoforma per
a cada un dels seus dos transcrits.
c) PML-RAR:
Per acabar l'estudi a nivell genòmic, a continuació es comenten característiques del gen híbrid resultant de la fusió. Aquestes, han estat obtingudes de la lectura de diferents articles i bases de dades:
El transcrit crucial de fusió que donarà lloc a la proteïna de fusió és el 5'PML-3'RARA, codificat per una porció del cromosoma 15 i una del cromosoma 17.
El punt de trencament o breakpoint del gen RARA està sempre localitzat entre els dominis A i B.
El breakpoint de PML resideix en els dos costats de l'exó resultant del splicing alternatiu. En el gen de PML existeixen 3 clusters amb breakpoints que són:
- bcr1 (en un 70% dels pacients)
- bcr2 (en un 10%)
- bcr3 (en un 20%)
Aquests donen lloc cadascun, transcrits híbrids de PML-RARA de longitud llarga (L), intermitja (V) i curta (S), respectivament. La forma V està relacionada amb una disminució de la sensibilitat a ATRA (All-Trans Retinoic Acid), un tipus de teràpia amb àcid retinoic. En canvi, la forma S està associada a un excés de canvis secundaris en el cromosoma.
2. Conservació en altres espècies
De la mateixa manera que s'ha fet amb l'estudi de l'estructura genòmica, en aquest apartat s'ha estudiat la conservació de cada un dels gens que conformen la proteïna de fusió per separat.
a) PML (Ensembl Gene ID: ENSG00000140464)
A partir de la base de dades d'Ensembl s'han trobat els gens ortòlegs a la proteïna PML d'Homo sapiens. Aquests són els que es mostren en la taula seqüent. Cal destacar que els resultats coincidèixen amb els obtinguts a partir de la base de dades Biomart7. A més a més ,aquesta última base de dades ha permès seleccionar només un dels transcrits amb el major percentatge d'identitat en aquelles espécies, com Gallus gallus, que presentaven (segons Ensembl) diferents ortòlegs pel gen de la proteïna PML.
| Nom de l'espècie | Imatge de l'espècie | Identificador del gen | Percentatge d'identitat |
| Bos taurus |  |
ENSBTAG00000015779 | 74% |
| Canis familiaris |  |
ENSCAFG00000017858 | 38% |
| Cavia porcellus |  |
ENSCPOG00000001304 | 29% |
| Dasypus novemcinctus |  |
ENSDNOG00000009847 | 35% |
| Echinops telfairi |  |
ENSETEG00000019736 | 45% |
| Felis catus |  |
ENSFCAG00000003098 | 78% | Gallus gallus |  |
ENSGALG00000001478 | 29% |
| Loxodonta africana |  |
ENSLAFG00000002717 | 53% |
| Macaca mulatta |  |
ENSMMUG00000002569 | 92% |
| Monodelphis domestica |  |
ENSMODG00000009808 | 42% |
| Mus musculus |  |
ENSMUSG00000036986 | 68% |
| Ornithorhynchus anatinus |  |
ENSOANG00000008921 | 19% |
| Oryctolagus cuniculus |  |
ENSOCUG00000002183 | 48% |
| Pan troglodytes |  |
ENSPTRG00000007266 | 99% |
| Rattus norvegicus |  |
ENSRNOG00000008400 | 69% | Tupaia belangeri |  |
ENSTBEG00000012807 | 63% |
A partir de les seqüències dels transcrits dels homòlegs trobats, s'ha realizat un alineament amb ClustaW, per tal de visualitzar més fàcilment la relació d'homologia existent entre el gen de la proteïna PML d'Homo sapiens i els ortòlegs en altres espècies.
D'altra banda, aquest alineament ha permès generar un arbre filogenètic que plasmi millor la relació i distàncies evolutives entre les espècies per aquest gen.
Finalment, per tal de determinar altres possibles ortòlegs per al gen de la proteïna PML, s'ha fet un BLASTN des de NCBI amb la seqüència de tot el gen de PML contra les bases de dades de tots els genomes (opció: nr) de NCBI. Els resultats obtinguts, doncs, permeten saber d'altres espècies en les quals s'hi troba un gen amb alta similaritat al de PML. D'aquests resultats se'n destaquen els següents per tenir un E-value de 0 i un elevat % d'identitat amb el gen, així com un valor de score significatiu (major de 3000):
| Nom espècie | Imatge de l'espècie | Identificador (NCBI) | Max identitat | Score màxim |
| Gorilla gorilla |  |
DQ231465.1 | 99% | 3376 |
| Pan paniscus |  |
DQ231463.1 | 99% | 3350 |
| Pongo pygmeus |  |
DQ231466.1 | 98% | 3247 |
| Nomascus leucogenys |  |
DQ231468.1 | 98% | 3168 |
| Hylobates lar |  |
DQ231467.1 | 98% | 3154 |
| Symphalangus syndactylus |  |
DQ231469.1 | 97% | 3130 |
b) RARA (Ensembl Gene ID: ENSG00000131759)
A partir de la base de dades d'Ensembl s'han trobat els gens ortòlegs a la proteïna RARA d'Homo sapiens. Aquests són els que es mostren en la taula adjunta. És important destacar que aquests resultats coincidèixen amb els obtinguts a partir de la base de dades Biomart. A més a més ,aquesta última base de dades ha permès seleccionar només un dels transcrits amb el major percentatge d'identitat en aquelles espécies, com Dario rerio o Tukifugu rubripes, que presentaven (segons Ensembl) diferents ortòlegs pel gen de la proteïna RARA.
| Nom de l'espècie | Imatge de l'espècie | Identificador del gen | Percentatge d'identitat |
| Canis familiaris |  |
ENSCAFG00000016060 | 54% |
| Cavia porcellus |  |
ENSCPOG00000007730 | 24% |
| Ciona intestinalis |  |
ENSCING00000006945 | 34% |
| Ciona savignyi |  |
ENSCSAVG00000006794 | 35% |
| Danio rerio |  |
ENSDARG00000034893/ENSDARG00000054003 | 52% |
| Dasypus novemcinctus |  |
ENSDNOG00000011604 | 37% |
| Echinops telfairi |  |
ENSETEG00000013732 | 64% |
| Erinaceus europaeus |  |
ENSEEUG00000006607 | 60% |
| Gallus gallus |  |
ENSGALG00000005629 | 8% |
| Gasterosteus aculeatus |  |
ENSGACG00000005297 | 48% |
| Loxodonta africana |  |
ENSLAFG00000016234 | 70% |
| Macaca mulatta |  |
ENSMMUG00000012486 | 97% |
| Monodelphis domestica |  |
ENSMODG00000013112 | 53% |
| Mus musculus |  |
ENSMUSG00000037992 | 54% |
| Ornithorhynchus anatinus |  |
ENSOANG00000012527 | 19% |
| Oryctolagus cuniculus |  |
ENSOCUG00000004903 | 49% |
| Oryzias latipes |  |
ENSORLG00000004373 | 34% |
| Pan troglodytes |  |
ENSPTRG00000009120 | 93% |
| Rattus norvegicus |  |
ENSRNOG00000009972 | 54% |
| Takifugu rubripes |  |
SINFRUG00000142757 | 52% |
| Tetraodon nigroviridis |  |
GSTENG00024106001 | 52% |
| Xenopus tropicalis |  |
ENSXETG00000024390 | 51% |
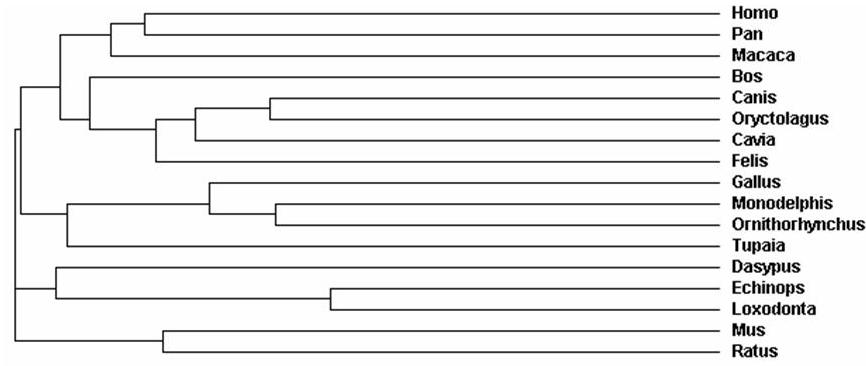
Tal i com s'ha realitzat amb la proteïna PML, a partir de les seqüències dels transcrits dels homòlegs trobats, s'ha realizat un alineament amb ClustaW, per tal de visualitzar més fàcilment la relació d'homologia existent entre el gen de la proteïna RARA d'Homo sapiens i els ortòlegs en altres espècies.
D'altra banda, aquest alineament ha permès generar un arbre filogenètic que plasmi millor la relació i distàncies evolutives entre les espècies per aquest gen.

Figura 9. Arbre filogenètic de la proteïna RARA (font: Ensebml)
Els resultats obtinguts a partir de les taules anteriors i dels arbres filogenètics seran analitzats en l'apartat de Discussió, tant per a la proteïna PML com per a la proteïna RARA.
3. Caracterització de l'expressió
Per tal d'estudiar l'expressió de la proteïnes PML i RARA que constitueixen la proteïna de fusió PML-RAR, s'han utilitzat diverses bases de dades. Aquestes són: Genome Browser de l'UCSC, Gene Sorter de l'UCSC, Gene-Hub Gepis, i Gene Expression Atlas.
Tal i com s'ha realitzat en els apartats anteriors, primer s'analitzarà l'expressió de la proteïna PML i seguidament, l'expressió de la proteïna RARA.
a) PML (Ensembl Gene ID: ENSG00000140464)
Segons la base de dades del Genome Browser de l'UCSC, els resultats obtinguts de l'anàlisi per microarray de la proteïna PML han estat els següents:
Per a l'anàlisi realitzat amb: GNF Expression Atlas 2 Data from U133A and GNF1H Chips no s'oberva, en cap dels teixits analitzats, una sobreexpressió calarament evident de la proteïna PML. Cal destacar però, que malgrat aquest fet és clar i dominant per a la majoria dels teixits, els resultats mostren una lleugera i tènue sobreexpressió d'aquesta proteïna en cèl·lules d’alguns teixits del fetge i del múscul esquelètic. Alhora, i paral·lelament a aquest fet, es poden observar teixits d'alguns òrgans (com la glàndula adrenal, el pàncrees, el tracte gastrointestinal, els pulmons fetals, i el fetge fetal) on per contra l'expressió tissular de la proteïna PML es veu minvada. Cal citar per últim, que per a la resta de teixits, no s'observa una expressió anormal (per sobreexpressió o per subexpressió) de la proteïna PML.
Per a l'anàlisi realitzat amb: Normal Human Tissue cDNA Microarrays no s'observa en cap dels teixits analitzats una sobreexpressió clara de la proteïna estudiada. Com en el cas de l'anàlisi anterior, només en un teixit (múscul abdominal) es pot observar una lleugera coloració vermellosa, fet que indica una petita sobreexpressió de la proteïna PML en aquest teixit. Per altra banda, aquest anàlisi ha permès obtenir un major nombre de teixits on sembla subexpressar-se de manera molt clara la proteïna d’interès. Alguns d'aquests teixits són: testicles, vesícula seminal, pròstata, ovaris, vagina, canal del cèrvix, placenta, glàndula salival i paròtida, ili, el cor(FT24 mRNA),entre d'altres.
Per últim, la base de dades Genome Browser ens facilita els resultats d'un altre anàlisi d'expressió realitzat amb GNF Expression Atlas 1 Human Data on Affy U95 Chips. En aquest anàlisi es pot veure subexpressió en alguns teixits limfoblàstics, fetge fetal, i glàndules pituïtàries, principalment. Alhora, es pot observar també una sobreexpressió tissular en melsa, fetge i cor.
En la imatge següent es poden veure, de manera general, aquells teixits on s'expressa la proteïna PML.

Figura 10. Expressió de la proteïna PML en anàalisi de microarrays
Seguidament, s'ha fet servir la base de dades del Gene Sorter de l'UCSC per tal d'estudiar l'expressió de la proteïna PML. En aquest cas els resultats obtinguts han estat:
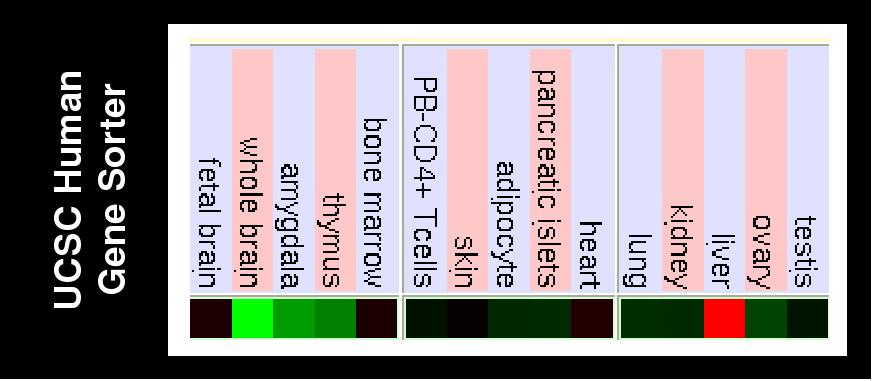
Figura 11. Expressió de la proteïna PML en anàalisi per Gene Sorter
En la imatge anterior, podem veure l'expressió de diferents teixits per mitjà de coloració vermella i verdosa. Aquests valors han estat configurats amb una brillantor de 10, per tal de veure més clarament els resultats.
D'aquests resultats és important destacar la sobreexpressió de la proteïna PML en teixits de fetge, resultat que coincideix amb l'obtingut en l'anàlisi GNF Expression Atlas 1 Human Data on Affy U95 Chips. Per altra banda, cal fer esment també de la baixa expressió de la proteïna estudiada en el cervell.
A partir de la base de dades de Gene-Hub Gepis s'han pogut obtenir diferents informació d'expressió de la proteïna PML en teixits normals, que han permè elaborar un gràfic. En aquest gràfic es pot veure clarament, que la proteïna PML s'expressa de manera molt important en diferents teixits com l'os, el múscul, l'ovari i el sistema nerviós perifèric. Aquests resultats coincidèixen amb els de l'anàlisi de Normal Human Tissue cDNA Microarrays on també s'observa, tal i com ja s'ha explicat, l'expressió d'aquesta proteïna cèl·lules del teixit muscular.
Per últim s'ha analitzat també la base de dades Gene Expression Atlas i s'ha pogut veure (mitjançant de les dades reflexades en un gràfic) que la proteïna estudiada presenta elevada expressió tissular en alguns òrgans com la melsa, el fetge d'adults i en cèl·lules HL60 (línia cel·lular que permet estudiar la diferenciació de les cèl·lules mieloides humanes). Aquests resultats concorden força amb els obtinguts en les anteriors bases de dades.
Finalment, després de fer aquest anàlisi comparatiu dels resultats de les diferents bases de dades, s'ha pogut concloure de manera general que la proteïna PML s'expressa de manera important, i de manera similar a com ho fa en cèl·lules HL60, en alguns òrgans com la melsa, el teixit muscular i el fetge de l'individu adult. De manera paral·lela es presenta subexpressat en alguns teixits com el fetge fetal i glàndula pituïtària.
Per altra banda en l'article PML inducer of Acute promyelocytic leukemia, s'explica la realització d'un experiment amb cèl·lules HeLa per tal d'estudiar si l'expressió de la proteïna PML està o no relacionada amb el cicle cel·lular. Es van utilitzar cèl·lules HeLa sincronitzades a diferents fases del cicle cel·lular i es va analitzar per mitjà de tècniques d'immunofluorescència i microscopia confocal, la fosforilació dels residus de tirosina i seria de la proteïna PML. Per mitjà d'aquest anàlisi, es va poder determinar que la proteïna PML presenta un baix nivell d'expressió, en cèl·lules en fase S, G2 i M. Per altra banda, l'article també cita que aquesta proteïna presenta un alt nivell d'expressió en fase G1.
b) RARA (Ensembl Gene ID: ENSG00000131759)
Segons la base de dades obtingudes a partir d'un gràfic del Gene Expression Atlas la proteïna RARA presenta un nivell alt d'expressió en cè·lules del teixit sanguini, melsa, pulmons, ovari i pàncreas.
Aquestes dades coincideixen clarament amb els resultats obtinguts en la base de dades del Genome Browser.
Per mitjà de l'anàlisi realitzat amb GNF Expression Atlas 1 Human Data on Affy U95 Chips s'han pogut corroborar els resultats d'expressió de la proteïna RARA obtinguts a partir del Gene Expression Atlas. En aquest anàlisi es mostra una important sobreexpressió de la proteïna RARA en teixits sanguini, melsa, pulmons i ovaris, tal i com es mostra en la base de dades anterior.
En l'anàlisi realitzat amb Normal Human Tissue cDNA Microarrays es pot veure clarament que existeix una sobreexpressió de la proteïna RARA en teixits com diafragma, bufeta, timus, entre d'altres.
Per últim, l'anàlisi de microarray realitzat amb GNF Expression Atlas 2 Data from U133A and GNF1H Chips, ha permès estudiar aquells teixits on la proteïna RARA es troba subexpressada sobretot en teixits d'òrgans del sistema nerviós com per exemple, l'hipotàlam, el lòbul occipital i el nucli subtalàmic, entre d'altres.
Finalment, en conclusió podem dir que per a la proteïna RARA existeix una expressió tissular diferencial a favor, sobretot del teixit sanguini, però també presenta sobreexpressió en altres òrgans com la melsa, els ovaris i els pulmons.
En la imatge següent es pot veure els principals resultats obtinguts en fer l'anàlisi amb microarray.
Figura 12. Expressió de la proteïna RARA en anàalisi per Gene Sorter
Paral·lelament a la cerca d'aquestes dades, també s'ha buscat informació en la base de dades Gene sorter de l'UCSC. Per a realitzar aquest anàlisi, igual que per a la proteïna PML, s'ha configurat el programa amb una brillantor de 10, per tal de poder visualitzar millor els resultats. Així el resultat obtingut ha estat:

Figura 13. Expressió de la proteïna RARA en anàalisi per Gene Sorter
És important també destacar l'informació obtinguda a partir de la base de dades de Gene-Hub Gepis que ha permè elaborar un gràfic que reflexa l'expressió de la proteïna RARA en teixits normals. En aquest gràfic es pot veure clarament, que la proteïna RARA s'expressa de manera molt important en diferents òrgans com el cor, la melsa i el sistema vascular. Aquests resultats coincidèixen amb els de l'anàlisi realitzat amb Genome Browser.
En l'apartat de discussió s'explica més detalladament les conclusions en quant a l'expressió de les proteïnes, l'alteració de la seva funció, i les repercusions que aquest fet comporta.
4. Caracterització de les regions promotores
A partir de la base de dades UCSC (per mitjà de l'opció DNA), s'ha obtingut la situació de les seqüència genòmica de cada un dels gens dins dels cromosomes corresponents. A partir d'aquestes dades, s'ha calculat la situació de la regió promotora per a cada un dels gens: 1kb upstream del lloc de començament de la transcripció(Transcription start site o TSS) i 100 parells de bases downstream del TSS. En la següent taula es mostren les seqüències promotores de cada un dels gens.
| Nom del gen | Localització de la regió promotora | Seqüència promotora |
| PML | Cromosoma 15: 72073193-72074292 | GACATGCTTTTCCATTGGCGAAGACCTAGCTGGCTCTCCCCTCACCCTCTTCATACCCG CTCTCCAGCTCTCCCCCTCCCTCCTGCCCCCACCACCTCAGATCCACCAGCCTTTCCAGC TGTCACTCAAGGCATCATCCTGCTCCCTCACCTCCAGTCACTGGCTTCCCAGGATCTCT GAATCAAGTCCCTCCTCTGCATCCCACAGCCACTGTCCTAGACCAGGGCACCTGCTCCT AGGTTGTCAGAGTGACTGTCCCATCATGCACAGCTGATCGTGTTGTTCCCTGGTTCCAA ATCCTTCAGCAACTTCCCATCACTCACAGAACTGGTTCAATCTCCTCAGCTAGGATCTT GTCCTAGCACACTCCTTGGGTATCATTTCTCATGAATTCCCTCTTAAATCTGAAAATGG CCAAAATTAGTTCCCAAACAGGCTTTTGCAAGAGCTATCTTTTCTGTTTCAAGTGTCCC CCGTACCCCACCACCTACAACCCTAAATCCCTGGTGAATTTCCACTGCTTTTGCAGGTC CCAGCTGTGGGCTCTCCTTTCCAGAATGTCTCCGTTTGCAGGCTTGCTCTTCCATTACAT CTACGGCTCTTAGAAAGCAGGGGCTGCGTGTGGCTCATCTTTGTACGCCCAGCTCCCGG CAGGACGTGGCACGAAGCAGTGCCAGTGTGAACGGATGAATGGATCAAAGCCGGGAG CAGGGCTGCCCCCCTGCAGCTCTGCCCTACCTCTCCCGCTTTACCGTAAGTCAGCGGTA GGTCTGCAGCTCTCCGCCTCTACCTCCTCCCCGCTCTGGGCGTGTCTTTAAAACCCACA GTCGGCCTCTCTGCCCCCTAGAACCGCCCCCAGCTTCTGTCTCACTTCCTCTCCAGAGG CGGGCCCTGAGCCGGCACCTCCCCTTTCGGACAGCTCAAGGGACTCAGCCAACTGGCT CACGCCTCCCCTTCAGCTTCTCTTCACGCACTCCAAGATCTAAACCGAGAATCGAAACT AAGCTGGGGTCCATGGAGCCTGCACCCGCCCGATCTCCGAGGCCCCAGCAGGACCCCG CCCGGCCCCAGGAGCCCACCATGCCTCCCCCCGAGACCCC |
| RARA | Cromosoma 17: 35742026-35740926 | CCATCCATGCTGTTAAGCATTGGAGGTGATACCTAGGAGTCCTTGGTTCCCATTTTCCT ATTAGTGTGTACGTAAATGGGCCTCCTCCCAGGGCCTGTGGGACAAACCCGTCTGGGG TGCTGGGGCTTGGATCTGGGGGGGAAGTGGTGTAAGAGGGGCCCCCTGGCTTAGGAGT CATTTTTCTGAGAAGTGCCAATGCTTTAGGGAAGCGGCTTTTCCTCCCCCTCCCTCTTCC CCCCCAGCACACGTTCTCTGCATCTTGACCTCAGGTAACCCTCACTGGGGGAAGGAGG GCCAGCTGCCCGTTTCTGCTGCAGTGATCCCTTGAGGTGGCACTGTGGACCCTTTCCCC TCAGGACTGTGGGTGAGAGGCCCTCCAAGGTTCTGGGCTCTGTCCCTGTCTCCCCAGCC TGGCTGTGCTATCTGCCACCCCCAGCAGTGTCTGGGCCCTGCTTCCAGGTCATCTCTGC CAGCCAGGCATGGTGCCAGCTGTCTGCTCAGCTGGGTGCCACGGGAAAATCTCCGGCA GCCCAGGTTGGGGAGCTCCACGGGGAATGCCTGTGTGCCTGTTCTTCAGTGCCCACTCA CCTGTCTTTCTTTTTTCTGCAGCATCACAGGACATGGCCCCCTCAGCCACCTAGCTGGG GCCCATCTAGGAGTGGCATCTTTTTTGGTGCCCTGAAGGCCAGCTCTGGACCTTCCCAG GAAAAGTGCCAGCTCACAGAACTGCTTGACCAAAGGACCGGCTCTTGAGACATCCCCC AACCCACCTGGCCCCCAGCTAGGGTGGGGGCTCCAGGAGACTGAGATTAGCCTGCCCT CTTTGGACAGCAGCTCCAGGACAGGGCGGGTGGGCTGACCACCCAAACCCCATCTGGG CCCAGGCCCCATGCCCCGAGGAGGGGTGGTCTGAAGCCCACCAGAGCCCCCTGCCAGA CTGTCTGCCTCCCTTCTGACTGTGGCCGCTTGGCATGGCCAGCAACAGCAGCTCCTGCC CGACACCTGGGGGCGGGCACCTCAATGGGTACCCGGTGCCTCCCTACGCCTTCTTCTTC CCCCCTATGCTGGGTGGACTCTCCCCGCCAGGCGCTCTGACCACTC |
El següent punt en l'estudi de la caracterització de les regions promotores dels gens PML i RARA, ha estat la cerca de diferents factors de transcripció per mitjà del programa PROMO.
Aquest programa ha permès trobar els factors de transcripció que s'uneixen a les seqüéncies promotores d'ambdos gens. Per a realitzar aquesta cerca s'han seleccionat diferents opcions de búsqueda. Primerament, s'ha limitant la búsqueda amb l'opció Select Species, de manera que només s'han buscarat aquells factors d'Homo Sapiens. Seguidament, s'ha realitzat pròpiament la cerca, amb l'opció Search Sites, i amb una dissimilaritat del 15% i del 10%.
| Nom del gen per cada seqüència promotora | Número de factors amb dissimilaritat 15% | Número de factors amb dissimilaritat 10% |
| PML | 61 | 57 |
| RARA | 63 | 52 |
Degut a que els resultats del PROMO realitzat amb un 15% de dissimilaritat i amb un 10% de dissimilaritat no difereixen gaire en quant al nombre de factors de transcripció obtinguts, s'ha decidit realitzar tot l'estudi de la caracterització de la regió promotora tenint en compte tots els factors de transcripció obtinguts en fer l'anàlisi amb una dissimilaritat del 15% amb el PROMO. Aquesta decisió ha estat presa per tal d'evitar la pèrdua de factors de transcripció importants, i que en fer l'anàlisi amb un 10% de dissimilaritat es puguin perdre.
El pas següent ha estat seleccionar de tots els factors de transcripció obtinguts pel programa PROMO, aquells que presenten un RE-equally inferior a 0,1.S'ha decidit centrar la selecció de factors de transcripció en aquest valor de RE-equally és, ja que podríem considerar aquest valor com un indicador del percentatge de llocs iguals a la seqüència estudiada que trobaríem en una seqüència a l'atzar.
Paral·lelament, a aquest fet s'ha desenvolupat un programa Perl per tal seleccionar aquells factors de transcripció que s'uneixen a cada una de les seqüències promotores.
El resultat obtingut per mitjà del programa Perl ha estat el que es mostra en el següent enllaç:
Posteriorment, s'han comprat els factors de transcripció obtinguts en fer l'anàlisi amb el PROMO, amb els resultats del programa perl per a les dues proteïnes, PML i RARA. Els resultats es mostren en les següents taules. Els factors de transcripció encerclats en vermell són els factors de transcripció obtinguts pel PROMO que coincidèixen amb els obtinguts pel programa perl:
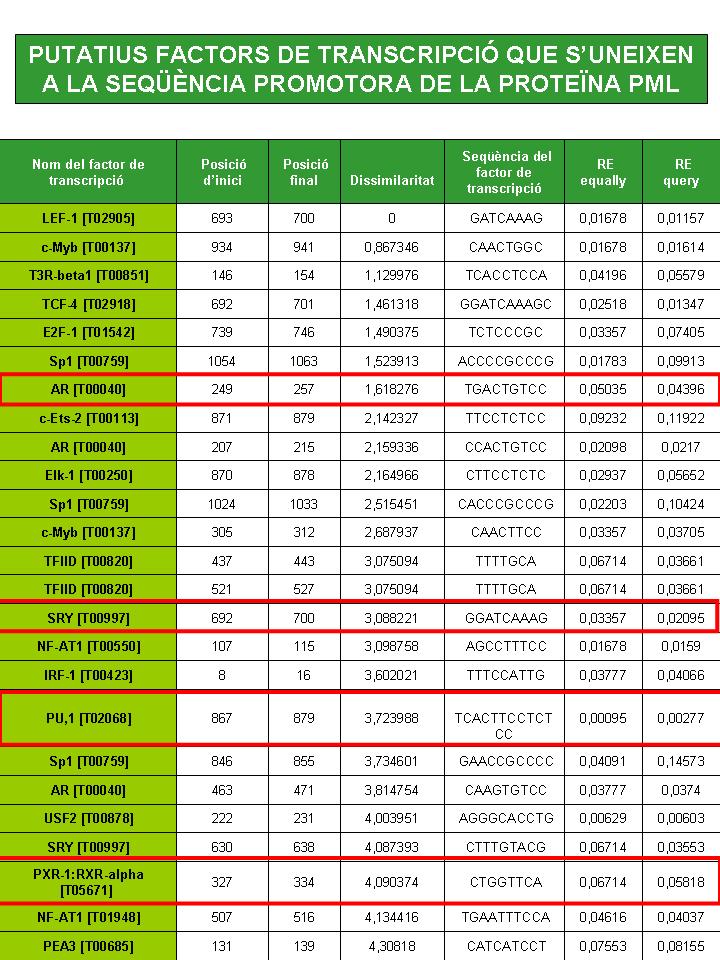
Aquests resultats es comenten més detalladament a l'apartat de Discussió.
5. Funció del gen
Un cop dut a terme l'estudi de la proteïna i els gens que la integren, s'ha analitzat la funció que aquesta té en Homo sapiens i en quins processos cel·lulars, funcions biològiques i components cel·lulars es troba aquesta proteïna de fusió. Per això, s'ha consultat la base de dades de Gene Ontology 7 i articles relacionats, per a les dues proteïnes inicials i després per a PML-RAR.
a) PML (GO ID: PML_HUMAN)
Les referències que s'han trobat per PML a la pàgina de Gene Ontology són les següents:| Identificador GO | Ontologia | Terme | Referència | Funció |
| GO:0005626 | Component cel·lular | Fracció insoluble | PMID:12915590 | Relocalització nuclear com a resposta a l'estrés cel·lular o dany del DNA |
| GO:0005730 | Component cel·lular | Nuclèol | PMID:15195100 | Regulació estabilitat p53 al nuclèol |
| GO:0005654 | Component cel·lular | Nucleoplasma | PMID:12915590 | Relocalització nuclear com a resposta a l'estrés cel·lular o dany del DNA |
| GO:0016605 | Component cel·lular | Cos PML | PMID:9448006 PMID:10910364 |
Forma complexes estables amb la proteïna retinoblastoma Regula l'acetilació de p53 i la senescència prematura induïda per Ras oncogènic |
| GO:0005515 | Funció molecular | Unió a proteïnes | PMID:14976184 PMID:15626733 PMID:15626733 |
Interacció amb MEF (Myeloid Elf-1-like factor) en els cossos nuclears PML, activant MEF que resulta en una up-regulation de la lisozima endògena Regula la funció de c-Jun en resposta al dany del DNA Inhibeix la traducció de HIF-1α i la neoangiogenesi mitjançant la repressió de mTOR |
| GO:0003713 | Procés biològic | Coactivador transcripcional | UniProt:P29590 | Entre diferents processos, PML regula selectivament la transcripció de factors proapoptotics segons diferents tipus de dany del DNA |
| GO:0006977 | Procés biològic | Resposta al dany del DNA i mediador del senyal de transdució de p53 resultant en aturada del cicle cel·lular i apoptosi | UniProt:P29590 | Entre diferents processos, PML regula selectivament la transcripció de factors proapoptotics segons diferents tipus de dany del DNA |
| GO:0016525 | Procés biològic | Regulació negativa de l'angiogènesi | PMID:16915281 | Inhibeix la traducció de HIF-1α i la neoangiogenesi mitjançant la repressió de mTOR |
| GO:0030308 | Procés biològic | Regulació negativa del creixement cel·lular | PMID:9448006 | Forma complexes estables amb la proteïna retinoblastoma |
| GO:0016481 | Procés biològic | Regulació negativa de la transcripció | PMID:9448006 | Forma complexes estables amb la proteïna retinoblastoma |
| GO:0006461 | Procés biològic | Ensemblatge de complexes proteics | PMID:12915590 | Relocalització nuclear com a resposta a l'estrés cel·lular o dany del DNA |
| GO:0001666 | Procés biològic | Resposta a la hipòxia | PMID:16915281 | Inhibeix la traducció de HIF-1α i la neoangiogenesi mitjançant la repressió de mTOR |
A partir d'aquestes dades ja es poden saber quines poden ser les funcions de la proteïna PML. Tot i així s'han buscat articles publicats (font: NCBI)8 sobre PML, per tal d'ampliar aquesta informació. Així doncs s'ha pogut saber que:
El gen de la leucèmia promielocítica (PML) és un gen supressor de tumors, que es troba inactivat en la leucèmia promielocítica aguda (APL) i que regula l'apoptosi induïda per dany del DNA. Tot i així, els mecanismes moleculars pels quals PML modula l'apoptosi seguida de l'estrés genotoxic són parcialment coneguts.
D'altra banda, si que s'ha pogut confirmar que PML és essencial per la inducció de la mort cel·lular programada depenent de p53 per irradiació gamma, mitjançant el control de l'acetilació de p53 mediada pels cossos nuclears PML.PML també rep el nom de Tripartite motif-containing protein 19 ring finger protein 71 perquè conté certs dominis importants per la seva funció, com és el domini Zinc finger de tipus anell.
Sembla que PML té un papaer important en el reclutament de ELF-4 en els cossos nuclears PML. A més, interacciona amb SIRT1, TOPBP1, TRIM27 i TRIM69, així com l'extrem C-terminal d'ELF-4.
- Interacciona amb la proteïna Z del virus Lassa.
Segons la base de dades de InterPro10 els principals dominis funcionals que conté la proteïna PML són:
| InterPro ID | Domini |
| IPR001841 | Zinc finger tipus anell |
| IPR000694 | Regió rica en prolines |
| IPR00315 | Zinc finger B-box |
Finalment, també s'ha consultat a la base de dades d'Ensembl quins altres dominis té PML segons altres bases de dades. L'esquema adjunt mostra el que s'ha trobat és el següent:
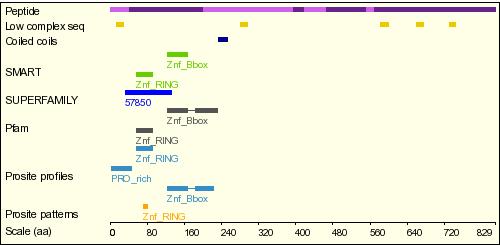
Figura 14. Dominis de la proteïna PML trobats en la base de dades d'Ensembl
b) RARA (GO ID: RARA_HUMAN)
Les referències que s'han trobat per RARA a la pàgina de Gene Ontology són les següents:| Identificador GO | Ontologia | Terme | Referència | Funció |
| GO:0005515 | Funció molecular | Unió a proteïnes | PMID:16432238 | Quan forma la proteïna de fusió amb PML, recluta diferents proteïnes com MBD1 i HDAC3 |
| GO:0003708 | Funció molecular | Receptor d'àcid retinoic | PMID:2825025 | Factor enhancer transactiu induïble per àcid retinoic, que té un mecanisme molecular semblant a la família de receptors nuclears implicats en els efectes dels retinoides (vitamina A) en el desenvolupament embrionari, i la diferenciació i creixement de cèl·lules tumorals |
| GO:0003713 | Funció molecular | Coactivador transcripcional | PMID:2825025 | Factor enhancer transactiu induïble per àcid retinoic, que té un mecanisme molecular semblant a la família de receptors nuclears implicats en els efectes dels retinoides (vitamina A) en el desenvolupament embrionari, i la diferenciació i creixement de cèl·lules tumorals |
| GO:0003700 | Funció molecular | Factor transcripció | PMID:2825025 | Factor enhancer transactiu induïble per àcid retinoic, que té un mecanisme molecular semblant a la família de receptors nuclears implicats en els efectes dels retinoides (vitamina A) en el desenvolupament embrionari, i la diferenciació i creixement de cèl·lules tumorals |
| GO:0007165 | Funció molecular | Transducció de senyals | PMID:2825025 | Factor enhancer transactiu induïble per àcid retinoic, que té un mecanisme molecular semblant a la família de receptors nuclears implicats en els efectes dels retinoides (vitamina A) en el desenvolupament embrionari, i la diferenciació i creixement de cèl·lules tumorals |
En aquest cas, cal recalcar que a la base de dades d'Ensembl apareixen altres codis de Gene Ontology, però s'ha decidit només centrar-se en el que es pot trobar a la pàgina en si de GO. Entre alguns dels GO que s'han trobat, els quals no es comentaran en detall, es poden destacar:
- Formació de la gemma uretèrica
- Unió a DNA
- Activitat de receptor nuclear dependent de lligand
- Receptor d'hormones estereoidees
- Unió a ions de zinc
- Regulació negativa de la transcripció
- Unió a una seqüència específica de DNA
- Regulació positiva de la transcripció del promotor de la RNA polimerasa II
- Unió a ions metall
A partir d'aquestes dades ja es pot saber quines són les funcions de la proteïna RARA. Tot i així s'han buscat articles publicats (font:NCBI)9 sobre RARA, per tal d'ampliar aquesta informació. Així doncs s'ha pogut saber que:
És un receptor (que es localitza a nivell nuclear) per àcid retinoic, que controla les funcions cel·lulars per la regulació directa de l'expressió dels gens. L'àcid retinoic és un metabòlit amb efectes importants sobre el desenvolpuament en vertebrats, ja que és un morfògen i un potent teratògen.
A part de l'aberració cromosòmica objecte d'aquest estudi computacional que dóna lloc l'APL, la proteïna RARA també està implicada en altres aberracions que donen lloc a leucèmies: una translocació t(11;17)(q32;q21) amb ZBTB16/PLZF, t(11;17)(q13;q12) amb NuMA, t(11;17)(q23;q12) / M5 amb MLL o t(5;17)(q32;q11) amb NPM.
Després d'unir-se al receptor, la proteïna RARA s'uneix amb alta afinitat com a heterodímer amb RXR (proteïna del receptor de retinoide X). Aquest heterodímer s'uneix al domini RARE (elements de resposta a l'àcid retinoic), una seqüència de DNA comuna en un cert nombre de gens i localitzada en el seu promotor.
- La resposta dels gens provocada per la unió de RARA està modulada per una sèrie de co-repressors i co-activadors.
Segons la base de dades de InterPro10 els principals dominis funcionals que conté la proteïna RARA són:
| InterPro ID | Domini |
| IPR001628 | Unió al DNA, receptor hormonal nuclear |
| IPR000536 | Unió al lligand, receptor hormonal nuclear |
| IPR001723 | Receptor hormones esteroidees |
| IPR000003 | Receptor retinoide X |
| IPR003070 | Receptor nuclear orfe |
| IPR003078 | Receptor àcid retinoic |
| IPR003074 | Receptor peroxisomes activat per proloferació |
| IPR00324 | Receptor vitamina D |
Finalment, també s'ha consultat a la base de dades d'Ensembl quins altres dominis té RARA segons altres bases de dades. L'esquema adjunt mostra el que s'ha trobat és el següent:
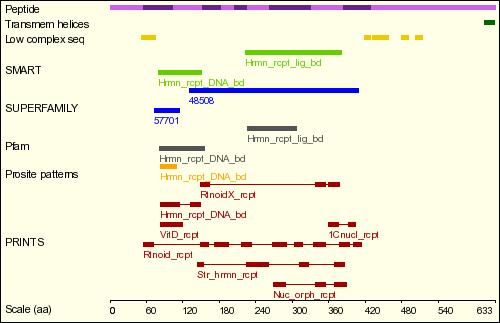
Figura 15. Dominis de la proteïna RARA trobats a la base de dades d'Ensembl
c) PML-RAR (GO ID: Q15156_HUMAN)
Les referències que s'han trobat per a la proteïna de fusió PML-RAR a la pàgina de Gene Ontology són les següents:| Identificador GO | Ontologia | Terme | Referència | Funció |
| GO:0005634 | Component cel·lular | Nucli | UniProt:Q15156 | Receptor d'acid retinoic i similar als de la família de receptors hormonals nuclears |
| GO:0003700 | Funció molecular | Factor de transcripció | PMID:1652368 | La proteïna PML-RAR no lligada s'ha considerat una nova classe de producte oncogènic dominant negatiu. |
| GO:0006355 | Procés biològic | Regulació de la transcripció DNA dependent | UniProt:Q15156 | La proteïna PML-RAR no lligada s'ha considerat una nova classe de producte oncogènic dominant negatiu. |
La proteïna de fusió de 106 kDa PML-RAR causa la Leucèmia Promielocítica Aguda (APL), ja que actua bloquejant la resposta del receptor d'àcid retinoic (RA), inhibint la diferenciació cel·lular. En el pacient aquesta malaltia es visualitza com un problema en la coagulació sanguínia.
Es pensa que PML-RAR causa leucèmia per interferència amb una via de senyalització. Això dóna lloc a la diferenciació i maduració de precursors mieloides, principalment per la disregulació dels gens induibles per retinoides implicats en la diferenciació mieloide. De fet, va ser el primer exemple d'una oncoproteïna generada per la fusió de dues molècules implicades en una mateixa via; concretament per la fusió d'un factor de transcripció amb un dels seus cofactors.
A nivell més concret, la translocació t(15;17), que es troba en cèl·lules leucèmiques de pacients amb APL produeix un únic mRNA codificat pel Receptor d'Àcid Retinoic Alpha (RARA) i la proteïna producte d'un gen mieloide anomenada PML. PML conté una regió rica en cisteïnes present en una nova família de proteïnes d'unió al DNA, que inclou un regulador del gen del Receptor d'Interleuquina-2 (Rpt-1) i el producte del gen activador de la recombinació (RAG-1). Segons aquest fet, PML representa un nou factor de transcripció o recombinasa.
També s'ha publicat que PML-RARα recluta el complex corepressor nuclear-histona deacetilasa (NCOR), a través de la CoR-box de RARA; donant lloc a una inhibició de la transcripció que resulta en un bloqueig de la diferenciació hematopoiètica.
D'altra banda, els homodímers PML-RARA tenen una alta eficàcia d'unió a corepressors i efectes inhibitoris sobre les respostes hormonals de la diferenciació mieloide. La proteïna de fusió també indueix la hipermetilació i el silenciament de gens, mitjançant el reclutament en promotors target de metiltransferases del DNA.
El producte de fusió PML-RAR, tot i respondre a àcid retinoic (RA), presenta diferències amb la forma wild-type RARα, en quant a tipus cel·lular i promotor. Com que els pacients amb APL poden ser tractats amb teràpies amb altes dosis d'àcid retinoic, s'infereix que PML-RAR no lligat és una nova classe de producte oncogènic dominant negatiu. L'àcid retinoic s'usa com a tractament per a l'APL, ja que té la capacitat desestabilitzar els complexes corepressors de PML-RAR. El tractament amb RA no només alleuja la inhibició, sinó que també fa que la proteïna activada PML-RAR pot promoure la diferenciació mielocítica.
A continuació s'ha realitzat un resum de tota la informació trobada en les diferents fonts:
L'expressió del gen PML-RARa produeix una proteïna que conté els dominis de dimerizació i d'unió al DNA del gen PML natiu, i els dominis d'unió al DNA i a altres lligands del receptor a RARA. Aquesta proteïna mostra un efecte dramàtic sobre l'arquitectura nuclear del promielocit, produïnt el trencament dels anomenats cossos nucleares de la PML, que són components estructurals crítics. Això provoca que la maduració del promielocit quedi bloquejada en l'estadio de promielocit i no pugui seguir la cadena de diferenciació mieloide. D'altra banda, la desorganizació dels cossos nucleares juga un paper fonamental en la patogènesi de la leucèmia promielocítica aguda, al inhibir l'apoptosi cel·lular. La presència de receptors de l'àcid a-trans-retinoic en les proteïnes híbrides explica que aquest fàrmac administrat terapèuticament, indueixi el pas de promielocit a mielocit, ocasionant un gran nombre de remissions en pacients amb APL.
Un dels més importants avenços en el coneixement de la patogènesi i del tractament de la leucèmia promielocítica aguda ha estat el poder aclarar la paradoxa aparent de que la destrucció de RARA provocada per la translocació PML-RARA bloqueja la diferenciacó del promielocit, mentre que l'administració del lligand anomenat RARA (Àcid retinoic en forma d'àcid t-retinoic o tretinoïna) permet la diferenciació de les cèl·les leucèmiques.
El receptor d'àcid retinoic posseeix varies regions funcionals que regulen la unió al DNA, la interacció amb varis co-activadors i co-repressors i els receptors per a varis lligands, entre ells l'àcid retinoic. L'anomenat complex de repressió trancripcional té la capacitat de modificar la cromatina en les cèl·lules normales impedint la transcripció, però hi ha una concentració fisiològica d'àcid retinoic suficient com per desplaçar el complex en si. En canvi, en el cas de les persones que pateixen APL, els promielocits leucèmics mostren una major quantitat de complex de repressió, degut a l'expressió del receptor procedent de la translocació. Per tant, seran necessàaries concentracions farmacològiques d'àcid retinoic per permetre l'activació de la transcripció.
La imatge següent mostra la formació del complex de repressió: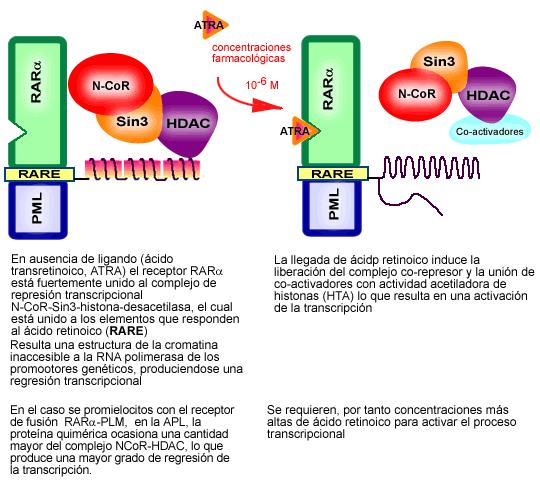
Figura 16. Esquema resum del funcionament del complex
Mètodes
Per tal de desenvolupar l'estudi de la proteïna PML-RARA, per a cadascun dels apartats s'han seguit els passos que es detallen a continuació:
A partir de la seqüència de la proteïna que se'ns va facilitar, es realitzar un BLASTP (proteïna-proteïna) a partir de la base de dades del genoma de NCBI. Aquest va consistir en buscar en tot el genoma humà aquelles seqüències proteiques amb un major percentatge de similitud amb la seqüència donada. Finalment, es va seleccionar la proteïna que presentava un valor de score més elevat per l'alineament amb la seqüència (veure apartat de Resultats), i així es va poder determinar de quina proteïna es tractava.
Per a trobar la informació referent a l'estructura genòmica dels gens s'ha utilitzat la base de dades de Ensembl, ja que va resultar més senzill extreure i interpretar la informació continguda, respecte les altres bases de dades com NCBI o UCSC. D'altra banda, en mirar les altres bases de dades, es van trobar certes diferències en quant a seqüències o isoformes, fet que no ha permetia establir un consens en l'informació. Per això, es va decidir fer servir només les dades provinents de la base de dades d'Ensembl.
Des d'aquesta base de dades es va poder accedir a la base de dades UniProt, ha permès l'obtenció de la informació i seqüències referents a les isoformes.Cal destacar també, el programa ClustalW que ha permès realitzar els diferents alineaments dels transcrits amb les diferents isoformes obtingudes. Aquest programa ha permès determinar la relació existen entre transcrits i isoformes i així estimar si es manté o no la pauta de lectura.
Pel que fa a l'estudi de l'homologia en altres espècies, la informació obtinguda s'ha extret de la base de dades de l'Ensembl. A partir d'aquesta, per l'opcio Export Gene Data s'ha pogut aconseguir les seqüències de DNA de cada una de les espècies i amb el programa ClustalW realitzar un alineament a partir del qual s'ha elaborartels arbres filogenètics. Cal citar també, que per tal de buscar la possible existència de més ortòlegs que no apareixin en l'Ensembl, s'ha realitzat per mitjà de 'NCBI un BLASTN.
El següent punt ha estat l'estudi de l'expressió de cada un dels gens que conformen la proteïna de fusió. Per a fer aquest anàlisi s'ha buscar informació a partir de les bases de dades de Genome Browser de l'UCSC i el Gene Sorter també de l'UCSC. Per altra banda, la cerca d'nformació també ha inclòs altres bases de dades com Gene-Hub Gesip i Gene Expressiom Atlas
Pel que fa a la caracterització de les regions promotores s'ha utilitzat, tal i com ja s'ha explicat en el corresponent apartat, el programa PROMO, per mitjà de l'opció Select Species i Search Sites (dissimilaritat del 10% i del 15%). Aquest ha permès obtenir els putatius factors de transcripció que s'uneixin a les seqüències promotores per a cada un dels gens. Gràcies al programa Microsoft Excel s'han ordenat els diferents factors de transcripció obtinguts de menor a major RE-equally, i s'ha acotat la búsqueda a només aquells factors amb un RE-equally inferior a 0,9.
El pas següent ha estat comparar aquests resultats amb els obtinguts amb el programa perl.Finalment, s'ha realitzat l'estudi de la funcionalitat de cada un dels gens que conformen la proteïna PML-RAR. Per tal de desenvolupar aquest apartat s'ha utilitzat la base de dades de Gene Ontology i altres fonts d'informació provinents d'articles (de la base de dades de l'NCBI) que es mostren en l'apartat de Referències.
Discussió
Després d'analitzar la informació obtinguda de les diferents bases de dades i d'analitzar els resultats anteriors, en aquest apartat es donarà una visió integradora i global dels principals conceptes a destacar per a la proteïna de fusió estudiada PML-RAR, intenant relacionar la informació obtinguda en els diferents apartats.
La proteïna de fusió PML-RAR s'origina per una translocació cromosòmica dels cromosomes 15 i 17, generant-se un transcrit 5'PML-3'RARA. Pel que fa a l'estructura genòmica dels gens per separat, tot i que els 4 transcrits de PML només presenten els 4 primers exons en comü, aquest fet no afecta a la formació de la proteïna de fusió, ja que aquesta només conté 3 exons provinents de PML. D'altra banda, en fer l'estudi dels dominis de la proteïna PML, s'ha vist que tots els dominis funcionals de la proteïna es troben a la part inicial de la proteïna (corresponent als primers 320 aa, aproximadament). Per tant, es pot concloure que al funcionalitat de PML resideix en els seus primers exons i, per això, són els que es troben conservats; tant a nivell dels 4 transcrits com formant part de la proteïna de fusió. Aquest fet també es veu reflexat en quant a les isoformes de PML, ja que aquestes també presenten en comú els 6 primers exons.
El que es pot destacar de la proteïna RARA, de la mateixa manera que passava amb PML, tot i que el nombre d'exons codificants en cadascun dels transcrits és diferent, aquest fet no interfereix en la formació de la proteïna de fusió; doncs els que consistueixen PML-RAR són comuns en ambdós transcrits. De les dues isoformes que té la proteïna RARA s'ha observat que una d'elles és igual a un dels transcrits i s'ha cregut que ve codificada per aquest, sense que s'hagi modificat la pauta de lectura. En canvi, la segona isoforma difereix en alguns aminoàcids de certs exons respecte la seqüència del segon transcrit, sent més curta que el segon transcrit, fet que permet concloure que en aquest cas si s'ha produït un trencament de la pauta de lectura.
De l'estudi dels ortòlegs en altres espècies, s'ha observat que per al gen que codifica per la proteïna PML, tant els percentatges obtinguts des d'Ensembl com els obtinguts en fer el BLAST des de NCBI, els més elevats en quant a homologia es corresponen amb aquelles espècies que formen part de l'ordre dels Primats. Aquest és un fet esperable, ja que es coneix que aquests organismes són els evolutivament més propers a Homo sapiens. Pel que fa al gen que codifica per RARA, els resultats obtinguts mostren també que les espècies de l'ordre dels Primats presenten un elevat percentatge d'homologia amb RARA d'Homo sapiens. Cal destacar també que les espècies de l'ordre dels Rossegadors són després del Primats, espècies evolutivament més properes als humans, per aquest gen.
Fixant-nos en els resultats obtinguts per aquest anàlisi de l'expressió de les proteïnes PML i RARA, s'ha vist que els majors nivells d'expressió de la proteïna PML es troben en la melsa, el teixit muscular i el fetge de l'individu adult. Per altra banda, la proteïna RARA s'expressa principalment en cor, sistema vascular, melsa, ovaris i pulmons. En conclusió per tant, es pot veure com ambdues proteïnes es troben clarament sobreexpressades òrgans relacionats amb la sang. Concretament, la proteïna PML presenta una major expressió en òrgans com la melsa i la proteïna RARA en el propi teixit sanguini. Basant-nos en aquest fet, és fàcil d'entendre que la translocació d'aquestes proteïnes per a la formació de la proteïna quimèrica o de fusió PML-RARA, comporti una alteració relacionada amb la el sistema sanguini; la Leucèmia Promielocítica Aguda.
Després de realitzar l'anàlisi de la caracterització de les regions promotores, s'ha vist que coincideixen, per ambdos proteïnes, diversos factors de transcripció dels obtinguts en el programa PROMO i amb el programa perl.
Per a la proteïna PML s'ha vist que coincideixen 3 factors de transcripció: AR[T00040], PU,1[T02068], PXR-1:RXR-alpha[T05671]. Aquests factors de transcripció apareixen (en els resultats del programa perl) com a putatius factors de transcripció que podrien unir-se a la seqüència promotora de la proteïna PML. Malgrat aquest fet, cal dir que les seqüències d'unió del factor que ens facilita el programa perl, no coincideixen amb les seqüències que ens dóna el PROMO. Aquest fet podria ser degut a que les matrius del PROMO i les que ens han estat facilitades per fer el programa perl són diferents.
En aquest anàlisi, és important destacar els valors de RE-equally i p-value dels factors seleccionats. Tenint en compte que el RE-equally és la probabilitat de trobar una seqüència igual a la nostra per atzar i que el p-value és la probabilitat d'error per unió de la seqüència per atzar, ens interessa trobar aquells factors que presenten aquests paràmetres el més baix possible.Els valors d'aquests paràmetres obtinguts per als putatius factors de transcripció de la proteïna PML són:
- AR[T00040]:RE-equally de 0,05035, p-value de 0,12
- PU,1[T02068]:RE-equally de 0,00095, p-value de 0,71
- PXR-1:RXR-alpha[T05671]:RE-equally de 0,0674, p-value de 0
En tots aquests factors transcripció, es pot observar valors de p-value i RE-equally prou baixos com per confirmar que aquests es podrien unir amb alta probabilitat a la seqüència promotora de PML.
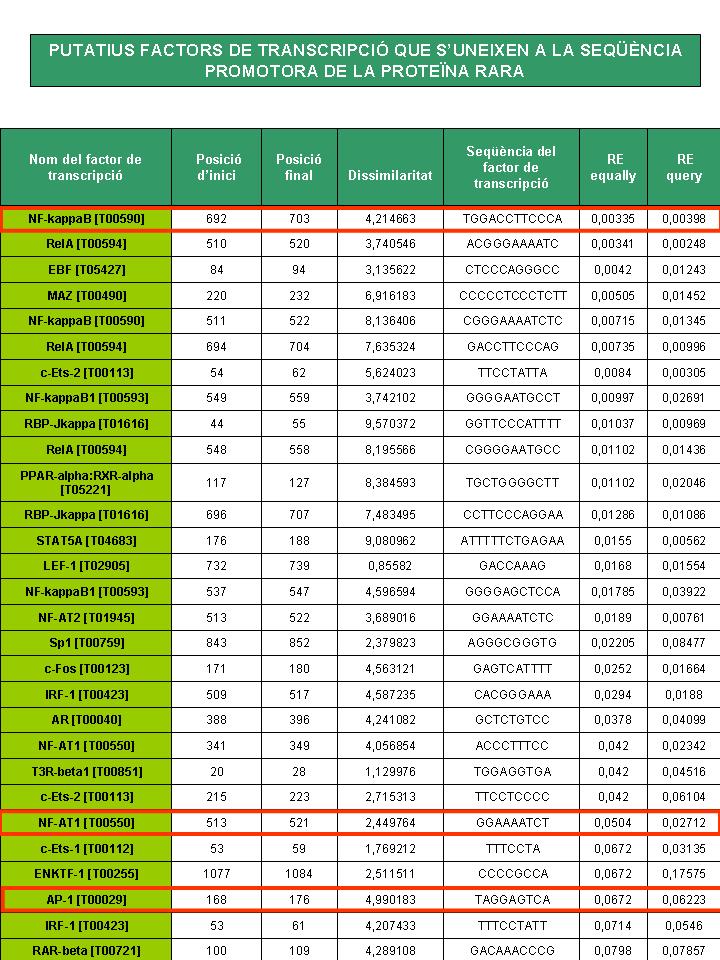
Paral·lelament a aquest fet, s'han analitzat també els putatius factors de transcripció que s'uniexen a la seqüència promotora del gen de la proteïna RARA, obtinguts de l'anàlisi amb el PROMO i amb el programa perl.
Els factors coincidents han estat 3: NF-KappaB[T00590], NF-AT1[T00550], i AP-1[T00029].
És molt important destacar que per a aquesta seqüència promotora, els putatius factors de transcripció obtinguts amb el PROMO i amb el programa perl sí que coincideix les seves seqüències de nucleòtids. Aquest fet doncs, fa pensar que aquests factors han de presentar valors de p-value i RE-equally baixos. Si analitzem els resultats obtinguts, podem concloure que efectivament aquest fet és així:
- NF-KappaB[T00590]:RE-equally de 0'00335, p-value de 0'24, seqüència: TGGACCTTCCCA
- NF-AT1[T00550]:RE-equally de 0'0504, p-value de 0'01, seqüència: GGAAATCT
- AP-1[T00029]:RE-equally de 0'0672, p-value de 0'08, seqüència: TAGGAGTCA
Tal i com mostren aquests resultats, els valors de p-value i RE-equally són prou baixos com per confirmar que aquests factors sí que s'uneixen a la seqüència promotora de la proteïna RARA.
Cal destacar, per altra banda, que el factor RXR-alpha, tot i no ser un dels factors de transcripció coincidents, també apareixia a la llista inicial obtinguda a PROMO. A més, com ja s'ha comentat, RXR-alpha é una proteïna del receptor de retinoide X que forma un heterodímer d'alta afinitat amb RARA.
A nivell de funció proteica, destacar que les funcions que s'han trobat segons Gene Ontology i llegint els articles són:
PML: Regula l'estabilitat de p53 al nuclèol i participa en processos de resposta a l'estrés cel·lular i apoptosi, és un coactivador transcripcional i s'uneix a diferents proteïnes, donant lloc a la formació de complexes. El domini més important que posseix és el Zinc finger tipus anell, que media les interaccions proteïna-proteïna. Com ja s'ha comentat anteriorment, cal destacar que els dominis funcionals de PML es troben ubicats a la part inicial de la proteïna, on resideix la part funcional d'aquesta, que a la vegada forma part de la porteïna de fusió PML-RAR.
RARA: És un receptor per àcid retinoic, que també actua com a coactivador transcripcional, transductor de senyals i com a factor de transcripció. Exerceix una regulació positiva sobre la transcripció del promotor de la RNA polimerasa II. Té dominis d'unió al DNA i als lligands.
Pel que fa a la proteïna de fusió PML-RARA a la que dóna lloc la translocació cromosòmica, aquesta ve codificada pel transcrit 5'PML-3'RARA. Conté els dominis de dimerizació i d'unió al DNA del gen PML, i els dominis d'unió al DNA i a altres lligands del receptor a RARA. Aquesta proteïna actua a nivell de la destrucció dels cossos PML, fet que provoca el bloqueig de la maduració del premielocits. PML-RAR actua inhibint la diferenciació hematopoiètica (per això, PML i RARA són proteïnes que es troben expressades principalment a òrgans relacionats amb els sistema sanguini), ja que recluta el complex corepressor nuclear-histona deacetilasa, que fa que la cromatina sigui menys accessible per la RNA polimerasa, produint-se una regressió transcripcional. La manera de revertir aquest efecte, que és un dels tractaments per pacients amb APL, és mitjançant l'aplicació d'àcid retinoic (RA). El RA indueix l'alliberació del complex corepressor i la unió dels co-activadors amb activitat acetiladora d'histones (HTA), el que resulta en una activació de la transcripció.
Referències
Bases de dades:Articles NCBI
- Kakizuka A, et al.(1991). Chromosomal translocation t(15;17) in human acute promyelocytic leukemia fuses RAR alpha with a novel putative transcription factor, PML.Cell Press.66(4):663-74
- PML inducer of acute promyelocytic leukemia
- Lin RJ et al.(1998). Role of the histone deacetylase complex in acute promyelocytic leukaemia. Nature 391: 811-814.
- Grignani F et al.(1998) Fusion proteins of the retinoic acid receptor-alpha recruit histone deacetylase in promyelocytic leukaemia. Nature 391: 815-818.
- Grignani F et al.(1993). The acute promyelocytic leukemia-specific PML-RAR-alpha fusion protein inhibits differentiation and promotes survival of myeloid precursor cells. Cell 74: 423-431
- RARA retinoic acid receptor