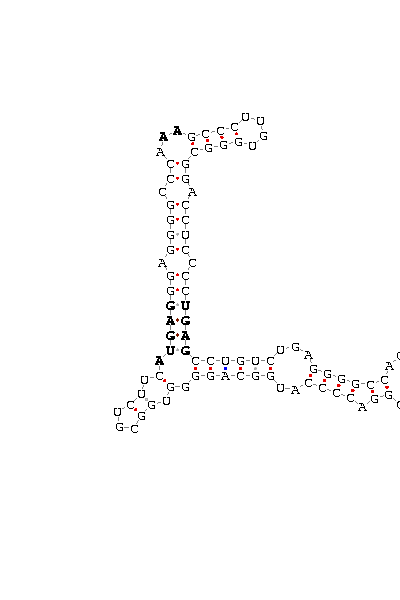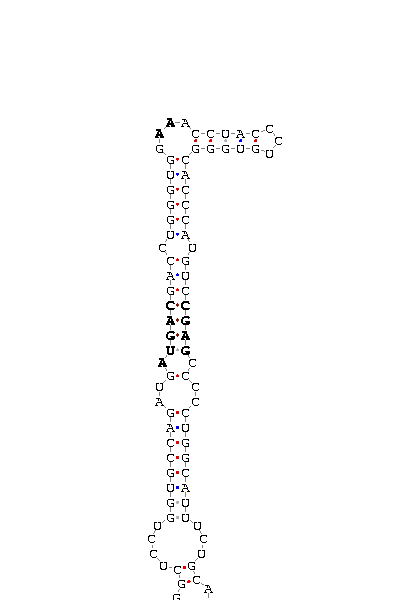De todas las selenoproteínas humanas estudiadas, no encontramos ningún SNP ni ninguna otra variación que afectara a un codón TGA o a uno similar cuyo polimorfismo pudiera dar lugar a este triplete. No obstante, este resultado concuerda con estudios anteriores realizados sobre este mismo tema.
Para poder estudiar el efecto de los polimorfismos sobre la estructura del elemento SECIS en la región 3'UTR, creamos un archivo para cada selenoproteína escogida que contuviera todas las posibles secuencias generadas a partir de la combinación de todos los polimorfismos detectados. El volumen de selenoproteínas estudiadas es notablemente inferior que al inicio del proyecto dado que las hemos ido descartando basándonos en los criterios descritos en Materiales y Métodos. Los archivos resultantes se muestran a continuación:
- Variaciones Selenoproteína de 15Kda (1er tránscrito)
- Variaciones Selenoproteína de 15Kda (2o tránscrito)
- Variaciones Selenoproteína GPx1
- Variaciones Selenoproteína GPx3 (1er tránscrito)
- Variaciones Selenoproteína GPx4
- Variaciones Selenoproteína P
- Variaciones Selenoproteína Z (3er tránscrito)
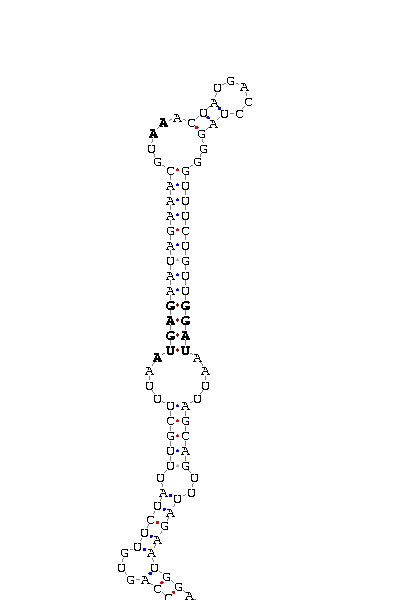
Cada una de las diferentes secuencias generadas por los polimorfismos estudiados fue analizada con ayuda del programa SECISearch para observar si estos SNPs afectaban a la estructura del elemento SECIS. El resultado de dicho análisis se muestra a continuación:
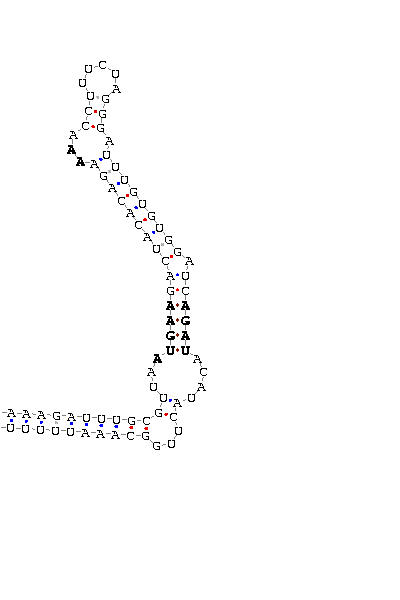
- Elemento SECIS de la selenoproteína de 15kda (1er tránscrito).
Las cuatro secuencias de esta proteína generadas a partir de los posibles polimorfismos dan lugar a un mismo elemento SECIS, que se ilustra en esta imagen.
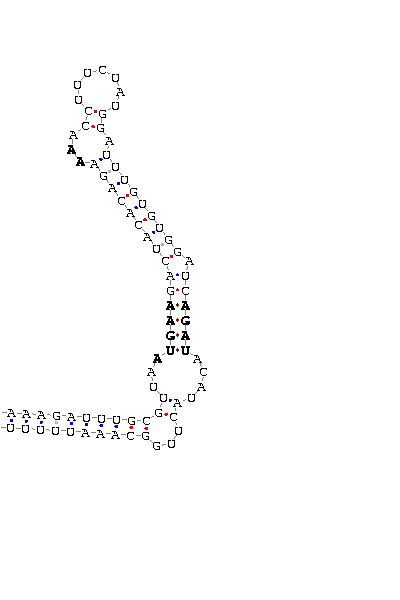
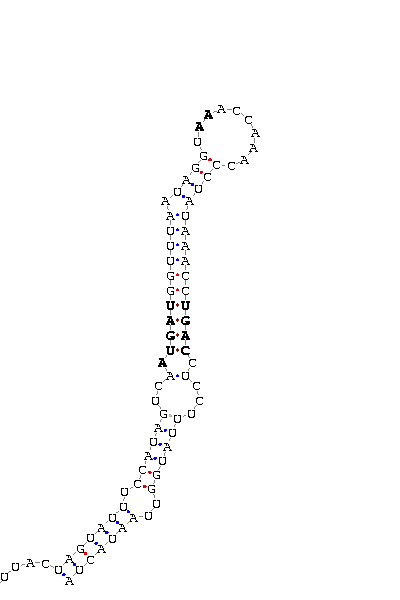
- Elemento SECIS de la selenoproteína de 15kda (2o tránscrito).
En este segundo tránscrito, las cuatro secuencias generadas dan lugar a un mismo elemento SECIS, que se ilustra en esta imagen.
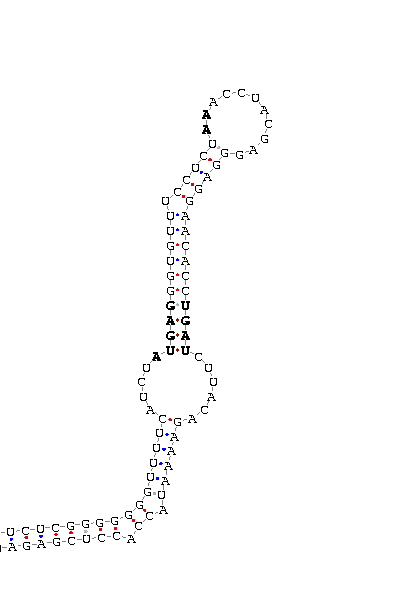
- Elemento SECIS de la selenoproteína GPx1.
En esta ocasión son dieciséis las diferentes secuencias generadas. En algunos casos, la posición del nucleótido donde empieza el SECIS predicho varía de una a tres unidades; de manera que el score asociado también oscila notablemente (pero siempre con un valor superior a 15, es decir, fiable). Sin embargo, estos cambios no afectan a la estructura del SECIS y por tanto, la figura de la imagen corresponde a todas las secuencias.
- Elemento SECIS de la selenoproteína GPx3 (1er tránscrito).
Las dieciséis secuencias de esta proteína generadas a partir de los posibles polimorfismos dan lugar a un mismo elemento SECIS, que se ilustra en esta imagen.
- Elemento SECIS de la selenoproteína GPx4.
Se generan dos secuencias a partir de los posibles polimorfismos y todas corresponden a la misma estructura de SECIS, que se ilustra en la imagen.
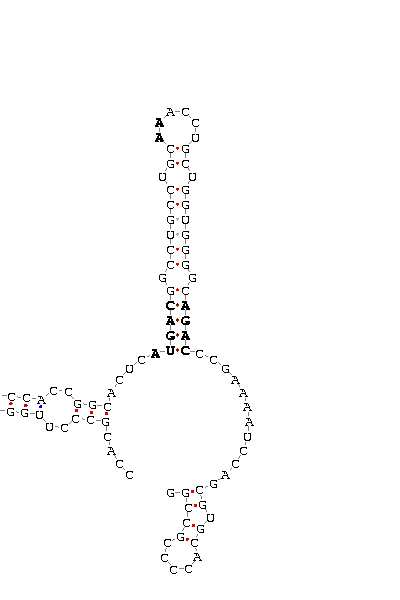
- Elemento SECIS de la selenoproteína SelP.
Se generan dos posibles elementos SECIS. El primero de ellos tiene un score de 30.80, mientras que el segundo de ellos tiene un score de 20.32. A pesar de esta puntuación, escogemos el segundo de ellos puesto que en la región del elemento SECIS predicho se encuentran tres posibles polimorfismos, cosa que no pasa con el primer SECIS. Considerando estos polimorfismos, se generan cuatro posibles secuencias, pero todas ellas dan lugar a los mismos elementos SECIS anteriormente comentados.
- Elemento SECIS de la selenoproteína SelZ.
Se generan dos secuencias diferentes a partir de los polimorfismos posibles que dan lugar a un elemento SECIS, que se ilustra en la imagen.
En esta etapa de nuestro estudio tan solo nos interesaban SNPs que afectaran a nuestros SECIS predichos por SECISearch. Pero como puede observarse, ninguno de los SNPs descritos afectan a la estructura del elemento SECIS del mRNA de las selenoproteínas humanas analizadas.
Otro punto interesante de este estudio es la comparación de los tres tránscritos de la selenoproteína SelZ o TR3. Como se ha comentado en otros apartados, mientras el tercer tránscrito sí que contenía Sec en su secuencia aminoacídica (aunque muy cerca del extremo 3'UTR), el primero y el segundo no la contenían, y además su codón de stop era el TGA. Este hecho nos llamó la atención, de manera que decidimos estudiarlo más a fondo. Tras analizarlo con diferentes bases de datos como Ensembl y Expasy, vimos que, de hecho, el cDNA de los tres tránscritos era muy similar, y en los alrededores del stop codon del primer y segundo tránscrito, los nucleótidos estaban unas pocas posiciones desplazados respecto al tercer tránscrito. Las similitudes entre las tres secuencias de los cDNAs de estos tres tránscritos se pueden corroborar en los enlaces que siguen: