Introducción
¿Qué son las selenoproteínas?
Las selenoproteínas son un grupo heterogéneo de proteínas que contienen Selenocisteína (Sec, U). La estructura de la selenocisteína es igual que la de la cisteína, cambiando el azufre por selenio. La incorporación de dicho aminoácido viene especificada por el codón TGA, que normalmente es un stop codon. Esta lectura alternativa del codón TGA está dirigida por la estructura secundaria que adopta una región del mRNA 3'UTR en los genes de las selenoproteínas, llamada elemento SECIS (Selenocysteine insertion sequence). Otros factores requeridos para la biosíntesis e inserción de Sec son Sec tRNA, Factor de elongación Sec-específico, Selenofosfato sintasa (SPS), Sec sintasa y SECIS-binding protein.
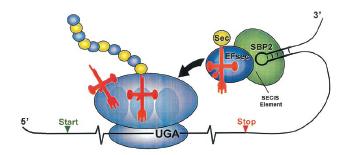 Mecanismo de inserción de Sec en eucariotas. Selenocisteil-tRNA (en rojo con la Sec marcada en amarillo) forma un complejo con EFSec (en azul), SBP2 (en verde) y el elemento SECIS (loop en negro) que se aproxima al bolsillo A del ribosoma y hace que el codón UGA del mRNA de las selenoproteínas sea traducido a Sec.
Mecanismo de inserción de Sec en eucariotas. Selenocisteil-tRNA (en rojo con la Sec marcada en amarillo) forma un complejo con EFSec (en azul), SBP2 (en verde) y el elemento SECIS (loop en negro) que se aproxima al bolsillo A del ribosoma y hace que el codón UGA del mRNA de las selenoproteínas sea traducido a Sec.
El problema que existe con la predicción de estas selenoproteínas es que los métodos informáticos corrientes de predicción de genes son incapaces de identificar el codón TGA que se traduce a Sec, ya que lo detectan como un codón de stop. Por esta razón, existen genes de selenoproteínas que no pueden ser identificados como tales, suponiendo una pérdida importante de información biológica y funcional.
El elemento SECIS forma un loop que contiene nucleótidos conservados y algunas bases apareadas que no siguen el modelo de Watson y Crick (ambos, importantes para su identificación mediante técnicas computacionales y para la formación de la estructura secundaria que le confiere la funcionalidad). Las secuencias de los elementos SECIS estudiados predicen que estos han divergido de una estructura consenso en la región del loop.
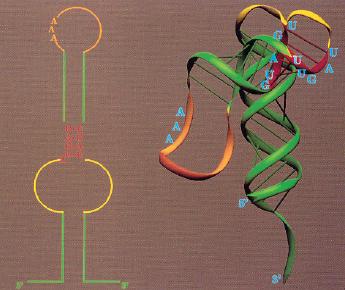 Estructura del elemento SECIS, esquemáticamente a la izquierda y en 3D a la derecha. En rojo se señalan las bases apareadas que no siguen el modelo de Watson y Crick, y en naranja los nucleótidos conservados del loop superior.
Estructura del elemento SECIS, esquemáticamente a la izquierda y en 3D a la derecha. En rojo se señalan las bases apareadas que no siguen el modelo de Watson y Crick, y en naranja los nucleótidos conservados del loop superior.
Hay dos tipos de elementos SECIS eucarióticos: forma 1 y forma 2, llamados así por el orden en que fueron descubiertos, aunque la forma 2 es más común que la 1. Los elementos de la forma 2 tienen estructuras secundarias adicionales que no presentan los elementos de la forma 1.
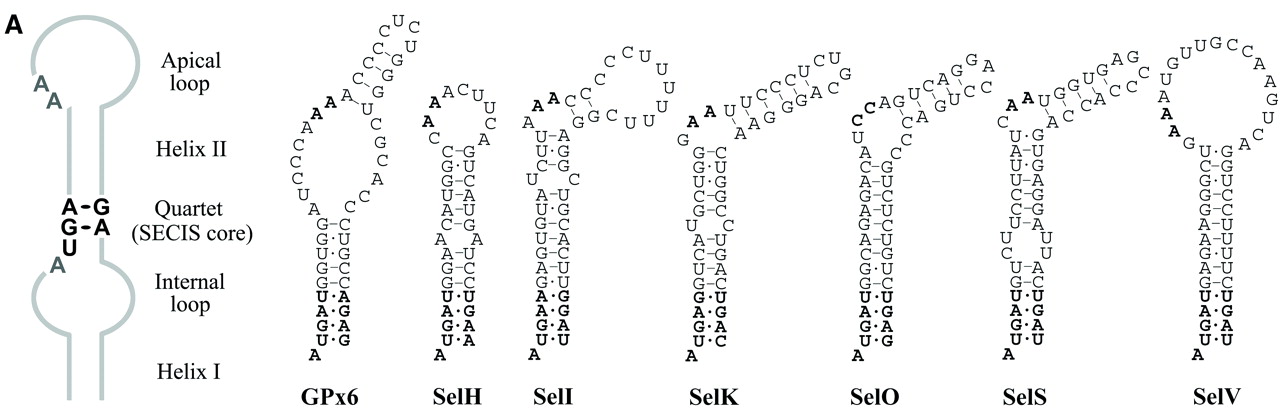 Estructura básica de SECIS y elementos SECIS de los genes de distintas selenoproteínas humanas. SelH, SelI y SelV son de la forma 1, mientras que GPx6 , SelK, SelO y SelS son de la forma 2.
Estructura básica de SECIS y elementos SECIS de los genes de distintas selenoproteínas humanas. SelH, SelI y SelV son de la forma 1, mientras que GPx6 , SelK, SelO y SelS son de la forma 2.
El selenoproteoma humano consiste en 25 selenoproteínas. Muchas de ellas contienen su residuo Sec localizado en el centro activo del enzima y son esenciales para su actividad.
| Nombre Selenoproteína |
Localización en el cromosoma |
Posición de la Sec en la proteína |
Enzima |
| 15kDa |
1p22.3 |
93 |
-Desconocido- |
| DI1 |
1p32.3 |
126 |
Deiodinasa tipo 1 |
| DI2 |
14q31.1 |
133 |
Deiodinasa tipo 2 |
| DI3 |
14q.32 |
144 |
Deiodinasa tipo 3 |
| GPx1 |
3p21.31 |
47 |
Glutatión peroxidasa 1 |
| GPx2 |
14q23.3 |
40 |
Glutatión peroxidasa 2 |
| GPx3 |
5q33.1 |
73 |
Glutatión peroxidasa 3 |
| GPx4 |
19p13.3 |
73 |
Glutatión peroxidasa 4 |
| GPx6 |
6p22.1 |
73 |
Glutatión peroxidasa 6.
Estructura globular |
| SelH |
11q12.1 |
44 |
Estructura globular |
| SelI |
2p23.3 |
387 |
-Desconocido- |
| SelK |
3p21.31 |
92 |
Proteína de membrana |
| SelM |
22q12.2 |
48 |
-Desconocido- |
| SelN |
1p36.11 |
428 |
-Desconocido- |
| SelO |
22q13.33 |
667 |
Estructura globular |
| SelP |
5p12 |
59, 300, 318, 330, 345, 352, 367, 369, 376, 378 |
-Desconocido- |
| SelR |
16p13.3 |
95 |
-Desconocido- |
| SelS |
15q26.3 |
188 |
Proteína de membrana |
| SPS2 |
? |
60 |
-Desconocido- |
| SelT |
3q24 |
36 |
-Desconocido- |
| TR1 |
3q24 |
36 |
Tioredoxin reductasa 1 |
| TR2 |
3q21.2 |
655 |
Testicular Receptor 2 |
| TR3 |
22q11.21 |
522 |
Tioredoxin reductasa 3 |
| SelV |
19q13.13 |
273 |
Estructura globular |
| SelW |
19q13.32 |
13 |
-Desconocido- |
La mayoría de los genes de selenoproteínas conocidos tienen homólogos, en los cuales la selenocisteína (Sec) es reemplazada por una cisteína (Cys). No obstante, la eficacia enzimática es mayor cuando se encuentra la selenocisteína en el centro activo. Los codones que codifican para cisteína son TGC y TGT.
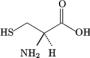
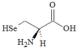
Estructura aminoacídica de la cisteína y la selenocisteína.
Hay muchos estudios que relacionan el Selenio con la prevención del cáncer, funciones del sistema inmune, fertilidad masculina, alteraciones musculares y cardiovasculares y el control sobre los procesos del envejecimiento. Se hipotetiza que las selenoproteínas son las responsables de la mayoría de estos efectos biomédicos del selenio, esenciales para los mamíferos.
La evolución del codón que codifica para la selenocisteína no está bien estudiada. Se encuentran selenoproteínas en los tres grandes reinos (eucariotas, eubacterias y arqueobacterias) pero tienen una distribución muy dispersa y, por ejemplo, no se encuentran ni en levaduras ni en plantas superiores.
Objetivos
La situación actual es que no se sabe nada respecto a la variación intraespecífica de los genes que codifican para las selenoproteínas. Son especialmente importantes tanto los polimorfismos que afecten al codón TGA que codifica para la Selenocisteína como los que se encuentren dentro de la secuencia del elemento SECIS. El objetivo de este proyecto es mapear los Single Nucleotide Polymorphisms que se encuentren dentro de la secuencia de genes que codifica para las selenoproteínas humanas e identificar aquellas que puedan afectar a su función.
El planteamiento inicial de este trabajo es analizar tres genes que codifiquen para selenoproteínas humanas, incluyendo el elemento SECIS e identificando los posibles SNPs que se encuentren en esta región. Tras analizar estas variaciones intraespecíficas comprobaremos si afectan a la codificación de Sec o bien a la estructura del elemento SECIS.


 Mecanismo de inserción de Sec en eucariotas. Selenocisteil-tRNA (en rojo con la Sec marcada en amarillo) forma un complejo con EFSec (en azul), SBP2 (en verde) y el elemento SECIS (loop en negro) que se aproxima al bolsillo A del ribosoma y hace que el codón UGA del mRNA de las selenoproteínas sea traducido a Sec.
Mecanismo de inserción de Sec en eucariotas. Selenocisteil-tRNA (en rojo con la Sec marcada en amarillo) forma un complejo con EFSec (en azul), SBP2 (en verde) y el elemento SECIS (loop en negro) que se aproxima al bolsillo A del ribosoma y hace que el codón UGA del mRNA de las selenoproteínas sea traducido a Sec.
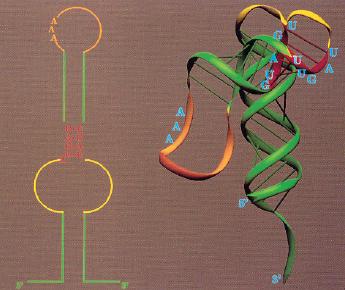 Estructura del elemento SECIS, esquemáticamente a la izquierda y en 3D a la derecha. En rojo se señalan las bases apareadas que no siguen el modelo de Watson y Crick, y en naranja los nucleótidos conservados del loop superior.
Estructura del elemento SECIS, esquemáticamente a la izquierda y en 3D a la derecha. En rojo se señalan las bases apareadas que no siguen el modelo de Watson y Crick, y en naranja los nucleótidos conservados del loop superior. Estructura básica de SECIS y elementos SECIS de los genes de distintas selenoproteínas humanas. SelH, SelI y SelV son de la forma 1, mientras que GPx6 , SelK, SelO y SelS son de la forma 2.
Estructura básica de SECIS y elementos SECIS de los genes de distintas selenoproteínas humanas. SelH, SelI y SelV son de la forma 1, mientras que GPx6 , SelK, SelO y SelS son de la forma 2.