Hemos corrido en nuestro programa el fichero "hs_EPD.fasta" correspondiente a varias secuencias de genes humanos desde la posición -674 respecto al inicio de transcripción (TSS) hasta la 375 (secuencias que corresponden por tanto al promotor proximal).
El programa indica que le hemos introducido 1713 secuencias y ha encontrado el motivo de unión a CREB (TGACGTGA) en 71 de ellas.
Esto supone que el programa ha encontrado posibles dianas de unión de CREB en el 4' 14 % de las secuencias introducidas.
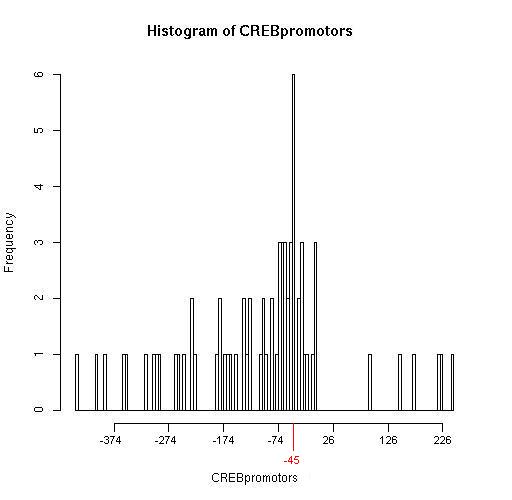
En aproximadamente 6 de las 71 secuencias en las cuales hemos encontrado la secuencia consenso TGACGTGA ésta se situa 45 pares de bases por encima del inicio de la transcripción (-45) como se observa en el histograma.
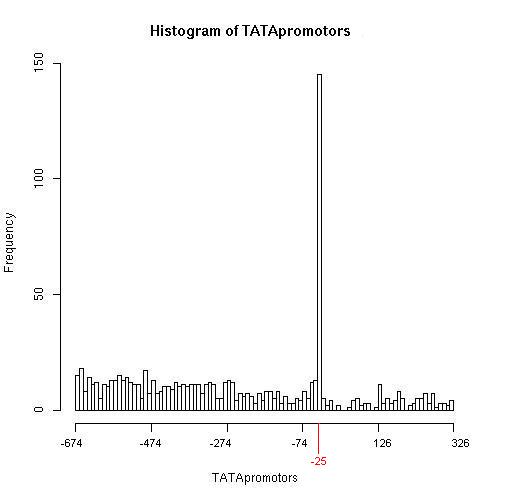
Asi, según nuestro programa las dianas del factor de transcripción CREB se situarían principalmente a distancias entre -100 y 0 respecto el TSS. Esto se corresponde con lo esperado según los artículos mencionados en la bibliografía.
En esta "figura" extraida del artículo "Genome..." se muestra la distribución de dianas de CREB conservadas en raton y humano y su localización se da principalmente entorno al 5'UTR.
También vemos una localización bastante similar de los motivos de unión de CREB y la caja TATA próximos al TSS. Esta proximidad no tiene porque suponer una localización conjunta en las regiones promotoras, puede tratarse simplemente de una posición preferente sobre la que actuarían CREB y otros factores de transcripción en la regulación de la expresión de diferentes genes, es posible encontrar en una secuencia solo el motivo de unión a CREB, solo el de TATA o que estén ambos o ninguno.
Volviendo a la información del artículo sabemos que existe una funcionalidad para la colocalización que se da en un 15% de casos, ya que la activación de CREB en respuesta a un aumento de los niveles intracelulares de AMPc va acompañada de un incremento en la expresión de aquellos genes cuyas secuencias promotoras tienen el motivo de unión para CREB y al mismo tiempo la caja TATA.