|
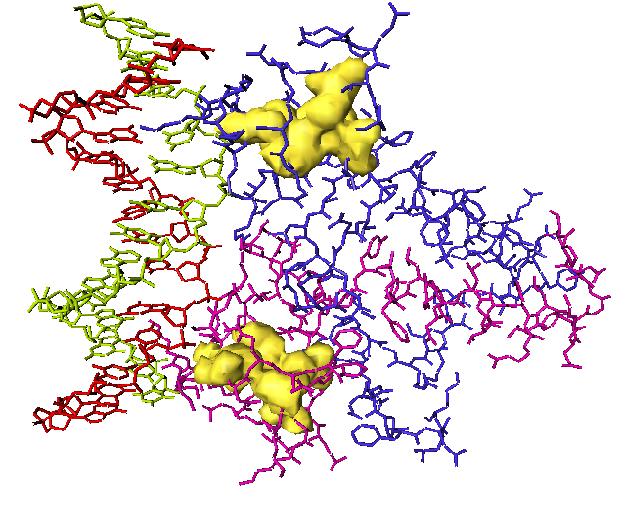
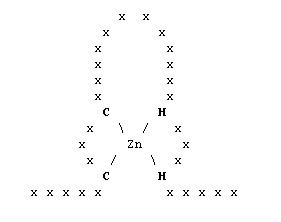
La figura de la izquierda y de la derecha corresponden a imagenes del motivo de reconocimiento "zincfinger" |
El elemento de unión a proteinas en respuesta a AMP ciclico (CREB), es un factor de transcripción de gran importancia. Puede ser activado debido a la presencia de factores hormonales y está implicado en la regulación de la función neuronal y el desarrollo. El objetivo de nuestro trabajo es intentar determinar la distribuón de las posibles dianas de unión sobre el DNA del factor CREB, así como de las proteínas de unión a la caja TATA (dominio del genoma altamente conservado en la región promotora de cualquier loci).
Para hacer el estudio desarollamos en PERL un sencillo programa, el RASTREADOR, capaz de reconocer determinados motivos conservados en el DNA que pueden ser candidatos a ser el sitio de unión de diferentes proteinas que interaccionan con el DNA. En nuestro caso estudiamos las posibles dianas de la proteina CREB, así como de las proteínas de unión a la caja TATA. El programa RASTREADOR se puede ejecutar desde internet en nuestro servidor, introduciendole dos tipos de datos:
- un archivo con las secuencias que se quieren analizar escritas en formato FASTA (sirve tanto para secuencias de proteínas como de DNA, escritas en mayúsculas o en minúsculas).
Estos son algunos ejemplos de las expresiones regulares que debemos introducir en el buscador:
-TATAA (dominio de unión para las proteínas de la caja TATA)
Para nuestro análisis corrimos en el programa los ficheros:
hs_EPD.fasta: Corresponde a secuencias de promotores proximales de genes humanos desde la posición -674 respecto al inicio de transcripción (TSS) hasta la +325.
(en persy.aules.upf.edu: /disc8/promoters/hs_EPD.fasta)
hs_cod.fasta: Corresponde a secuencias codificantes del mismo tamaño a partir del codón de inicio de la traducción ATG.
( en persy.aules.upf.edu: /disc8/coding/hs_cod.fasta)
Debido su gran volumen en el apartado de materiales y metodos incluimos solo unos ficheros ejemplos obtenidos de un fragmento de cada uno.
Después analizamos los ficheros obtenidos al correr el programa y obtuvimos 4 histogramas correspondientes a la distribución del motivo TATAA (unión para las proteínas de la caja TATA)en regiones codificantes y promotores, y la distribución del motivo TGACGTCA (unión para el factor de transcripción CREB)en regiones codificantes y promotores.
- una expresión regular que represente el motivo que se quiera encontrar.
-TGACGTCA (dominio de union para el factor de transcripción CREB)
-C-x(2,4)-C-x(3)-[LIVMFYWC]-x(8)-H-x(3,5)-H (Expresion regular del dominio proteico de unión a " zinc finger")


|
La figura de la izquierda y de la derecha corresponden a imagenes del motivo de reconocimiento "zincfinger" |
La interpretación de estos resultados puede ser complementada con la información de los dos articulos que resumimos a continuación: