 | RESULTATS I CONCLUSIONS |  |
 | RESULTATS I CONCLUSIONS |  |
L'objectiu del projecte era conèixer la distribució dels SNPs en el genoma i al llarg dels gens.
Així doncs podem separar els resultats en dos grans grups, el que inclou l'anàlisi sobre la distribució d'SNPs al genoma, i un altre més específic per la distribució dins els gens.
DISTRIBUCIÓ D'SNPs AL LLARG DEL GENOMA___________________________________________________
Com ja hem dit, el nombre d'SNPs descrits per humà és de 3.175.658 i de 199.441 per ratolí.
A continuació mostrarem una taula que resumeix el nombre d'SNPs per cromosoma de les dues espècies de l'estudi:
Com es pot veure a la taula, el nombre totals d'SNPs que trobem de sumar tots aquells SNPs que es troben en cromosomes és diferent al total d'SNPs que hi ha a l'arxiu SNPs snpNih.txt, aquesta és la prova que existeixen 4.712 SNPs humans que es desconeix el cromosoma al que pertanyen i 719 SNPs de ratolí.
Però encara podem anar més lluny. El nostre programa només calcula en nombre SNPs que es troben en regió gènica, així que la diferència entre el nombre de SNPs dins de cromosomes menys els que ha calculat el nostre programa ens dóna el nombre de SNPs EN REGIONS INTERGÈNIQUES.
H.SAPIENS, 3170946 - 3154996 = 15.950 SNPs en regions intergèniquesQue en el ratolí no haguem trobat cap, no vol dir que no en tingui, simplement que fins ara no s'han trobat.
M.MUSCULUS, 198722 - 198722 = 0 SNPs en regions intergèniques
DISTRIBUCIÓ D'SNPs AL LLARG DE LES REGIONS GÈNIQUES_____________________________________
Ara mirarem si existeix alguna regió gènica on hi trobem major proporció de SNPs per sobre de la resta.
A la taula següent podem veure el percentatge de SNPs en les diferents regions gèniques per humà i ratolí:
| H.sapiens | M.musculus | |
| SNPs en regió exònica codificant | 1.05% | 1.40% |
| SNPs en regions UTR | 1.17% | 2.64% |
| SNPs en llocs d'splicing | 0.45% | 0.61% |
| SNPs en introns | 97.78% | 95.95% |
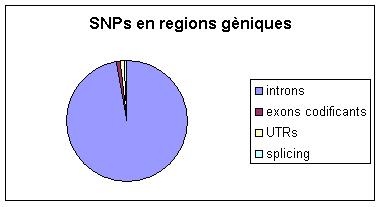 | 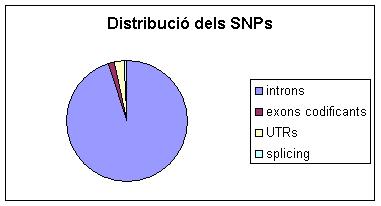 |
El programa ens calcula el total d'SNPs que hi ha a cada regió gènica.
Dins els que es troben a la regió d'splicing podem trobar d'acceptors o donadors. Com que l'splicing és un procés essencial a l'hora de processar l'mRNA, aquesta informació pot ser molt important. Aquí us mostrem el percentatge d'acceptors vs donadors per les dues espècies:
CONCLUSIONS_________________________________________________________________________________
Destacarem que el percentatge de SNPs en regions NO-codificants és molt més gran que en regions codificants. Com hem vits al gràfic anterior estem davant d'un 97,78% (H.sapiens) i 95,95% (M.musculus) d'SNPs intrònics versus el 1,05% i 1,40% exònics.
No sembla sorprendre'ns si tenim en compte que canvis nucleotídics en exons són la causa principal de canvis proteïcs. Com sabem, són regions amb gran pressió selectiva i estrictes a l'hora de patir variacions.
També cal comentar que depenent de si l'SNPs en regió exònica implica canvi de aninoacídic o no, es veurà més o menys afectat per la selecció natural. Si no hi ha canvis de proteïna la selecció no és tan forta.
Trobem una proporció més gran de SNPs en regions d'UTRs que en regions exòniques. Són regions que no es tradueixen a proteïna i en certa manera poden ser més susceptibles a petites variacions, peró per una altra banda es tracta de regions essencials per la transcripció i traducció dels gens així que tampoc ens sorprèn que es trobi en una proporció molt menor que els SNPs en introns.
Una menor proporció d'SNPs en una regió del genoma està relacionat amb la conservació d'aquestes i conseqüentment amb una pressió selectiva superior.
Les regions amb menor quantitat d'SNPs són les d'splicing. Hem volgut comprovar si exitien diferències entre els donadors i els acceptors, peró els resultats obtinguts mostren que les proporcions són del 50%(aprox.) per tots dos.
Pel que fa al nombre de SNPs per cromosoma hem de fer un parell d'aclaracions, tot i que pot semblar que els cromosomes 3-6 en humans tenen un nombre major d'SNPs, no acaba de ser cert del tot. El nombre d'SNPs per cromosoma es troba restringit al nombre de gens seqüenciats d'aquell cromosoma, això significa que si un cromosoma només té un gen seqüenciat el nombre total d'SNPs del cromosoma es veu reduït als possibles SNPs d'aquell únic gen.
Així que si tobem el nombre d'SNPs per cromosoma i dividim entre el nombre de gens per cromosoma, les diferències entre cromosomes són pràcticament indistingibles. Això significa que no hi ha cap cromosoma humà amb una pressió selectiva significativament superior a la resta.
Per una altra banda en M.musculus veiem que el cromosoma 16 té una incidència d'SNPs molt superior a la resta de cromosomes, si corregim les dades tenint el compte el nombre de cromosomes s'observa que aquesta incidència es manté.
La diferència en quantitat d'SNPs entre els cromosomes doncs s'explica per:
Podem concloure que la Selecció Natural no actua amb diferent intensitat al llarg del genoma, al menys en humans, però sí que ho fa segons les regions tenen més o menys significat biològic. Les regions amb més SNPs són les que tenen menys implicació funcional a nivell de proteïna.
L'estudi de les regions codoficants i d'splicing són de gran rellevància en la recerca biomèdica, ja que, com comentàvem a l'inici del treball, algunes de les proteïnes truncades per SNPs poden ser la causa de malalties. Conéixer els polimorfismes responsables d'aquestes malalties permetria un ràpid diagnòstic i tractament.
