Resultados
Análisis de la sobreexpresión de genes del estadío T1 respecto a Ta
Iniciamos nuestro estudio calculando el p-value ajustado y seleccionamos aquellos genes que presenten un valor inferior a 0'01. En este paso obtenemos unos 430 genes,que son los que utilizamos para realizar el análisis de la expresión génica diferencial mediante los programas cluster y treeview.
Para seleccionar los genes sobreexpresados en T1 respecto a Ta hemos de fijarnos en aquellos que presenten color rojo en las muestras pertenecientes a T1 y color verde en las pertenecientes a Ta. Al hacer esto, observamos que hay una gran cantidad de genes que se encuentran sobreexpresados en el estadío T1 (cerca de 150), que se pueden ver en la siguiente tabla (Tabla T1-Ta)
A continuación mostramos el resultado obtenido en el cluster-treeview (utilizando los 430 genes).

|
Aquí podemos observar más detalladamente los genes que se encuentran sobreexpresados.
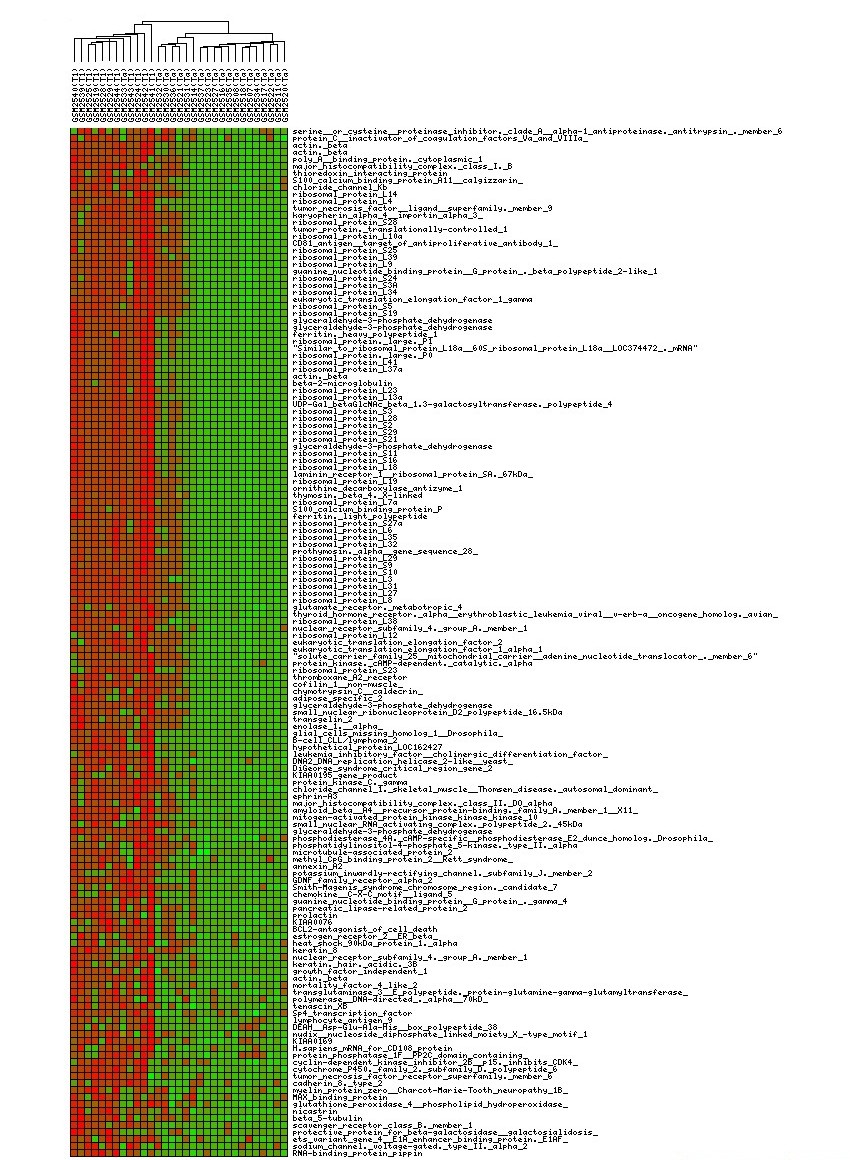
|
Comparamos estos 150 genes con los del artículo ("Gene discovery in Bladder Cancer Progression using cDNA Microarrays") y observamos que no hay ningún gen igual, es decir que en este caso, analizando muestras similares por métodos diferentes no nos da el mismo resultado.
Decidimos buscar similitudes entre las funciones de estos genes, ya que, aunque no encontremos exactamente los mismos genes, queremos comprobar si se conservan las vías metabólicas.
Para realizar el análisis de las las vías metabólicas, tanto de los genes sobreexpresados en T1 como de los genes descritos en el artículo, utilizamos el programa Fatigo.
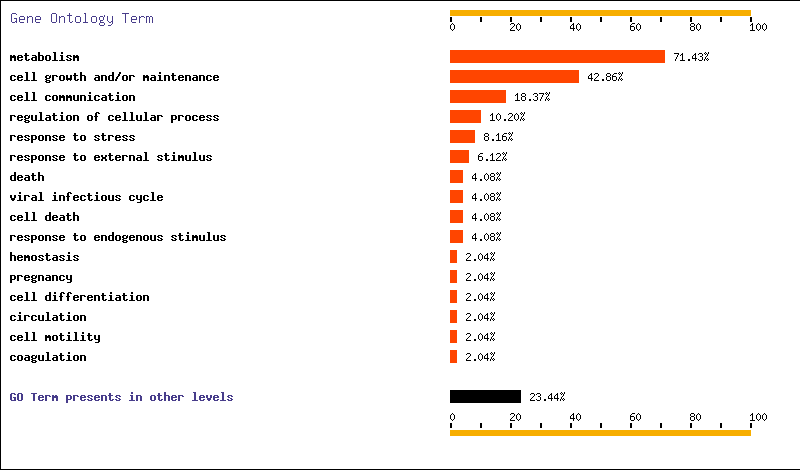
Vías metabólicas implicadas en estadío T1

|
En este análisis es importante destacar que más de la mitad de los genes están implicados en procesos metabólicos, otra parte importante de ellos, en crecimiento y en comunicación celular. Por último encontramos algunas funciones menos mayoritarias como muerte celular, morfogénesis, movilidad celular,...
Vías metabólicas en que están implicados los genes del artículo
|
Podemos observar que hay notables semejanzas entre la función de estos genes. Igual que en el caso anterior encontramos que una gran parte de ellos están implicados en procesos metabólicos mientras que otra parte también importante lo están en el crecimiento y la comunicación celular. También coinciden otras vías minoritarias como respuesta a estímulos externos, muerte o movilidad celular entre otros.
Por último, utilizamos el programa EASE para obtener una descripción específica de la funcionalidad de cada gen. Utilizaremos estos datos para comentar más extensamente algunos de los genes en el apartado de discusión y conclusiones.
Análisis de la sobreexpresión de genes del estadío T2 respecto a Ta
Como en el apartado anterior empezamos calculando el p-value ajustado y seleccionamos aquellos genes que presenten un valor inferior a 0'01. En este caso obtenemos unos 399 genes,que son los que utilizamos para realizar el análisis de la expresión génica diferencial mediante los programas cluster y treeview.
Al fijarnos en el número de genes sobreexpresados en T2 observamos que el número es bastante inferior a los que veíamos en T1. Esta vez hemos encontrado únicamente 20 genes sobreexpresados que son los que vemos en la tabla T2-Ta
El resultado del cluster-treeview (utilizando los 399 genes) es el siguiente:

|
A continuación mostramos únicamente los genes sobreexpresados en T2.

|
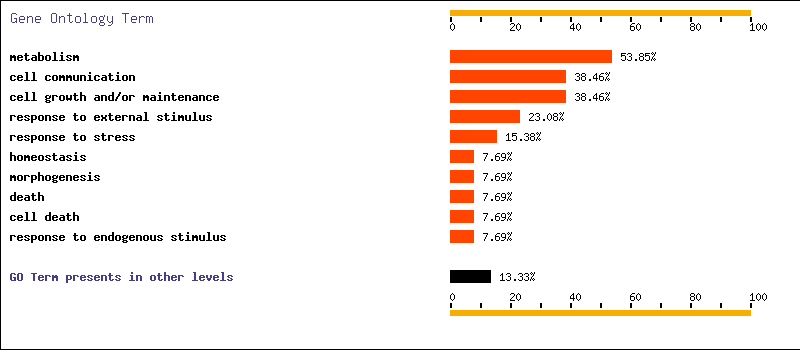
Vías metabólicas implicadas en estadío T2
|
En este análisis hemos encontrado que los genes sobreexpresdos están relacionados principalmente con el metabolismo celular y en un porcentaje algo menor pero también considerable están relacionados con la comunicación y el crecimiento celular.
Si comparamos los estudios realizados con genes de T1 y T2 , vemos que los resultados son muy similares, ya que se encuentran altamente representadas las mismas vías celulares, que también son las mismas que encontramos de forma mayoritaria en los genes del artículo.
También utilizamos el programa EASE para obtener una descripción de la funcionalidad de cada gen. Esta información la utilizaremos posteriormente para describir algunos genes en particular ( apartado discusión y conclusiones).
 |
 |