Discusión y conclusiones
Tras el análisis de los datos y resultados obtenidos en la realización de este trabajo, hemos podido observar que hay ciertos genes que se encuentran sobreexpresados en estadíos avanzados del cáncer, y que no lo están en estadíos tempranos.
Esto nos hace pensar que dichos genes deben estar relacionados con la fisiología del cáncer, la capacidad que tienen las células tumorales de dividirse incontroladamente y la metástasis. Según los resultados obtenidos del Fatigo y del EASE, los genes que han aumentado su capacidad transcripcional son genes implicados en vías metabólicas, en crecimiento, motilidad diferenciación, comunicación y muerte celular, respuestas a estímulos externos y a estrés, etc.
Aquí mostramos algunos ejemplos de estos genes:
Genes T1
| Nombre del gen | |
| Proteinas ribosomales (L3, L27, L31,...) | Los ribosomas son los orgánulos que catalizan la síntesis proteica y consisten en 2 subunidades, una pequeña (40 S) y otra grande (60 S). Estas dos subunidades estan compuestas por 4 RNAs y aproximadamente 80 proteínas estructuralmente diferentes. |
| Factor 2 de elongación de la traducción | Este gen codifica para un miembro de la familia de factores de elongación de la traducción con actividad GTPasa. Promueve la translocación dependiente de GTP de la cadena proteica del locus A al P en el ribosoma. Esta proteina se inactiva completamente por la fosforilación de la quinasa EF-2 |
| BCL2 | Este gen codifica para una proteína de membrana de la mitocondria que bloquea la apoptosis en algunas celulas como los limfocitos. La expresión costitutiva de Bcl2 se cree que puede ser la causa del linfoma folicular. |
| Factor de necrosis tumoral | Pertenece a la superfamilia del receptor-TNF. Este receptor contiene un death domain. Tiene un papel muy importante en la regulación fisiológica de la muerte celular programada y también está implicado en diversas enfermedades relacionadas con el sistema inmune. |
Relación de estos genes con el cáncer
Durante un proceso canceroso, las células pierden el control del ciclo celular y empiezan a dividirse de forma constitutita. En este proceso se ven implicadas numerosas proteínas, que verán muy incrementada su síntesis ya que sus requerimientos también aumentarán notablemente.
Algunas de estas proteínas las encontramos sobreexpresadas en el estadío T1 de este tipo de cáncer, por ejemplo encontramos un aumento de muchas de las proteínas ribosomales que participan en la formación del ribosoma y por tanto son imprescindibles para el proceso de traducción, estas proteínas también se ven incrementadas en el estadío T2. Otro ejemplo sería el factor 2 de elongación que es muy importante en la elongación de la cadena proteica, proceso que también se da durante la traducción.
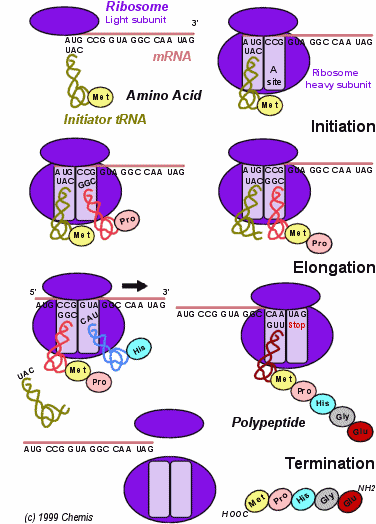 |
Otro proceso que también se ve alterado durante el cáncer es la apoptosis o muerte celular programada, pero en este caso no se produce un aumento de esta vía sino todo lo contrario, se produce una inhibición o lo que es lo mismo un aumento de las proteínas que inhiben la apoptosis. También encontramos ejemplos de éstas proteínas en el estadío T1 del cáncer de vejiga, como el aumento de BCL2, que es capaz de bloquear la apoptosis en algunos tipos celulares o el factor de necrosis tumoral tiene un papel muy importante en la regulación de la apoptosis.
Genes de T2
| Nombre del gen | |
| Factor de necrosis tumoral (ligando) | La proteína codificada por este gen es una citoquina que pertenece a la familia de ligandos de TNF. Esta citoquina y su receptor están implicadas en el proceso de presentación del anfígeno y en la generación de células T citotóxicas. Se ha encontrada expresada en diversos carcinomas y se cree que está implicada en la interacción entre las células T y las células tumorales. |
| Receptor proliferativo de peroxisomas (PPARG) | La proteína codificada por este gen es un miembro de la familia de receptores nucleares (PPAR). PPARs son receptores nucleares involucrados en el transporte de lípidos y en el metabolismo. Su actividad transcripcional está regulada por la unión a ácidos grasos. Hay identificados 3 isotipos, alfa, beta y gamma. PPAR gamma estimula la lipólisis de los triglicéridos circulantes y la consiguiente entrada de ácidos grasos en las células. Solo puede unirse al DNA cuando forma heterodímeros con los receptores RXRs (retinoid X receptors) y estos heterodímeros regulan la transcripción de diversos genes. PPAR gamma, está implicado en la patología de numerosas enfermedades como obesidad, diabetes, arterosclerosis y cáncer. | peroxiredoxina 3 | Esta proteína tiene una función antioxidante y se localiza en la mitocondria. Pertenece a una familia de genes responsable de la regulación de la proliferación celular, diferenciación y funciones antioxidantes. |
Relación de estos genes con el cáncer
Como en el estadío T1, también vemos que en este estadío se ven alteradas diversas vías que promueven un aumento de la división celular y del metabolismo que permite que estas células que no paran de formarse puedan sobrevivir. Por una parte encontraríamos factores como el ligando del factor de necrosis tumoral, que como ya hemos explicado anteriormente es un factor importante en la inhibición de la apoptosis y por tanto en el aumento de la división celular.
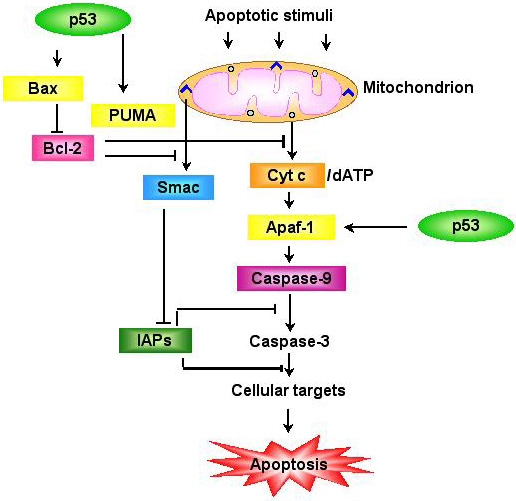 |
Por otra parte encontramos moléculas implicadas en el metabolismo celular, como PPAR gama, que produce un aumento de la degradación de los triglicéridos circulantes y por tanto, un aumento de la entrada de ácidos grasos a las células, cosa que permite cubrir el aumento de la demanda de nutrientes que se produce por el incremento del número de células. Por último, también encontramos genes implicados en la regulación de la proliferación celular como la peroxiredoxina 3.
Genes del artículo
| Nombre del gen | |
| 1 MAX interacting protein 1 | La expression del gen c-myc, que produce un factor de transcripción oncogénico, está fuertemente regulada en células normales pero en células tumorales está frecuentemente desregulado. La proteína codificada por c-myc es un represor transcripcional que regula negativamente la función de MYC y es, por lo tanto, un buen supresor de tumores. La proteína codificada por este gen inhibe la actividad transcripcional de MYC, compitiendo con MAX que es otra proteína que se une a MYC y que es necesaria para su función. |
| 2 defender against cell death 1 (DAD1) | DAD1 protege a la célula frente a la muerte celular programada o apoptosis. La proteïna DAD1 desaparece en células temperatura-sensibles cuando se les aplica un shift, cosa que sugiere que la pérdida de la proteína DAD1 provoca apoptosis. |
| 3 BH3 interacting domain death agonist | Este gen codifica para una proteína agonista de BAX o antagonista de BCL2. La proteína que se forma es de la familia de BCL2. Es un mediador de daño mitocondrial inducido por la caspasa-8 (CASP8). CASP8 rompe la proteína codificada por este gen y el extremo COOH terminal se transloca a la mitocondria donde produce la liberación del citocromo c. |
Relación de estos genes con el cáncer
Igual que en nuestros análisis, los genes que se encuentran sobreexpresados en este estudio están implicados tanto en el proceso proliferativo, como en el metabolismo o el control de la apoptosis. Por ejemplo, encontramos el gen MAX 1 que provoca una desregulación de c-myc y por tanto que no haya represión de la transcripción o genes implicados en el control de la apoptosis como DAD1, que es una proteína antiapoptótica o 3 BH3 que también está relacionada con el proceso de muerte celular programada.
La tecnología de los microarrays de ADNc se ha convertido en una herramienta muy importante para profundizar en los mecanismos moleculares que gobiernan los distintos procesos que tienen lugar en los seres vivos.
Los genes sobreexpresados que hemos encontrado al comparar T1 o T2 (estadíos avanzados del tumor) con Ta (estadíos iniciales) no son los mismos que vieron sobreexpresados en el artículo que hemos analizado, pero dichos genes están relacionados con las mismas rutas y vías, la cual cosa nos hace pensar que si se produce un aumento de la transcripción y la consecuente traducción de estos genes y proteínas, esto se relacionaría con la tumoración de las células.
Los estudios de análisis de datos mediante experimentos de microarrays pueden servir para encontrar genes y proteínas target que podrían utilizarse en el diagnóstico y tratamiento del cáncer. Hemos visto que en estadíos avanzados hay genes que estan sobreexpresados y por lo tanto, debería profundizarse en su implicación en cáncer para posder establecer cuales de ellos podrías utilizarse como posibles dianas terapeuticas. La identificación de nuevos genes marcadores se realiza mediante el rastreo masivo de todos los genes que componen el genoma humano, analizando aquellos que muestren una expresión génica diferente entre células normales y tumorales.
Los resultados que se encuentran mediante la técnica de microarrays de DNA permiten conocer los mecanismos moleculares del cáncer. El análisis sistemático de los patrones de expresión génica contribuirá a un mejor conocimiento molecular de muchas enfermedades. Si se sabe con más precisión qué pasa, se tienen más armas para evitarlo o para combatirlo. El estudio de enfermedades de gran complejidad y que implican alteraciones genéticas, como el cáncer, parece adecuado abordarse a partir de una perspectiva global.
Estas técnicas también puede utilizarse para descubrir nuevas herramientas diagnósticas y clasificar tumores. Hasta ahora, los tumores se han venido clasificando siguiendo criterios histólogicos; esta categorización, aunque de gran valor, ha mostrado ser insuficiente: tumores que bajo el microscopio presentan el mismo aspecto engloban procesos neoplásicos con distintas manifestaciones clínicas y de respuesta a fármacos.
Este trabajo nos ha ayudado a descubrir la importancia de las herramientas informáticas para analizar las grandes cantidades de datos que provienen de experimentos como los microarrays. Gracias a las nuevas tecnologías es cada vez más sencillo obtener información de las secuencias biológicas (ya sea DNA o proteína),pero ahora la dificultad reside en el análisis y tratamiento de estos datos cosa que requiere programas informáticos cada vez más completos.
 |
 |