

Això ens permet validar les prediccions realitzades anteriorment, donat que els ESTs representen fragments de gens codificants reals.
Els Expressed Sequence Tags o ESTs són petits fragments d'ADN, normalment entre 200 i 500 nucleòtids, que han estat generats a partir de la seqüenciació de l'inici i/o del final del ARNm de gens expressats.
- Obtenció de les validacions:
Per tal de validar els gens predits es van seguir els següents passos:
| hg16dna_A.fa | gen 1 de Geneid, Genscan, Grail i FGenes
gen 2 de Grail |
|||
| hg16dna_B.fa | 107.000 | 147.000 | gen 2 de FGenes | |
| hg16dna_C.fa | 126.700 | 268.700 | gen 2 de FGenes i Geneid
gen 3 de FGenes i Genscan gen 4 de Grail |
|
| hg16dna_D.fa | 355.000 | 457.000 | gen 3 de Geneid i Genscan (fragment)
gens 5, 6 i 7 de FGenes gen 7 de Grail |
|
| hg16dna_E.fa | 264.800 | 305.000 | gen 4 de FGenes
gen 6 de Grail |
|
| hg16dna_F.fa | 301.600 | 402.300 | gens 5 i 6 (fragment) de FGenes
gen 3 (fragment) de Genscan i Geneid gen 7 (fragment) de Grail |
Mapa 4. hg16dna_A (.ps) (.pdf)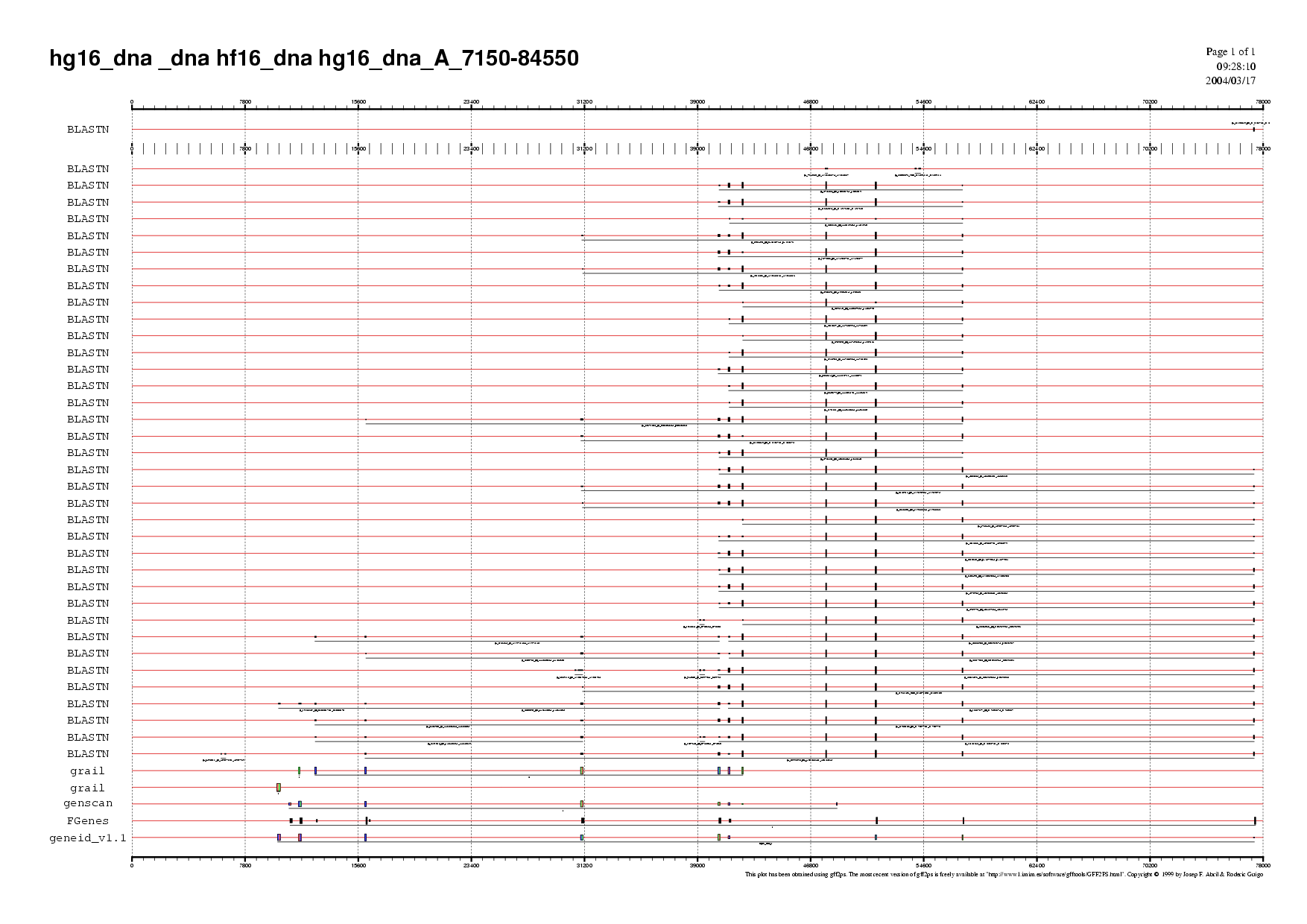 |
Mapa 5. hg16dna_B (.ps) (.pdf)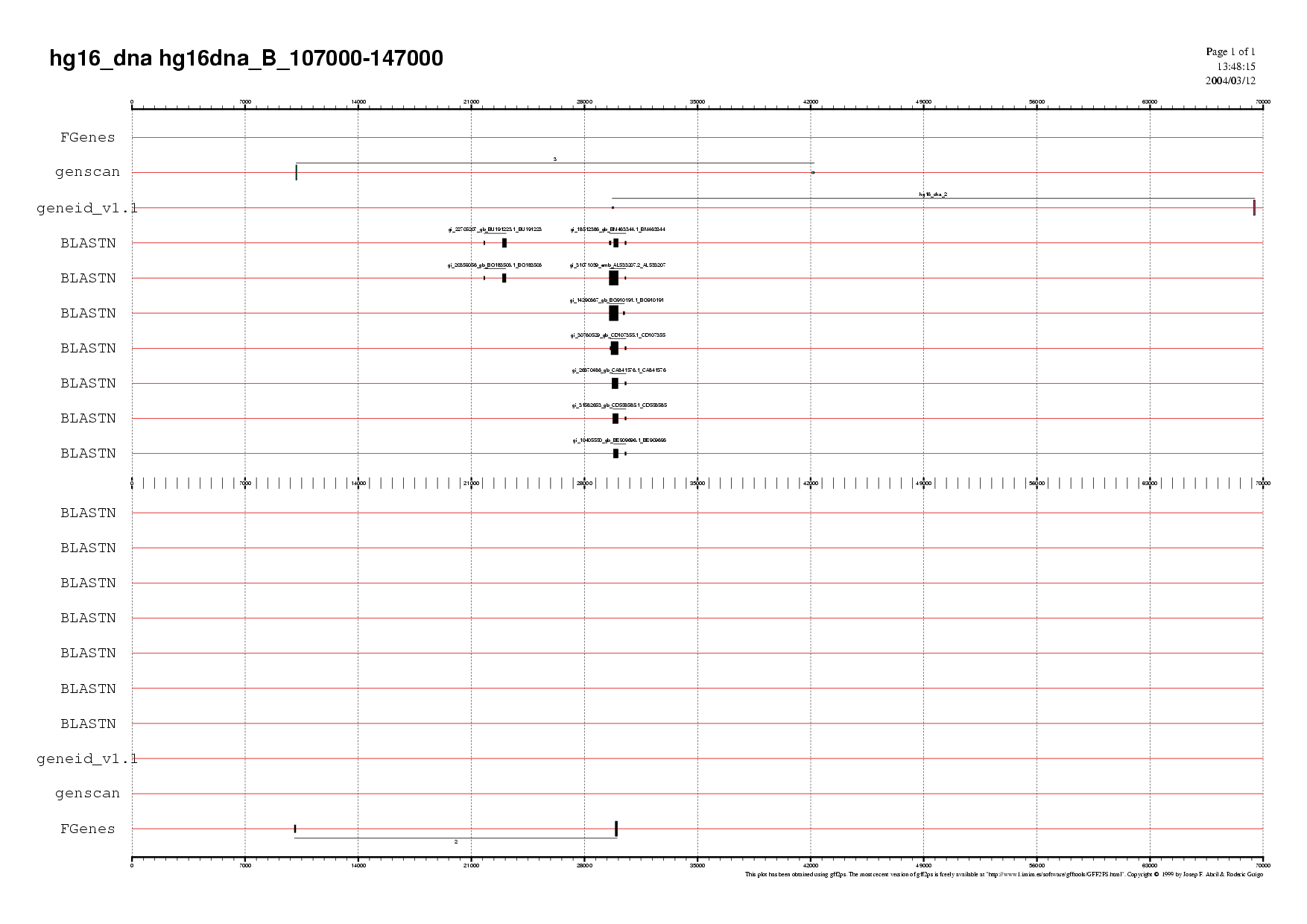 |
Mapa 6. hg16dna_C (.ps) (.pdf)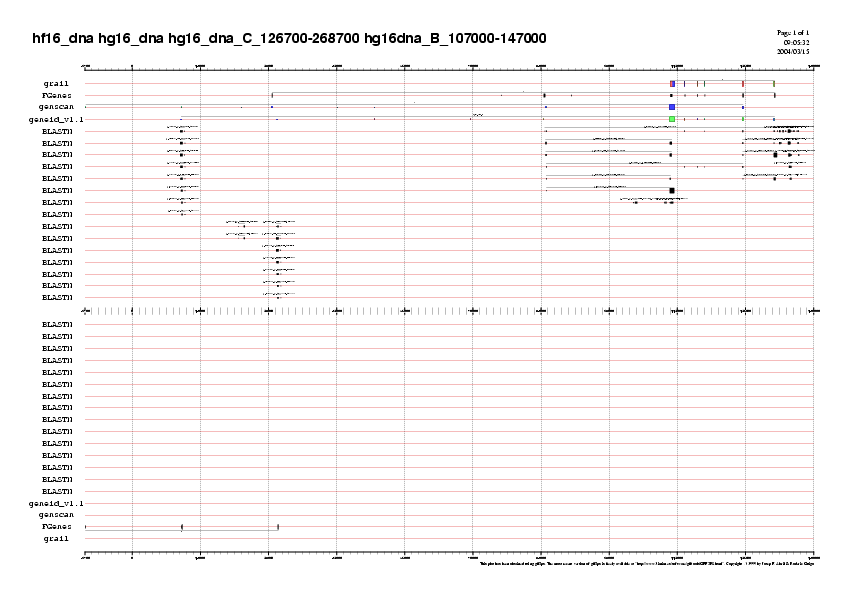 |
Mapa 7. hg16dna_D (.ps) (.pdf)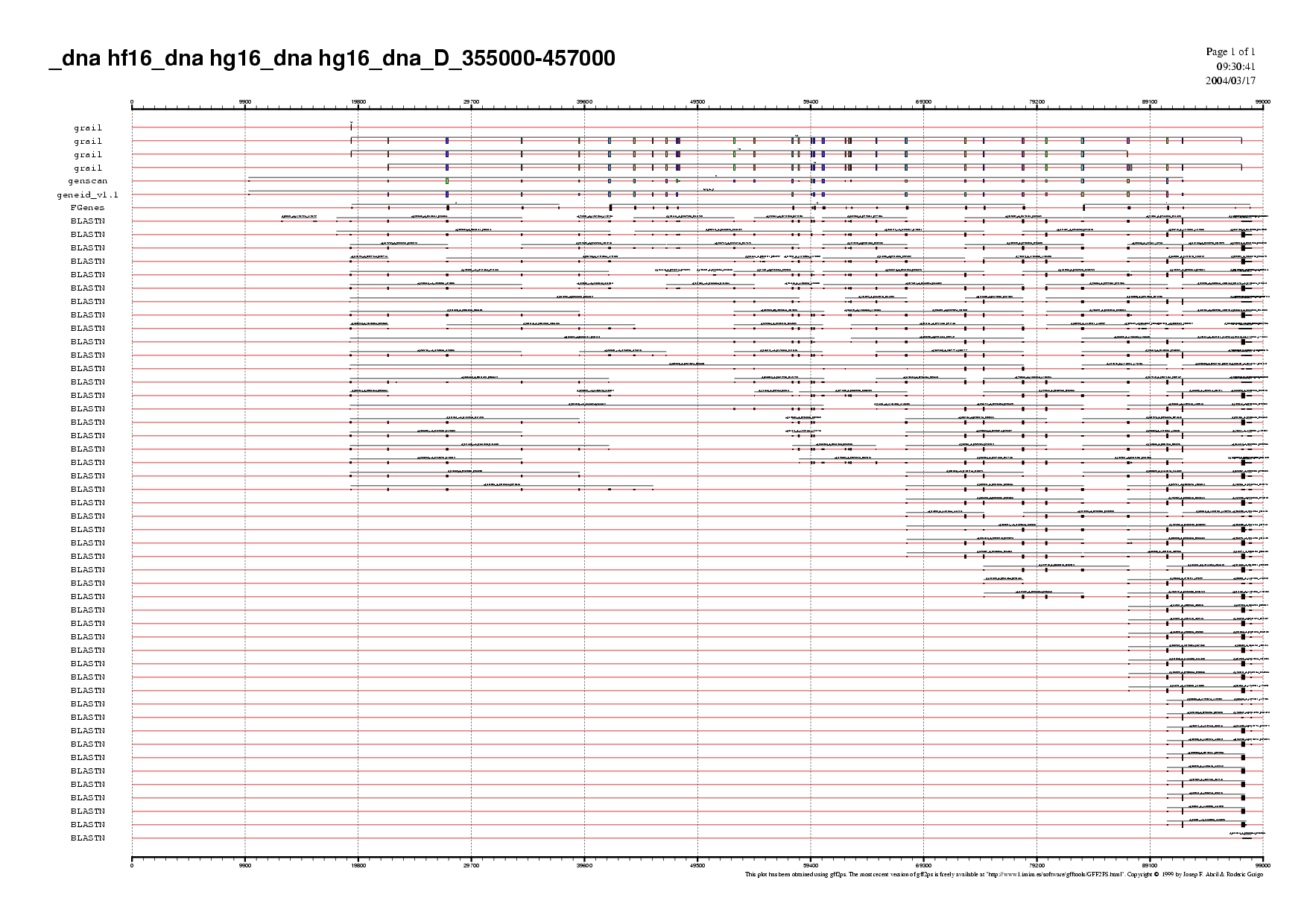 |
Mapa 8. hg16dna_E (.ps) (.pdf)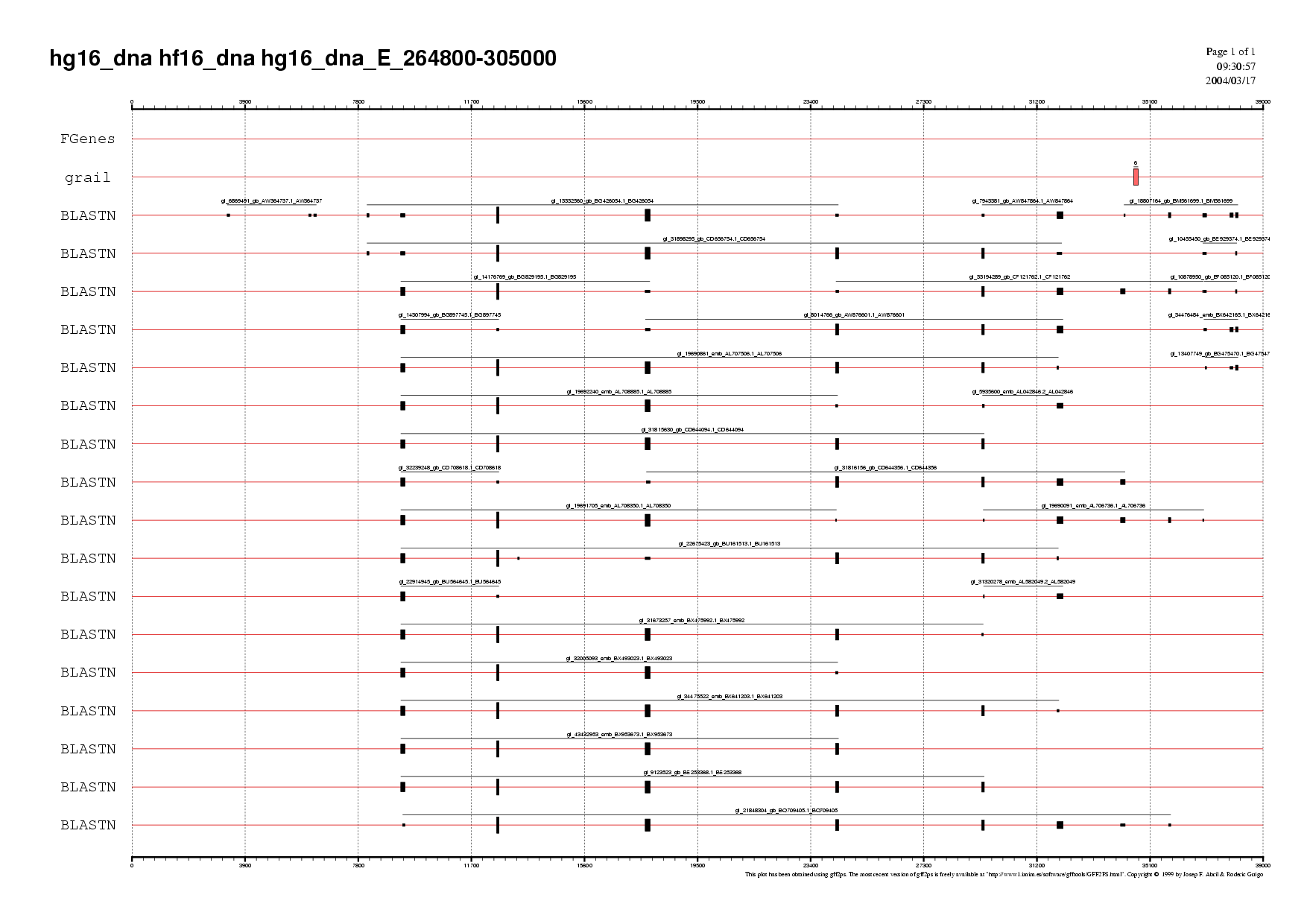 |
Mapa 9. hg16dna_F (.ps) (.pdf)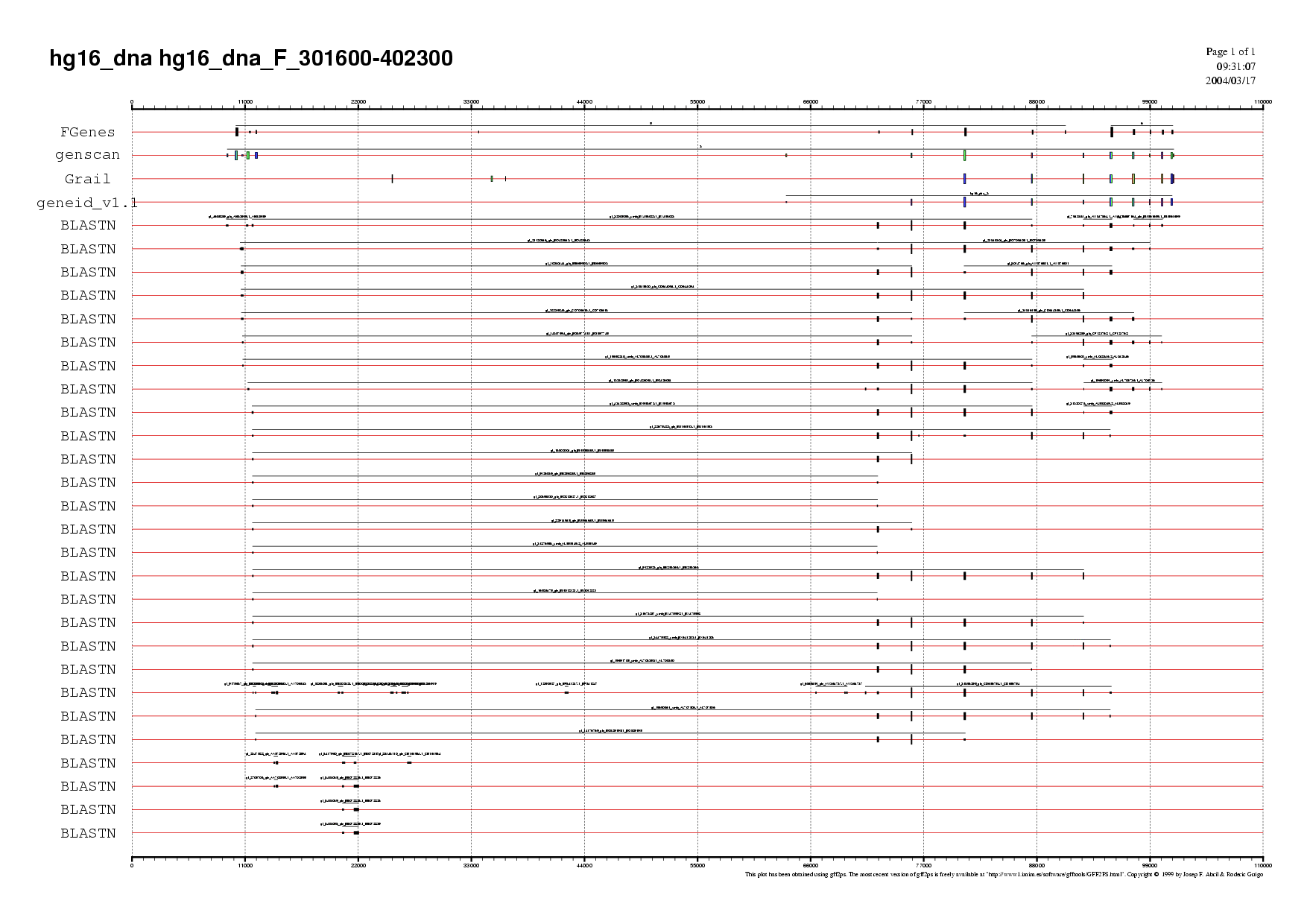 |
 |
 |
 |