

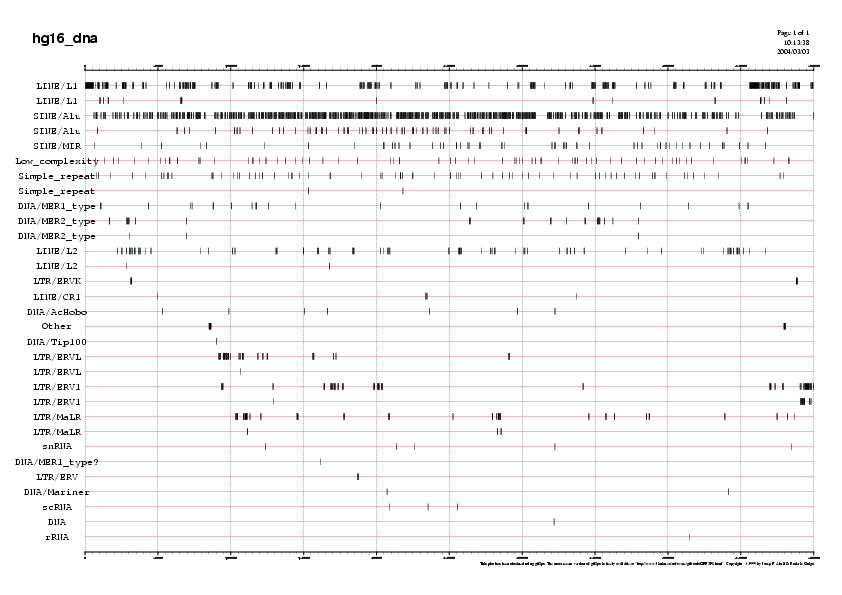
Una gran part del genoma humà correspon a famílies de petit nombre de repeticions. El primer pas a l'hora de realitzar l'anàlisi d'una seqüència és l'emmascarament de les regions repetitives, per tal de reduir el soroll que suposa la presència d'aquestes, que dificultaria l'adequada predicció de gens i, en conseqüència, l'anàlisi del contig.
Aquest programa ens va proporcionar la seqüència emmascarada en la qual les diferents repeticions detectades estan representades amb una N.
A més, el Repeat Masker també va donar la següent taula en la qual es representen les diferents repeticions, el nombre d'elements, la longitud ocupada i el precentatge del total del genoma:
================================================== file name: repeat.seq sequences: 1 total length: 499950 bp (499950 bp excl N-runs) GC level: 41.18 % bases masked: 299532 bp ( 59.91 %) ================================================== number of length percentage elements* occupied of sequence -------------------------------------------------- SINEs: 607 156643 bp 31.33 % ALUs 557 150085 bp 30.02 % MIRs 50 6558 bp 1.31 % LINEs: 160 83307 bp 16.66 % LINE1 102 68623 bp 13.73 % LINE2 55 13836 bp 2.77 % L3/CR1 3 848 bp 0.17 % LTR elements: 58 38951 bp 7.79 % MaLRs 22 12078 bp 2.42 % ERVL 10 8294 bp 1.66 % ERV_classI 23 16200 bp 3.24 % ERV_classII 2 1975 bp 0.40 % DNA elements: 45 10161 bp 2.03 % MER1_type 20 3210 bp 0.64 % MER2_type 13 5549 bp 1.11 % Unclassified: 2 2761 bp 0.55 % Total interspersed repeats: 291823 bp 58.37 % Small RNA: 9 653 bp 0.13 % Satellites: 0 0 bp 0.00 % Simple repeats: 76 4103 bp 0.82 % Low complexity: 74 2984 bp 0.60 % ================================================== * most repeats fragmented by insertions or deletions have been counted as one element The sequence(s) were assumed to be of primate origin. RepeatMasker version 2002/07/13 , sensitive mode run with cross_match version 0.990329 RepBase / RepeatMasker database versions unknown
Mapa 1. Seqüències repetitives
 |
 |
 |