

Los herpesvirus son virus complejos de gran tamaño y ampliamente diferentes en cuanto a su genoma y a sus proteínas, pero muy semejantes por lo que se refiere a la estructura del virión y su organización genómica. Provocan una infección aguda que posteriormente pasa a ser crónica y latente, dando lugar a infecciones recurrentes.
Los viriones de todos los miembros de la familia Herpesviriae comparten una misma estructura: un core con una doble cadena lineal de ADN, una cápside de unos 100-110 nm. de diámetro formada por 162 capsómeros, el tegumento y una cubierta con glicoproteínas virales en su superficie. Este virión puede tener hasta 35 proteínas, entre las cuales enzimas relacionadas con el metabolismo de los ácidos nucleicos, síntesis del ADN y procesamiento proteico13.
El tamaño del virión va desde los 120 hasta los 300 nm. La variación del tamaño depende, en parte, del grosor del tegumento y del estado de la cubierta, pudiendo variar de unos Herpesvirus a otros incluso dentro de la misma especie9.
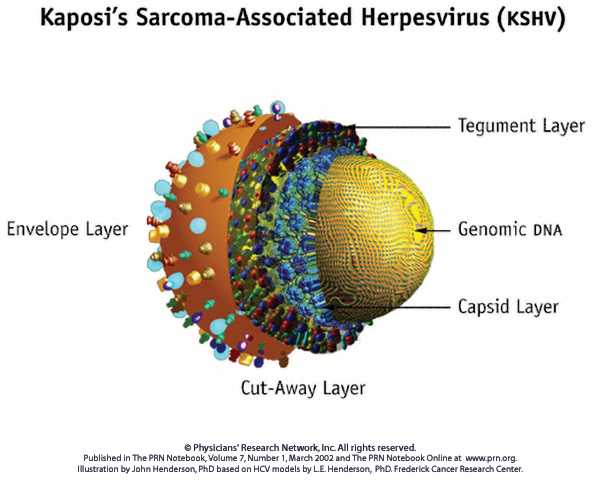
| ADN | 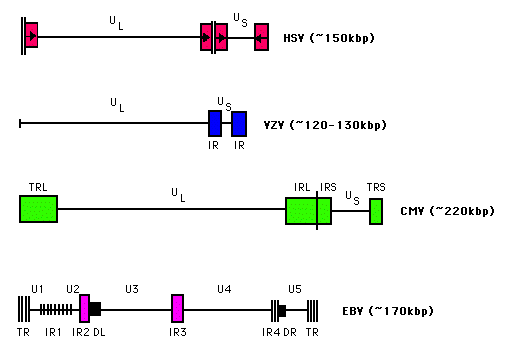 | El material genético de los herpesvirus consiste en una doble cadena de ADN lineal que se circulariza inmediatamente después de ser liberado de la cápside en el núcleo de la célula infectada. El ADN (figura 1), tiene una única región corta (US) y una única región larga (UL), flanqueadas por múltiples repeticiones, invertidas (IR) y/o terminales (TR). Este ADN tiene un tamaño aproximado de entre 120 y 230 kbp, dependiendo de las secuencias repetitivas internas y terminales que pueda tener el virión |
|---|---|---|
| CORE |  | Presenta una forma toroidal (Figura 2) y contiene el ADN viral que en algunos de los casos está asociado a una estructura proteica |
| CÁPSIDE |  | Es una de las estructuras más características de todos los Herpesvirus: es icosahédrica y tiene un diámetro entre 95 y 105 nm. Está constituida por 162 capsómeros de forma tanto pentamérica como hexamérica (Figura 3). Normalmente está constituida a partir de seis proteínas. |
| TEGUMENTO |  | Es una estructura de material amorfo que rodea la cápside (Figura 4). Se halla entre la cápside y la cubierta y aparece como fibrosa en una tinción negativa. |
| CUBIERTA |  | Es una estructura con apariencia típicamente trilaminar debido a que proviene de membranes celulares parcialmente modificadas y ligeramente pleomórficas (Figura 5). Así pues, la cubierta contiene lípidos y numerosas espículas formadas por glicoproteínas virales de unos 8 nm. de tamaño. El número de glicoproteínas diferentes es variable. Por ejemplo, HSV-1 contiene al menos 11 glicoproteínas diferentes. |
![]()
Los Herpesvirus equinos tipo 1 y 4
El herpesvirus equino (EHV) es la principal causa de enfermedad respiratoria, abortos y desórdenes neurológicos en caballos. Aunque originalmente se creía que habían dos subtipos de EHV, posteriormente se vió que en realidad nos hallabamos frente dos miembros diferentes del género Varicellovirus, dentro de la subfamilia Alphaherpesvirinae de la familia Herpesviridae (relación filogénica de los Herpesvirus1), que han recibido el nombre de EHV-1 y EHV-42,3.
De ambos, sólo el EHV-1 causa desórdenes neurológicos, mientras que el EHV-4 raramente causa aborto.

El virión del EHV, como el resto de Herpesvirus (ver los detalles de la estructura de los Herpesvirus), consta de un core con DNA lineal de doble cadena, una cápside icosaédrica de 100-110 nm. de diámetro formada por 162 capsómeros, tegumento envolviendo la cápside y una cubierta lipídica con glicoproteínas de superficie10.
Ambos genomas son colineares y tienen la misma estructura, presentando una secuencia única larga (UL) unida a una secuencia única corta (US) flanqueada por repeticiones invertidas (TRS IRS). Además, presentan 12 sets de repeticiones en tandem que no son codificantes exceptuando tres de ellas, localizadas una en el gen 24 y dos en el gen 712.
Otra similaridad es el número de genes, ya que tanto EHV-1 como EHV-4, tienen 76 orfs que representan 77 gens, debido a que los orfs 44 y 47 sufren un proceso de splicing. A pesar de estas similaridades, los dos genomas presentan diferencias tanto en el tamaño de sus componentes como en su contenido en Cs y Ts2:
| EHV-1 | EHV-4 | |
|---|---|---|
| Tamaño del genoma | 145597 pb | 150223 pb |
| Tamaño UL | 112871 pb | 112398 pb |
| Tamaño US | 11861 pb | 12789 pb |
| TRS IRS | 12714 pb | 10178 pb |
| TRL IRL | 32 pb | ----- |
| Promedio C/T | 56.7 % | 50.5 % |
Nuestro trabajo se basa en el estudio de los genomas de los herpesvirus equinos 1 y 4 a nivel de predicción y posterior análisis de los orfs presentes en su secuencia.
Nuestros objetivos, pues, son:
- Creación de un programa (Findorf 2003, versión 1.0) capaz de identificar orfs en secuencias genómicas carentes de intrones (como es el caso de los genomas de herpesvirus). Ésto incluye la creación de un segundo programa de soporte (Transfasta 2003, versión 1.0) destinado a la transformación de las secuencias a formato fasta, el formato aceptado por el programa de búsqueda de orfs.
- Predicción de todos los posibles orfs en los genomas de EHV1 y EHV4 a través del programa creado e identificación de aquellos orfs ya descritos en la literatura.
- Detección de orfs aún no descritos en los genomas estudiados y su potencial significado biológico y función.