
|
Des de l'obtenció del
Genoma Humà el nombre de seqüències conegudes
és cada cop més superior al nombre de proteïnes
descrites. És per això que l'anàlisi
bioinformàtic es fa cada cop més important i
necessari. Inicialment obtenim la seqüència de DNA a partir de
la base de dades EMBL, la transformem a format fasta i emmascarem
els elements repetitius i de baixa complexitat amb el
RepeatMasker. Posteriorment, utilitzem els programes Geneid i
Genscan per predir l'existència de gens dins la nostra
seqüència. Verifiquem la predicció resultant
mitjançant diferents mètodes:
Posteriorment, fem un estudi dels possibles promotors de les
millors prediccions gèniques obtingudes.
El nostre treball es basa en l'estudi d'una seqüència
genòmica, fins ara no analitzada, gràcies amb
diferents tècniques bioinformàtiques. El contig
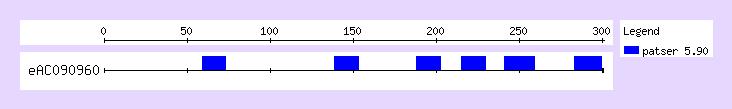
analitzat es troba al cromosoma 3p24.3 humà, situat entre
les posicions 17264874-17441617 d'aquest cromosoma.
Aquest contig va ser clonat amb BAC RP11-83E7, i està anotat
com a AC090960.

Localització del contig AC090960 al cromosoma 3.
Finalment,
analitzem el transcrit resultant de la predicció més
fiable per homologia amb proteïnes a través de
BLASTP.
Torna a l'índex
grep '^ ' AC090960.embl | sed 's/[ 0-9]//g'
> AC090960.fa
i, a través de l'editor de text EMACS, afegim el nom de
la seqüència precedit del símbol >, de manera
que obtenim el fitxer AC090960.fa. awk '{printf "%s", $0}' AC090960.fa &gv;
AC090960.tbl
obtenint el fitxer AC090960.tbl.
A partir d'aquests fitxers, podem obtenir la
informació bàsica de la seqüència
AC090960:
Aproximadament el 40% del genoma humà correspon a
elements repetitius i/o de baixa complexitat. Per tal
d'evitar que aquests puguin ocasionar falsos positius a
l'hora de predir els gens presents, s'han d'emmascar aquestes
regions. Ho fem mitjançant el programa
RepeatMasker.
Després d'introduir la seqüència en format fasta
obtenim els següents arxius:
Per tal de visualitzar la distribució dels
elements emmascarats al llarg de la seqüència,
convertim el fitxer resulant a format GFF. Utilitzem la comanda
grep AC090960 AC090960.seq.out | awk
'BEGIN{OFS="\t"}{print $5, $11, "repeat", $6,$7,".", ".", "."}'
> AC090960.seq.out.gff
Un cop hem obtingut la seqüència emmascarada en
format GFF: AC090960.seq.out.gff,
la podem passar al programa gff2ps,
que ens hem intal.lat localment: gff2ps local.
Gràcies a aquest, podem transformar el fitxer de manera que sigui visualitzable amb el
programa GhostView.
./gff2ps AC090960.seq.out.gff >
AC090960.seq.out.ps gv AC090960.seq.out.ps
Ho convertim en una imatge: convert
-antialias -rotate 90 AC090960.seq.out.ps AC090960.seq.out.jpeg Per a més informació, clikeu sobre l'arxiu següent.
Per tal que aquest seguit de repeticions es puguin visualitzar
conjuntament amb les prediccions que realitzarem, les projectem
en dues línees, separant els repeats dels elements de
baixa complexitat, per la comanda awk:
grep AC090960 AC090960.seq.out | awk
'BEGIN{OFS="\t"}{if($11~/Low_complexity/){SRC="LowComplexity";}
else { SRC="Repeat";} {print $5, $11, "repeat", $6,".", ".", "."}'
> AC090960.repeat.gff )
La predicció de gens es realitza normalment a partir
de programes informàtics com són
Geneid i
Genscan.
Aquests dos programes es basen en el fet que al llarg del proteoma les
proteïnes utilitzen els mateixos aminoàcids en
diferents freqüències. A més a més, les
freqüències de l'usatge de codons sinònims per
al mateix aminoàcid també són
diferents. Aquests dos fets provoquen un gran biaix de codons a les
regions codificants que, per norma general, no trobem a les no
codificants. Tant Geneid com Genscan utilitzen el patró
periòdic que és creat per aquest biaix per tal de poder
distingir les regions codificants de les que no ho són.
També té en compte els senyals detectats a la seqüència
(acceptors i donadors d'splicing, inici i final de transcripció).
En tots dos programes hem d'introduir la seqüència
problema emmascarada en format fasta. En el Geneid, podem demanar
directament els resultats en GFF però en el Genscan no. Per tant,
haurem de pasar aquests resultats a GFF per poder-los visualitzar:
gawk 'BEGIN{OFS="\t"}$2 ~
/Term|Intr|Init/{print "AC090960", "genscan", $2, start=($4<$5 ? $4
: $5), end=($5<$4 ? $4 : $5),$13,$3,$7, $1}' AC090960.genscan | sed
's/\.[0-9][0-9]$//' > AC090960.genscan.gff
Un cop tenim les prediccions en format GFF
(AC090960.geneid.gff i
AC090960.genscan.gff)
les podem visualtizar juntament gràcies al gff2ps, al GhostView i a
les comandes:
gff2ps AC090960.geneid.gff AC090960.genscan.gff >
AC090960.genepredictions.ps gv AC090960.genepredictions.ps Les prediccions de gens dels programes Geneid i Genscan tenen
una fiabilitat aproximada del 50%, fet que fa que sigui necessari validar-les per
diferents mecanismes per tal de millorar-les. Primerament, hem comparat la seqüència contra una
base de dades de EST (Expressed Sequence Tags) humans.
Utilitzem l'aplicació Megablast del
BLAST, per tal que
només apareguin els ESTs que siguin quasi
idèntics, ja que megablast empra paraules
llargues per cercar homologies (és més restrictiu).
Els resultats del megablast són els següents:
AC090960.blast.est
Els convertim a format GFF mitjançant el programa
PARSEBLAST:
Donat que els EST presenten molts errors, ens centrem
només en els EST que presenten splicing, ja que
això concedeix més probabilitat que siguin
trancrits d'aquesta seqüència. Els ESTs que
s'alinein en diferents fragments separats a la
seqüència, són els que experimenten processos
d'splicing. Val a dir que, tot i que aquests EST amb splicing
són més fiables, no estem tenint en compte que a
més els hsp que composen l'EST siguin contigus i
continus, ja que això és també una
condició necessària per acceptar-los com a una
evidència biològica vàlida. Mitjançant el gff2ps, generem un gràfic
juntament amb les prediccions de Geneid i Genscan, i els
fragments emmascarats, projectats en dues línies (tots
els repeats i els elements de baixa complexitat).
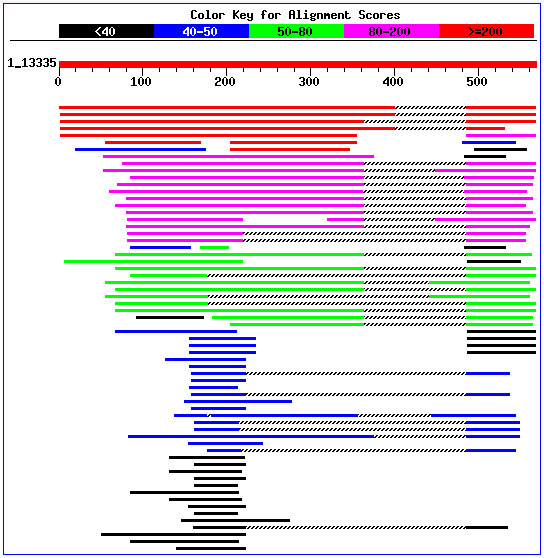
Clicant sobre el gràfic o mirant el fitxer dels EST amb splicing,
es pot veure que apareixen 25 ESTs diferents. A grans trets,
podem veure que aquests suporten la majoria d'exons predits tant
per Geneid com per Genscan, tot i que també hi ha exons
que no es troben suportats per cap EST i ESTs que no
estan recollits en les prediccions.
Podem observar que un gran nombre d'ESTs
(concretament 12) es troben en la mateixa posició (31724-31796)
i no suporten l'existència de cap gen predit.
Degut a aquesta evidència biològica no recollida en
la nostra predicció i a que els programes de predicció
tenen més dificultat en trobar els exons inicial i terminal,
pensem que podria tractar-se de l'exó inicial que ens falta.
Forcem al Geneid a que trobi tots els possibles exons inicials de la nostra
seqüència. Ens fixem en els exons incials que es situen
al voltant de l'hsp que estem analitzant (posicions 31724 -
31796): Veiem que aquests possibles exons inicials tenen puntuacions
molt baixes i, per tant, descartem que aquests exons corresponguin
realment a l'exó inicial. L'exó inicial pot estar
localitzat fora del contig que estem analitzant, o bé pot correspondre a aquest hsp però
que es trobi emmascarat. Per aquesta raó, en apartats
posteriors aquest hsp seran dianes d'estudis més
acurats.
Val a dir també que els ESTs trobats no sempre presenten els mateixos hsp, de
manera que això ens pot indicar la presència d'splicing alternatiu
en el processament del mRNA de la predicció gènica.
Vist els resultats anteriors, vam decidir validar les prediccions
extenent la nostra seqüència (referida com a eAC090960),
per tal de saber si els exons inicial i final es trobaven fora del
nostre contig.
Per tal d'això, vam emprar el programa
Golden Path
on vam introduir el nom del nostre contig (AC090960) i, gràcies a l'opció
Get DNA
vam poder allargar la nostra seqüència 30000 pb upstream
i 30000 pb downstream. D'aquesta manera, mentre que el contig AC090960
era d'aproximadament 177000 pb ( ch3: 17264874-17441617), la seqüència allargada
és d'aproximadament 237700 pb (ch3: 17234874 al 17471617).
El fet que el Golden Path allargui la seqüència a
partir d'un cromosoma ensamblat i no a partir d'un contig major, fa
que la seqüència no només sigui la
complementària sinó que a més, hi hagi
fragments en aquesta representats com a N degut a gaps en la
seqüenciació cromosòmica.
Grabem el resultat com a eAC090960.fa
i a partir d'aquest tansformat en format tabular
awk '{print "%s", $0} $0~/^>/ { printf "t" }' eAC090960.fa | gawk '{OFS="\t"; gsub(">"," ",$1); print $1, $NF }'> eAC090960.tbl
Aquesta seqüència allargada ha d'experimentar els
mateixos processos que l'AC090960 i, per tant, ha de ser emmascarada amb el programa
RepeatMasker obtenint-ne, de nou, els següents tipus d'arxius:
Novament, l'arxiu amb el tipus de repeticions ha de ser transformat
a format GFF per tal de poder ser
visualitzat gràcies al gff2ps i al GhostView:
Fem la predicció gènica amb Geneid i
Genscan a partir de la seqüència extesa emmasarada i
ho convertim a format GFF per la comanda gawk abans emprada.
Ho convertim també a format TABULAR amb la comanda
(comandes awk '{print $2}' AC090960.tbl | fold -1 |
sort | uniq -c | gawk '{print $2, $1}' i
awk '{print $2}' AC090960.tbl | fold -1 | sort |
uniq -c | gawk '{print $2, $1/176744}' )
A : 54078 0.305968
C : 30494 0.172532
G : 32166 0.181992
T : 60005 0.339502
N : 00001 5.6579e-6
==================================================
file name: repeat.seq
sequences: 1
total length: 176744 bp (176744 bp excl N-runs)
GC level: 35.45 %
bases masked: 70875 bp ( 40.10 %)
==================================================
number of length percentage
elements* occupied of sequence
--------------------------------------------------
SINEs: 72 15625 bp 8.84 %
ALUs 43 11517 bp 6.52 %
MIRs 29 4108 bp 2.32 %
LINEs: 52 35512 bp 20.09 %
LINE1 26 25945 bp 14.68 %
LINE2 25 9318 bp 5.27 %
L3/CR1 1 249 bp 0.14 %
LTR elements: 16 8837 bp 5.00 %
MaLRs 8 5476 bp 3.10 %
ERVL 1 544 bp 0.31 %
ERV_classI 7 2817 bp 1.59 %
ERV_classII 0 0 bp 0.00 %
DNA elements: 26 8315 bp 4.70 %
MER1_type 16 2820 bp 1.60 %
MER2_type 7 4937 bp 2.79 %
Unclassified: 0 0 bp 0.00 %
Total interspersed repeats: 68289 bp 38.64 %
Small RNA: 1 40 bp 0.02 %
Satellites: 0 0 bp 0.00 %
Simple repeats: 15 1008 bp 0.57 %
Low complexity: 42 1581 bp 0.89 %
==================================================
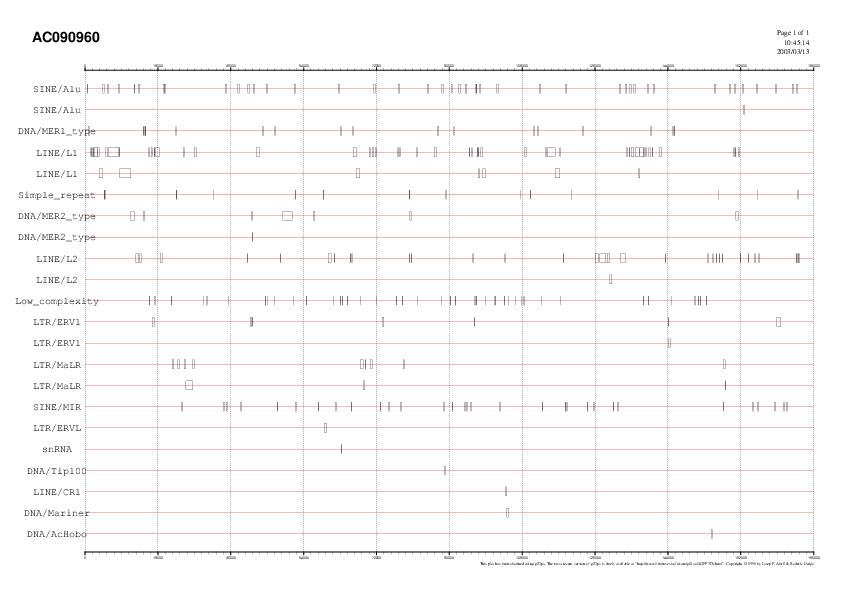
Clicant a la imatge trobem en el gràfic les diferents
repeticions presents a la nostra seqüència.
Observem el tipus de repeticions, la quantitat en que
es troben i la posició exacta de cadascuna d'elles.
Nosaltres hem cregut oportú realitzar tot un seguit de validacions
addicionals ja que les prediccions incials han resultat
especialment incomplertes.
parseblast -Gi AC090960.blast.est | gawk
'BEGIN{OFS="\t"}{$2=$9;$6=$7=".";print}' > AC090960.blast.est.gff
Utilitzem un programa en awk per filtrar aquests ESTs amb splicing
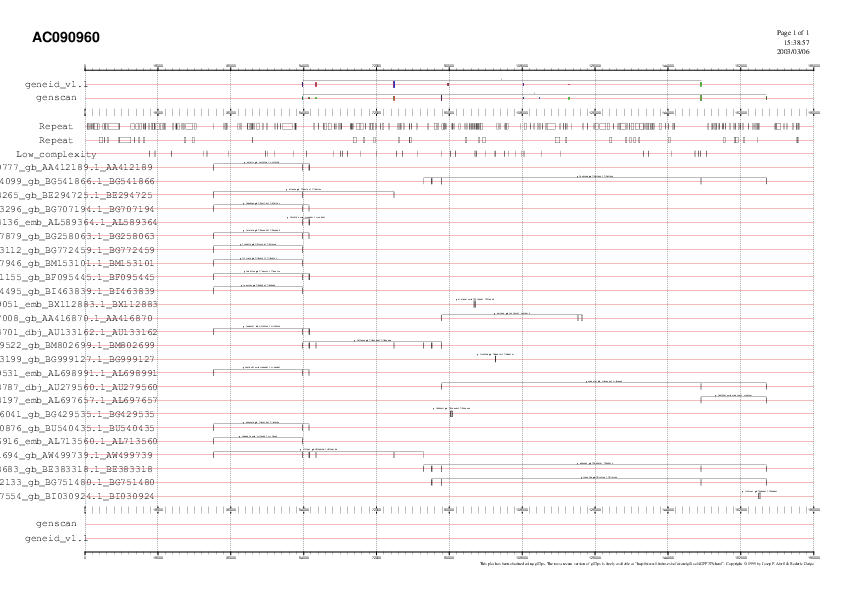
Link amb la predicció gènica juntament amb els
ESTs amb splicing trobats.
Exons predits per Geneid no suportats per ESTs: 108258 108330
119550 119590
Exons predits per Geneid no suportats per ESTs: 108263 108399
112174 112196
119555 119595
===================================================== =========================================================
POSICIONS nº de HSP EXONS SUPORTATS POSICIONS nº de HSP EXONS SUPORTATS
===================================================== =========================================================
31724 31796 12 Cap 87996 88170 2 Geneid ? (89650 89828
31752 31796 1 87996 88175 1 Genscan (88003 88175)
87996 88170 1
53714 53827 13 Geneid (53719 53827) 88105 88170 1
53717 53827 17 Genscan (53724 53832) 88111 88170 1
53780 53827 1
90267 90332 1 Cap
55282 55373 6 Genscan (55289 555378)
55282 55344 1 90363 90596 1 Cap
55282 55373 3
90716 90755 1 Cap
55477 55513 1 Genscan (55482 55556)
55477 55551 4 96002 96210 1 Cap
55480 55548 1
96281 96330 2 Cap
56996 57066 2 Geneid (56997 57064)
Genscan (57002 57069) 96401 96542 1 Cap
76230 76333 3 Geneid (76230 76332) 101304 101350 1 Cap
Genscan (76235 76337) 101381 101429 1 Cap
101381 101429 1 Cap
83629 83720 1 Cap
83629 83661 1 121670 121797 1 Cap
83635 83720 1 122607 122856 1 Cap
83693 83720 1
152111 152253 2 Geneid (152111 152253)
85564 85616 4 Cap 152117 152253 1 Genscan (152116 152258)
85705 85775 4 Cap 166429 166479 1 Cap
166603 166900 1 Cap
168244 168350 5 Genscan (168249 16835)
========================================================== =========================================================
POSICIÓ SCORE
====================================
31741 - 31755 -7.69
31965 - 31976 -10.19
Obtenim informació bàsica com:
A : 76739
C : 41136
G : 39577
T : 69291
N : 10001
A : 0.324143
C : 0.173757
G : 0.167172
T : 0.292683
N : 0.0422439
==================================================
file name: repeat.seq
sequences: 1
total length: 236744 bp (226744 bp excl N-runs)
GC level: 35.60 %
bases masked: 93199 bp ( 39.37 %)
==================================================
number of length percentage
elements* occupied of sequence
--------------------------------------------------
SINEs: 95 20960 bp 8.85 %
ALUs 59 15811 bp 6.68 %
MIRs 36 5149 bp 2.17 %
LINEs: 72 44784 bp 18.92 %
LINE1 36 32602 bp 13.77 %
LINE2 35 11933 bp 5.04 %
L3/CR1 1 249 bp 0.11 %
LTR elements: 20 10753 bp 4.54 %
MaLRs 11 6484 bp 2.74 %
ERVL 2 607 bp 0.26 %
ERV_classI 7 3662 bp 1.55 %
ERV_classII 0 0 bp 0.00 %
DNA elements: 35 13659 bp 5.77 %
MER1_type 21 5199 bp 2.20 %
MER2_type 11 7902 bp 3.34 %
Unclassified: 0 0 bp 0.00 %
Total interspersed repeats: 90156 bp 38.08 %
Small RNA: 1 40 bp 0.02 %
Satellites: 0 0 bp 0.00 %
Simple repeats: 19 1128 bp 0.48 %
Low complexity: 49 1875 bp 0.79 %
==================================================
Gràfic de les repeticions trobades
a la nova seqüència extesa.
# eAC090960.GENEID
Gene 1 (Reverse). 9 exons. 303 aa. Score = 6.336276
eAC090960 geneid_v1.1 Internal 5006 5091 0.74 - 0 1
eAC090960 geneid_v1.1 Internal 38395 38501 -0.21 - 2 1
eAC090960 geneid_v1.1 Internal 54492 54634 1.46 - 1 1
eAC090960 geneid_v1.1 Internal 87155 87195 -1.01 - 0 1
eAC090960 geneid_v1.1 Internal 98415 98487 -0.22 - 1 1
eAC090960 geneid_v1.1 Internal 116917 117095 0.49 - 0 1
eAC090960 geneid_v1.1 Internal 130413 130515 2.88 - 1 1
eAC090960 geneid_v1.1 Internal 149681 149748 0.98 - 0 1
eAC090960 geneid_v1.1 Internal 152918 153026 1.22 - 1 1
# eAC090960.GENSCAN
eAC090960 genscan Intr 5012 5097 5.10 - 0 1
eAC090960 genscan Intr 38401 38507 5.11 - 0 1
eAC090960 genscan Intr 54498 54640 11.68 - 0 1
eAC090960 genscan Intr 87161 87201 1.90 - 0 1
eAC090960 genscan Intr 94560 94582 -1.76 - 2 1
eAC090960 genscan Intr 98357 98493 0.09 - 2 1
eAC090960 genscan Intr 118581 118753 10.32 - 1 1
eAC090960 genscan Intr 130419 130521 7.26 - 2 1
eAC090960 genscan Intr 149687 149754 0.98 - 0 1
eAC090960 genscan Intr 151200 151274 0.59 - 2 1
eAC090960 genscan Intr 151378 151467 0.57 - 0 1
eAC090960 genscan Intr 152924 153032 3.47 - 1 1
eAC090960 genscan Init 232985 233100 3.33 - 0 1
Per tal de validar-les, fem una cerca per MEGABLAST per mirar si apareixen nous ESTs i convertim el resultat a format GFF a través del PARSEBLAST, filtrant també només els que presenten splicing: eAC090960.blast.est.spliced.gff
Un cop fet això, generem un gràfic amb les noves prediccions i els ESTs:
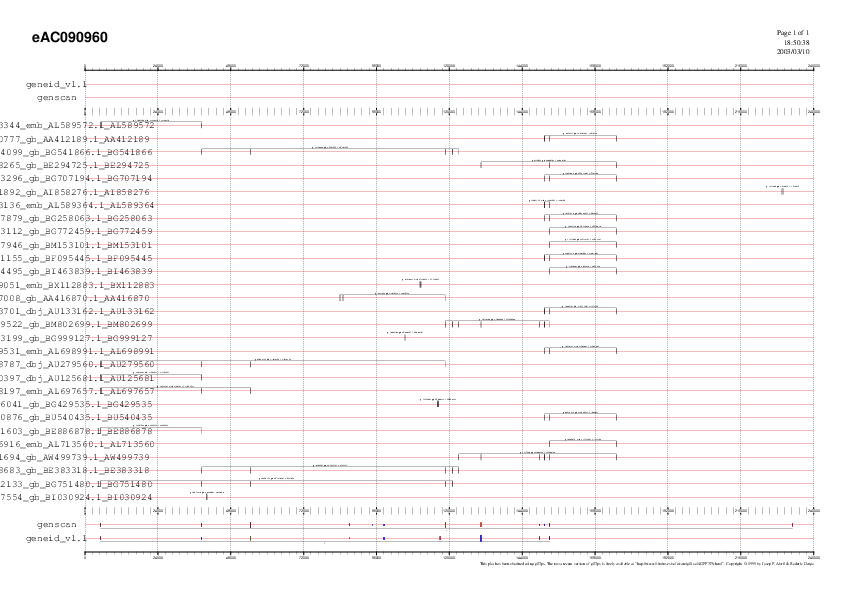
Predicció gènica amb la seqüència extesa juntament
amb els ESTs amb splicing trobats.
Si mirem aquestes prediccions amb la seqüència
extesa veiem que els dos programes ens prediuen un gen en
revers. Si ens fixen bé, aquest gen correspon al gen
predit en el contig AC090960 inicial però, com ja s'ha comentat
anteriorment, la seqüència extesa és la
complementària de la inicial i, per tant, el gen trobat en
forward ara es trobarà en revers.
Un estudi exastiu del gen predit en eAC090960 permet visualitzar
dos nous exons interns
a l'extrem final del gen situats entre les bases 5006-5091 i 38395-38501.
Però el més remarcable és que la predicció
de Genscan troba un exó inicial!
Malgrat això, tot i que aquest exó inicial
tingui un score relativament bo (3.33), no presenta ESTs que el
suportin. L'EST AIB58276 és molt proper a aquest exó
inicial, però malauradament no hi coincideix exactament (es
troba lleugerament més downstream).
Val a dir també que la predicció continua sent incomplerta per l'extrem 3', on tot i extendre 30,000 pb no s'ha trobat cap exó terminal.
La predicció obtinguda després d'extendre la seqüència no acaba de ser del tot concluent. Per això decidim fer una validació addicional. En aquest cas, variem els paràmetres d'emmascarament. D'aquesta manera podrem guanyar sensitivitat en la predicció, tot i que hem de tenir en compte que també estem perdent especificitat.
En la predicció anterior els 12 ESTs de l'estrem 5' que es situaven entre les posicions 31724 - 31796, continuen sense estar recollits. Donat que aquests suposen un evidència biològica no despreciable, refem la prediccó gènica: aquest cop no emmascarerem els simple repeats ni els low complexity (fragments de baixa complexitat) lAC090960.seq.masked. També refarem la predicció partint de la seqüència sense cap tipus d'emmascarament nAC090960.
Per tant, podrem veure si la raó per la qual la predicció no recull aquestes posicions 5' on apareixen tants ESTs, és que aquestes estan emmascarades. En cas que aquí es trobés l'exó que busquem, ara la predicció el podria recollir. A més també podr&iaucte;n apareixer altres exons addicionals o un exoó inicial situat en una posició diferent.
Procedim amb les dues seqüències (lAC090960 i nAC090960) com en la ocasions anteriors:
| lAC090960 | nAC090960 | |
| Emmascarament: | Excloem simple repeats i low complexity. | No emmascarem |
| Predicció amb Geneid: | lAC090960.geneid.gff | nAC090960.geneid.gff |
| Predicció amb Genscan: | lAC090960.genscan.gff | nAC090960.genscan.gff |
| Cerca d'EST: | lAC090960.blast.est.gff | nAC090960.blast.est.gff |
PREDICCIONS lAC090960:
 Link a la predicció amb lAC090960 |
lAC090960:GENEID
Gen1:
lC090960 geneid_v1.1 Internal 53719 53827 1.23 + 1 1
lC090960 geneid_v1.1 Internal 56997 57064 0.99 + 0 1
lC090960 geneid_v1.1 Internal 76230 76332 2.87 + 1 1
lC090960 geneid_v1.1 Internal 87998 88170 2.84 + 0 1
lC090960 geneid_v1.1 Internal 108258 108330 -0.22 + 1 1
lC090960 geneid_v1.1 Internal 119550 119590 -1.01 + 0 1
lC090960 geneid_v1.1 Internal 152111 152253 1.46 + 1 1
|
lAC090960:GENSCAN
lAC090960 genscan Intr 1453 1607 3.59 - 1 1
lAC090960 genscan Intr 1643 1742 -0.05 - 0 1
lAC090960 genscan Intr 9256 9846 17.94 - 1 1
lAC090960 genscan Intr 10173 10409 11.46 - 0 1
lAC090960 genscan Init 11238 11283 4.50 - 0 1
lAC090960 genscan Init 40190 40261 4.72 + 1 2
lAC090960 genscan Intr 44280 44386 -1.31 + 2 2
lAC090960 genscan Intr 53719 53827 3.47 + 1 2
lAC090960 genscan Intr 55284 55373 0.57 + 2 2
lAC090960 genscan Intr 55477 55551 0.59 + 0 2
lAC090960 genscan Intr 56997 57064 0.98 + 2 2
lAC090960 genscan Intr 61378 61537 0.47 + 1 2
lAC090960 genscan Intr 64064 64173 -0.24 + 1 2
lAC090960 genscan Intr 67569 67648 5.68 + 0 2
lAC090960 genscan Intr 68976 69066 -5.37 + 1 2
lAC090960 genscan Intr 76230 76332 7.26 + 0 2
lAC090960 genscan Intr 87998 88170 12.92 + 1 2
lAC090960 genscan Intr 108258 108394 0.09 + 0 2
lAC090960 genscan Term 116452 117329 16.05 + 2 2
lAC090960 genscan Init 130339 130392 3.63 + 0 3
lAC090960 genscan Intr 140619 140697 3.61 + 2 3
lAC090960 genscan Intr 154902 155180 4.23 + 1 3
lAC090960 genscan Intr 168244 168350 5.11 + 2 3
lAC090960 genscan Intr 170837 171116 3.63 + 1 3
lAC090960 genscan Term 171268 171488 6.02 + 2 3
|
La predicció de Geneid no varia substancialment repecte les prediccions anteriors.
Per contra, observem que Genscan ara prediu tres gens:
- El primer és un petit fragment que apareix en
revers a l'extrem 5'. Donat que les prediccions anteriors
no hi coincideixen i no té suport d'ESTs, ho atribuïm a la
manca d'especificitat causada al disminuir l'emmascarament.
- El segon gen predit és més destacable, ja que
presenta exons inicial i terminal, amb scores de 4.72 i
16.05, respectivament. Malgrat això, l'exó
inicial es situa downstream de 13 HSP procedents de 13 ESTs
diferents. Aquest fet implica que per donar aquesta
prediccó com a bona haguem de prescindir del suport
d'aquests EST, ja que la presència de transcrits a 5' de
l'exó inicial no té sentit biològic. Per que
fa als exons interns, recull els mateixos exons que s'obtenien en
els prediccions anteriors (seqüència incial i extesa),
tot i que ara també hi ha exons addicionals (marcats en
verd), però sense suport d'ESTs.
Pensem que és una predicció prometedora. Per tant, més endavant
realitzarem més comprobacions per verificar si realment hem
trobat l'inici i la terminació del gen que
cercàvem.
- El tercer gen predit no presenta massa suport d'ESTs i tampoc
conincideix amb cap altra predicció.
PREDICCIONS nAC090960:
 Link a la predicció amb nAC090960 |
nAC090960:GENEID
# Gene 1 (Reverse). 3 exons. 272 aa. Score = 9.849256
nAC090960 geneid_v1.1 Internal 1643 1742 0.68 - 2 1
nAC090960 geneid_v1.1 Internal 9256 9846 9.56 - 2 1
nAC090960 geneid_v1.1 First 11160 11283 -0.39 - 0 1
# Gene 2 (Forward). 7 exons. 519 aa. Score = 16.441912
nAC090960 geneid_v1.1 First 25054 25099 -1.93 + 0 2
nAC090960 geneid_v1.1 Internal 31726 31795 -0.60 + 2 2
nAC090960 geneid_v1.1 Internal 53719 53827 1.22 + 1 2
nAC090960 geneid_v1.1 Internal 56997 57064 0.99 + 0 2
nAC090960 geneid_v1.1 Internal 76230 76332 2.88 + 1 2
nAC090960 geneid_v1.1 Internal 87998 88170 2.83 + 0 2
nAC090960 geneid_v1.1 Terminal 116342 117329 11.05 + 1 2
|
nAC090960:GENSCAN
nAC090960 genscan Intr 1453 1607 3.59 - 1 1
nAC090960 genscan Intr 1643 1742 -0.05 - 0 1
nAC090960 genscan Intr 9256 9846 17.94 - 1 1
nAC090960 genscan Intr 10173 10409 11.46 - 0 1
nAC090960 genscan Init 11238 11283 4.50 - 0 1
nAC090960 genscan Init 40190 40261 4.72 + 1 2
nAC090960 genscan Intr 44280 44386 -1.31 + 2 2
nAC090960 genscan Intr 53719 53827 3.47 + 1 2
nAC090960 genscan Intr 55284 55373 0.57 + 2 2
nAC090960 genscan Intr 55477 55551 0.59 + 0 2
nAC090960 genscan Intr 56997 57064 0.98 + 2 2
nAC090960 genscan Intr 61378 61537 0.47 + 1 2
nAC090960 genscan Intr 64064 64173 -0.24 + 1 2
nAC090960 genscan Intr 67569 67648 5.68 + 0 2
nAC090960 genscan Intr 68976 69066 -5.37 + 1 2
nAC090960 genscan Intr 76230 76332 7.26 + 0 2
nAC090960 genscan Intr 87998 88170 12.92 + 1 2
nAC090960 genscan Intr 108258 108394 0.09 + 0 2
nAC090960 genscan Term 116452 117329 16.05 + 2 2
nAC090960 genscan Init 130339 130392 3.63 + 0 3
nAC090960 genscan Intr 140619 140697 3.61 + 2 3
nAC090960 genscan Intr 154902 155180 4.23 + 1 3
nAC090960 genscan Intr 168244 168350 5.11 + 2 3
nAC090960 genscan Intr 170837 171116 3.63 + 1 3
nAC090960 genscan Term 171268 171488 6.02 + 2 3
|
Si no emmascarem la seqüència en absolut:
Geneid:
- Ara Geneid també prediu el gen en
revers, però atribuint-li només tres dels
exons (és més específic que Genscan). Les
posicions predites coincideixen exactament amb els resultats de
Genscan. Però no hi ha ESTs que els suportin i hem de
considerar que estem treballant amb la seqüència sense
emmascarar, cosa que fa disminuir molt la fiabilitat de les
prediccions, ja que els repeats poden donar falsos positius.
- Pel que fa al segon gen predit per Geneid, ara també
dóna exons inicial i terminal, tot i que les posicions no
coincideixen amb els exons incial i terminal predits per Genscan
amb la seqüència lAC090960. El més interessant
però, és la troballa de l'exó indicat pels
13 ESTs (31726-31795). Aquest
exó recull les posicions on es presentaven aquests HSP.
Podem concloure doncs, que no apareixia en les altres prediccions
perquè es trobava emmascarat.
Genscan:
- Les prediccions amb nAC090960 no varien respecte a les de lAC090960
(seqüència sense emmascarar els simple repeats i
low complexity), abans comentades.
L'últim mètode emprat per a obtenir una bona predicció es basa en triar els ESTs més representatius que vàrem trobar en les nostres primeres prediccions i fer córrer un Geneid amb aquests ESTs.
El primer pas va ser obsevar el Ghost View inicial i triar aquells ESTs que suportaven millor les prediccions inicials, intentant no perdre cap hsp (High Score Peptid) que suporti algun dels gens predits ni triar cap hsp que no en suporti cap. Nosaltres van triar els EST següents:
Per tal de triar aquests ESTs i unificar-los en un arxiu en format GFF realitzem, respectivament, les següents comandes:
egrep "gi_7111694_gb_AW499739.1_AW499739 | gi_2077008_gb_AA416870.1_AA416870 | gi_13534099_gb_BG541866.1_BG541866" AC090960.blast.est.spliced.gff > AC090960.est.represent
gawk '{OFS="/t";for(frame=1;frame=<4;frame=frame+1) {print $1,$2,$3,$4,$5,100,"+",frame,1}}' AC090960.est.represent > AC090960.est.represent.gff
Ens instal.lem el Geneid localment i amb la comanda següent el podem fer córrer emprant la informació d'homologia amb els ESTs. Els resultats els obtenim directament en format GFF (comanda -GF):
geneid -GP param/human3iso.param -S AC090960.est.represent.gff AC090960.seq.masked > AC090960.est.represent.geneid.gff
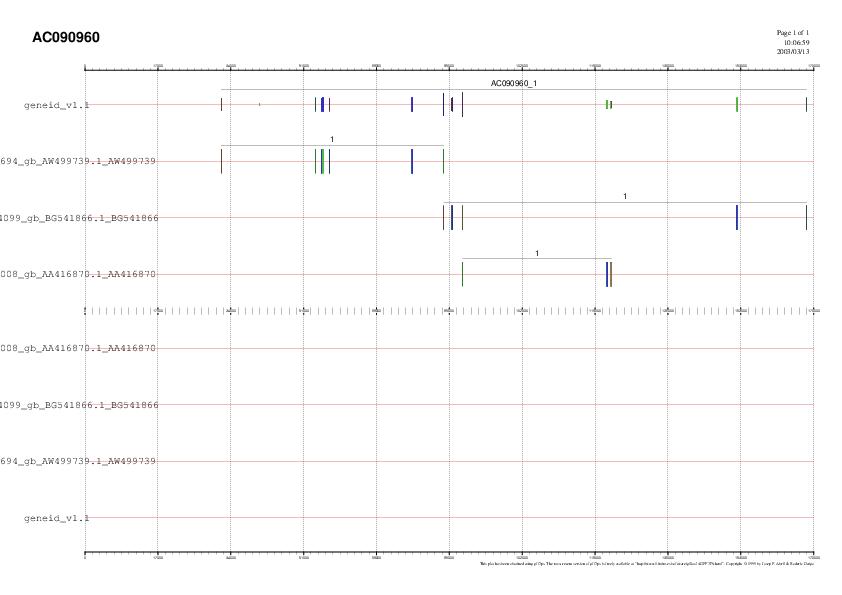
Predicció gènica final, amb Geneid local tenint
en compte els EST més representatius.
Anàlisi dels resultats:
Fent un anàlisi acurat de les noves prediccions, podem
veure que la nova predicció millora substancialment, ja que cada
exó predit bé suportat al menys per l'extrem 3'
d'algun EST (ESTs i
predicció
gènica).
No obstant, poden veure que el Geneid ens ha predit tres exons no suportats per cap EST. Tot i que els seus scores són força baixos comparat amb els de la resta d'exons, no significa que siguin incorrectes.
==========================================================================
EST Posició del hsp Posició de
l'exó suportat
==========================================================================
gi_7111694_gb_AW499739.1_AW499739 31724-31796 31726-31795
gi_7111694_gb_AW499739.1_AW499739 53714-53827 53655-53827
gi_7111694_gb_AW499739.1_AW499739 55477-55551 55477-55551
gi_7111694_gb_AW499739.1_AW499739 55282-55373 55284-55373
gi_7111694_gb_AW499739.1_AW499739 56996-57066 56997-57064
gi_7111694_gb_AW499739.1_AW499739 76230-76333 76230-76332
gi_7111694_gb_AW499739.1_AW499739 83629-83661 83632-83724
gi_2077008_gb_AA416870.1_AE416870 88111-88170 87998-88170
gi_2077008_gb_AA416870.1_AE416870 122167-121797 122587-122721
gi_2077008_gb_AA416870.1_AE416870 122607-122856 122750-122900
gi_13534099_gb_BG541866.1_BG541866 83693-83720
gi_13534099_gb_BG541866.1_BG541866 85564-85616 85495-85616
gi_13534099_gb_BG541866.1_BG541866 85705-85775 85705-85774
gi_13534099_gb_BG541866.1_BG541866 87996-88170
gi_13534099_gb_BG541866.1_BG541866 152211-152253 152111-152253
gi_13534099_gb_BG541866.1_BG541866 168244-168350 168244-168350
==========================================================================
|
========================
Posició Score
=======================
31726-31795 (95.30)
40734-40759 (-2.76)
53655-53827 (100.10)
55284-55373 (97.12)
55477-55551 (99.27)
56997-57064 (96.76)
76230-76332 (101.91)
83632-83724 (186.70)
85495-85616 (95.60)
85705-85774 (99.02)
87998-88170 (201.56)
121670-121690 (9.18)
121717-121796 (56.88)
122587-122721 (39.53)
122750-122900 (36.89)
152111-152253 (101.46)
168244-168350 (99.79)
========================
|
Així doncs, aquesta
és la predicció més fiable de totes les
obtingudes.
Aquests resultats són força
satisfactoris, ja que gairebé cada exó es troba
suportat,
si bé és incomplerta (no presenta exons inicial ni final).
Un cop hem fet les validacions pertinents, analitzarem l'existència de
regions promotores en les prediccions més convincents:
D'aquestes prediccions seleccionades, per a les dues primeres
hem cercat la presència de la TATA-box.
Primerament calculem la posició on es situen els 300pb upstream
a partir de d'inici de la transcripció d'aquestes dues prediccions i obtenim
la seqüència a partir de la base de dades de Golden Path.
En el cas de lAC090960, que s'ha extret de EMBL, cal tenir en compte que al Golden Path
la seqüència que s'obté és la complementària de la d'EMBL.
Com que la seqüència extesa també es va textreure del Golden Path,
no presenta aquest problema.
Accedim a TRANSFAC (base de dades de factors de transcripció) per tal d'obtenir la matriu que necessitem més endavant:
Accedim a RSA toots server
(Regulatory Sequence Analysis Tools) per trobar les TATA box
a partir de la matriu anterior i la seqüència promotora.
Possibles TATA-box pel gen predit per Genscan a eAC090960 (233100-5097).

Possibles TATA-box pel segon gen predit per Genscan a lAC090960 (40190-117329).
En el cas de la tercera predicció seleccionada, no disposem d'exó inicial, de manera que necessitem fer un pas previ per localitzar la regió on es troba el promotor: Si ens fixem, els hsp que es trobem a l'extrem més 5' (31724 - 31796) pertanyen a ESTs que tenen un inici en una situació encara més upstream (fora del nostre contig). Això es pot comprobar mirant l'alineament d'un d'aquests ESTs (per exemple l'EST AU133162) amb la seqüència genòmica, on s'observa que l'alineament no comença des de la primera posició:
>gi|10993701|dbj|AU133162.1|AU133162 [UniGene info] AU133162 NT2RP4 Homo sapiens cDNA clone NT2RP4001437 5'.
Length = 838
Score = 226 bits (114), Expect = 3e-54
Identities = 114/114 (100%)
Strand = Plus / Plus
Query: 53714 tataggaaagaatgggaagaactatttgtaaacaacaattacttggcaacaataaggcag 53773
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct: 424 tataggaaagaatgggaagaactatttgtaaacaacaattacttggcaacaataaggcag 483
Query: 53774 aaggggattaatgggcagctgagaagcagcaggttccgcagcatttgctggaag 53827
||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct: 484 aaggggattaatgggcagctgagaagcagcaggttccgcagcatttgctggaag 537
Aquesta comprobació també es pot fer aplicant la comanda:
./parseblast -F eAC090960.blast.est | grep AU133162 | sort +3n > eC090960.estAU.posicio
L'opció -F de Parseblast ens dóna les posicions de la seqüència genòmica i la seva correspondència a les posicions de la seqüència de l'EST.
Vist això cercarem l'inici d'aquest EST, encara
que es trobi fora del nostre contig (inclús extenent-lo
30,000pb pels extrems).
Busquem la seqüència
complerta de l'EST AU133162 al
NCBI,
per l'opció nucleotide: AU133162.ncbi
Un cop tenim la seqüència complerta, la mapem al
Golden Path per veure on es troben les bases inicials dins
del cromosoma.
Utilitzem el BLAT,
que és una opció de GoldenPath que funciona com el BLAST de NCBI però
per buscar la localització la seqüència al Golden
Path. S'introdueix la seqüència den nostre EST i es
compara contra el Genoma.
En aquest fitxer de la seqüència, els nucleòtids marcats en blau marí corresponen als nucleòtids que es troben alineats amb la nostra seqüència. Per contra, els que apareixen en blau clar representen els senyals de splicing (donors i acceptors). Per últim, les bases negres són aquelles que no s'han pogut alinear.
Mirem les posicions on l'EST presenta alineament:
POSICIÓ A L'EST POSICIÓ AL GENOMA
=============== =================
1 - 161 17568910 - 17568747
357 - 428 17409894 - 17409823
429 - 537 17387899 - 17338791
538 - 627 17386334 - 17386067
628 - 699 17386138 - 17386067
700 - 767 17384621 - 17384554
Tenint en compte que el nostre contig ampliat ocupa les posicions
17471617 - 17234874, veiem com el primer fragment d 'EST (1-161)
està fora del contig, que representen l'inici de
transcripció putatiu del nostre gen.
Si mirem la seqüència d'aquest primer fragment, veiem que no
comença per ATG. Això en fa pensar que podria
haver-hi un fragment UTR (Untranslated Region), que es
transcriu però no es tradueix, abans de l'aparició
de l'inici de traducció.
Ara ja podem buscar regions promotores en les 300pb
upstream d'aquest fragment de l'EST AU133162 (1- 161):
17568610- 17568747.
Creem un fitxer en format FASTA amb aquestes
300 bases: AU133162.prom300.
A continuació ens conectem al TRANSFAC, per tal de trobar les potencials regions promotores amb el MatInspectorV2.2 utilitzant les TRANSFAC 4.0 matrices. Introduïm la nostra seqüència i enviem la nostra petició amb els paràmetres que hi ha per defecte:
Sense modificar els paràmetres, ja obtenin un nombre de resultats addient.
Per generar el gràfic convertim l'output obtingut de TRANSFAC (AU133162prom.transfac) a format GFF mitjançant la següent comanda gawk:
gawk '{OFS="\t";print "Promotors", $10, $1, $3, $11, $8, $4, ".", $1 }' AU133162prom.transfac > AU133162prom.transfac.gff .
Un cop la tenim en format GFF (AU133162prom.transfac.gff), generem un gràfic que representi aquestes possibles regions promotores a través del programa gff2ps. En aquest cas, per tal de poder distingir els diferents tipus de promotors, donem un paràmetre addicional al programa: es tracta d'un fitxer on s'associa cada promotor a un color diferent (proporcionat per G.Parra): matrices.gff2ps.param
Passem aquest fitxer pel gff2ps per tal d'obtenir un resultat més visual:
./gff2ps -C matrices.gff2ps.param AU133162prom.transfac.gff > AU133162prom.transfac.ps
El gràfic es presenta al link següent:

|
Llegenda:
|
Veiem doncs, que a la regió 5' del transcrit que hem seleccionat hi ha diversos candidats a ser el promotor del nostre gen. Malgrat tot, cal tenir en compte que, d'entre aquests candidats, només podríem acceptar-ne un com a vàlid i que la fiabilitat d'aquestes predicions no és molt alta.
Un cop hem obtingut les diferents prediccions que hem anat explicant al llarg de tota la web, farem un estudi dels productes proteics.
Recuperant aquests transcrits, vam realitzar un Blastp(protein-protein) per a cadascun d'aquests tres transcrits obtenint-ne els següents resulats:
No obstant, ens centrarem en l'estudi de les proteïnes que té més homologia amb aquest últim Blastp (amb homologia amb ESTs) ja que, com s'ha comentat amb anterioritat, és la millor de totes les prediccions realitzades (veure "Anàlisi dels resultats" del punt 4 de l'apartat "Validació de les prediccions").
De tots els hits que trobem, triem el primer ja que té el major score (599) i el menor E-value (e-170). No obstant, la resta de hits també presenten, en general, uns bons valors. Val a dir que aquest primer hit coincideix amb el millor hit que apareix a la cerca amb la predicció trobada a partir de la seqüència extesa (eAC090960.genscan1), tot i que en aquesta segona predicció, el nombre de residus coincidents a l'alineament és menor, l'score és de 325 i E-value de 9e-88 (la predicció correspon a la mateixa proteïna però aquesta està pitjor predita).
Es tracta del membre 5 de TBC1 domain family , que és el producte gènic de KIAA0210. Presenta una identitat del 76% amb la nostra seqüència. Al ser tant elevada, ens indica que el nostre transcrit pot ser homòleg a la proteïna trobada, idea que es troba suportada pel fet de tenir un E-value tan baix (e-171). No obstant, aquest valor només ens informa de la possibilitat de trobar aquesta proteïna per atzar i no per homologia però no podem assegurar al cent per cent que sigui una proteïna homòloga.
Observem l'alineament corresponent:
Score = 602 bits (1551), Expect = e-171
Identities = 309/406 (76%), Positives = 319/406 (78%), Gaps = 69/406 (16%)
Query: 1 GADSNKNGRRTSSTLDSEGTFNSYRFLLQYIRMELLSKQRNFNKRILMLWFLSYRKEWEE 60
G DSNKNGRRTSSTLDSEGTFN SYRKEWEE
Sbjct: 32 GGDSNKNGRRTSSTLDSEGTFN------------------------------SYRKEWEE 61
Query: 61 LFVNNNYLATIRQKGINGQLRSSRFRSICWKLFLCVLPQDKSQWISRIEELRAWYSNIKE 120
LFVNNNYLATIRQKGINGQLRSSRFRSICWKLFLCVLPQDKSQWISRIEELRAWYSNIKE
Sbjct: 62 LFVNNNYLATIRQKGINGQLRSSRFRSICWKLFLCVLPQDKSQWISRIEELRAWYSNIKE 121
Query: 121 IHITNPRKVVGQQDLMINNPLSQDEGSLWNKFFQDKELRSMIEQDVKRTFPEMQFFQQEN 180
IHITNPRKVVGQQDLMINNPLSQDEGSLWNKFFQDKELRSMIEQDVKRTFPEMQFFQQEN
Sbjct: 122 IHITNPRKVVGQQDLMINNPLSQDEGSLWNKFFQDKELRSMIEQDVKRTFPEMQFFQQEN 181
Query: 181 VRKILTDVLFCYARENEQLLYKQGMHELLAPIVFVLHCDHQAFLHASESAQPRQNKANSA 240
VRKILTDVLFCYARENEQLLYKQGMHELLAPIVFVLHCDHQAFLHASESAQP
Sbjct: 182 VRKILTDVLFCYARENEQLLYKQGMHELLAPIVFVLHCDHQAFLHASESAQP-------- 233
Query: 241 SLIIQKLHILIACIAFLSEEMKTVLNPEYLEHDAYAVFSQLMETAEPWFSTFEHDGQKGK 300
SEEMKTVLNPEYLEHDAYAVFSQLMETAEPWFSTFEHDGQKGK
Sbjct: 234 -----------------SEEMKTVLNPEYLEHDAYAVFSQLMETAEPWFSTFEHDGQKGK 276
Query: 301 ETLMTPIPFARPQDLGPTIAIVTKVNQIQDHLLKKHDIELYMHLNRLEIAPQIYGFSEMR 360
ETLMTPIPFARPQDLGPTIAIVTKVNQIQDHLLKKHDIELYMHLNRLEIAPQIYG +R
Sbjct: 277 ETLMTPIPFARPQDLGPTIAIVTKVNQIQDHLLKKHDIELYMHLNRLEIAPQIYGLRWVR 336
Query: 361 IYLGFLGLLCFVRRWPVRKEKI------DDSQHLGGLSFVLIRMLL 400
+ G R +P++ + D LG + ++ + MLL
Sbjct: 337 LLFG--------REFPLQDLLVVWDALFADGLSLGLVDYIFVAMLL 374
Veiem que el 76% d'homologia no és uniforme; és a dir, no és que hi hagi una homologia constant al llarg de la seqüència, sinó que hi ha fragments amb homologia del 100% i zones amb 0% (gaps). Aquest fet ens porta a pensar que dificilment és una proteïna homòloga, ja que en aquests casos el responsable de la manca d'homologia són els nucleòtids missmatches al llarg de la seqüència i no la presència de gaps tant extensos com en el nostre cas. Per tant, una primera hipòtesi és que es tracta de la mateixa proteïna però que presenta defectes de seqüenciació.
Per tal de validar la nostra hipòtesi hem de mapejar aquesta proteïna. Accedim al Ensembl on introduim el nom de la proteïna. En el resultat podem veure que el seu inici es troba en el contig anterior (AC090644) però s'extén al nostre (AC090960). Per tant, podem afirmar que es tracta de la mateixa proteïna (TBC1D5). A més, confirmem que la predicció que hem acceptat com a més versemblant efectivament no té exó inicial (recordem que hem trobat els possibles promotors a partir d'un EST, l'inici del qual també es troba fora del contig analitzat).
Passem a l'anàlisi bàsic d'aquesta proteïna mitjançant la base de dades Pfam (Protein FAMilies database of aligments and HMMs). Veiem que forma part d'una família de proteïnes present només en eucariotes. Fou inicialment descrita en els llevats Gyp6 i Gyp7. Es tracta d'una GTPasa, amb una funció activadora de GTP en les RAb-like small GTPases. Concretament, al Genoma humà s'han descrit 160 dominis TBC diferents.
Entrant al NCBI, hem obtingut també el seu cDNA NM_014744.
Després del complex procés d'anàlisi descrit,
podem concloure que en el contig AC090960 es localitza part del gen
KIAA0210, que codifica per a la GTPasa TBC1D5.
L'extrem 5' es troba en el contig anterior (AC090644), i per tant, hem
hagut de cercar el promotor putatiu en aquest últim contig,
mitjançant l'obtenció de 300 pb upstream de
l'extrem 5' de l'EST AU133162, que és un dels que recull el
primer hsp trobat, i que té el seu inici al contig AC090644.
A partir de tots els diferents mètodes emprats per obtenir prediccions gèniques millorades que estaven al nostre abast, concluim que la predicció més verossímil correspon a la que s'ha obtingut a partir de la consideració per part del Geneid dels 3 EST que recullen tots els hsp apareguts.
El gen trobat consta de 17 exons, localitzats a les següents posicions:
Exó 1: 31726-31795
Exó 2: 40734-40759
Exó 3: 53655-53827
Exó 4: 55284-55373
Exó 5: 55477-55551
Exó 6: 56997-57064
Exó 7: 76230-76332
Exó 8: 83632-83724
Exó 9: 85495-85616
Exó 10: 85705-85774
Exó 11: 87998-88170
Exó 12: 121670-121690
Exó 13: 121717-121796
Exó 14: 122587-122721
Exó 15: 122750-122900
Exó 16: 152111-152253
Exó 17: 168244-168350

Comprobem si aquesta predicció de proteïna coincideix amb l'anotació existent a Ensembl:
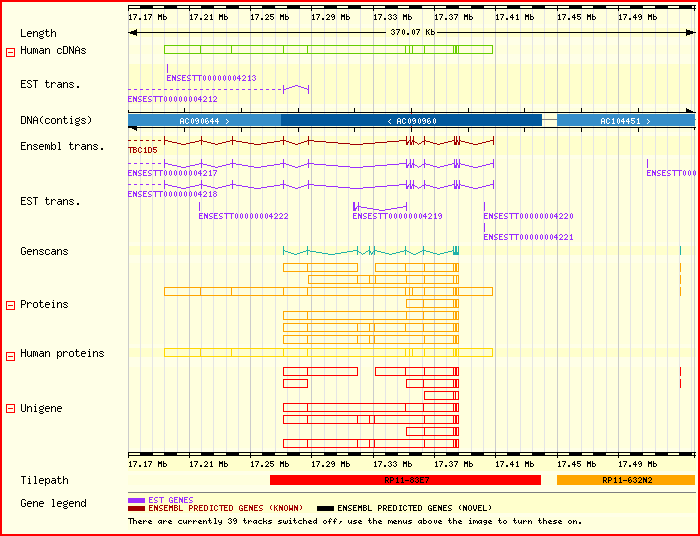
Visió general de la regió estudiada,
on també apareixen els contigs contigus.
Entre d'altres, apareixen els ESTs coneguts, la predicció gènica
d'Ensembl i la proteïna codificada.
Podem veure com Ensembl té mapada a la nostra regió un gen que és congut,
el TBC1D5, que efectivament
coincideix amb el resultat de la cerca d'homologia per BLASTP amb el nostre transcrit predit per Geneid.

Carme Cortina Duran (carme.cortina01@campus.upf.es)
Sara Fernández Castillejo (sara.fernandez02@campus.upf.es)
Voldríem agrair la col·laboració,
seguiment i ajut proporcionat per Genís Parra i Josep
F. Abril. Gràcies per les visites a qualsevol hora i per les
incomptables converses via e-mail.
En segon lloc, també donem les gràcies pels
ànims i suport a la resta de grups d'Anàlisi de Seqüències
Genòmiques durant hores infinites passades a les aules d'informàtica:
A.Boix, A.Edo, M.Ferrer, C.Fuentes, I.Joval, M.Pellicer, M.Oliver,
P.Roger i A.Segura.
Gràcies a tots!
C.Cortina, S.Fernández
Bioinformàtica (4rt Biologia UPF).
Barcelona, Març-Abril 2003.