ANÀLISI DE FAMÍLIES DE PROTEÏNES
PROTEÏNA LIFEGUARD
Introducció
Aquest treball caracteritza una família de proteïnes, partint d'una
seqüència problema (Lifeguard Q8K1F6 de Mus musculus). En primer
lloc, analitzarem les seqüències de proteïnes que presentin un bon
alineament amb la nostra proteïna (Programes: Swissprot, BLASTP, ClustalW,
Pfam). Seguidament, estudiarem les relacions filogenètiques entre les
proteïnes escollides tot relacionant les funcions respectives (Programes:
ClustalW (Pasteur i EBI), TreeView).
I per últim, estudiarem els gens codificants pels membres d'aquesta
família proteica (cromosoma, exons) per humà i ratolí (Programes:
ENSEML, NCBI).
Entrem la nostra proteïna a la base de dades de Swissprot.
Hi trobem informació sobre funcions, seqüència d'aminoàcids en format FASTA i
format TrEMBL, origen, referències...
Prenem la seqüència en format FASTA i fem córrer el programa BLASTP. Aquest programa compara la proteïna entrada amb totes les de la base
de dades en parells (dos a dos) a l'atzar, és a dir, la proteïna problema amb una proteïna a l'atzar, després respecte una altra
proteïna també a l'atzar, i així successivament. Atribueix a cada alineament una puntuació ("score") i un valor d'esperança
("E-value").
BLASTP té un match amb altres bases de dades de dominis proteics (Pfam, COG).
En les bases de dades de dominis, la proteïna que entrem és comparada amb el
conjunt de proteïnes semblants que troba dins la base
de dades. Les seqüències d'aminoàcids conservades són més
significatives que si usem BLASTP ja que estan
contextualitzades únicament dins el grup de proteïnes
més similars.
Domini conservat
Les barres en vermell indiquen la predicció del domini conservat per a la proteïna "Lifeguard Q8K1F6"
de Mus musculus, segons les bases de dades Pfam i COG. El domini UPF0005 pertany a una família proteica no caracteritzada
("Uncharacterized Protein Family") segons la base de dades Pfam. I segons la base de dades COG el
domini COG0670 és un domini de proteïna integral de membrana que interacciona amb FtsH.
Les dues bases de dades suporten que el domini de conservació es troba en l'extrem C-terminal de la
seqüència proteica, aproximadament entre els aminoàcids 140 i 310.

Escollim les seqüències proteiques de diferents espècies, inclosa la
proteïna Q8K1F6 amb funcions diverses que compleixin un "score" més alt (millor
alineament) i una esperança ("E-value") màxima de 0.001 (si superem aquest rang
els valors no són estadísticament significatius). Obtenim així una mostra
representativa de diferents espècies per tal de relacionar-les
evolutivament.
| Proteïna |
espècie |
score |
E-value |
| Lifeguard; Q8K1F6 |
Mus musculus |
545 |
e-154 |
| Lifeguard; NMP35 |
Rattus norvegicus |
521 |
e-147 |
Lifeguard; KIAA0950 protein |
Homo sapiens |
501 |
e-140 |
| NMDA receptor glutamate-binding chain; LAG protein |
Mus musculus |
177 |
3e-43 |
| NMDA receptor glutamate-binding subunit |
Rattus norvegicus |
176 |
6e-43 |
| NMDA selective glutamate receptor; CG814-PA |
Drosophila melanogaster |
164 |
3e-39 |
| NMDA receptor associated protein |
Caenorhabditis elegans |
149 |
e-34 |
| NMDA1; CG3798-PA |
Drosophila melanogaster |
148 |
2e-34 |
| agCP6292 |
Anopheles gambiae |
148 |
2e-34 |
| transmembrane protein OTMP |
Ovis aries |
78 |
4e-13 |
| homolog of Drosophila NMDA1 protein, putative |
Plasmodium falciparum |
77 |
7e-13 |
| NMDA receptor-like protein; CMLV006 |
Camelpox virus |
75 |
2e-12 |
| z-protein |
Homo sapiens |
72 |
2e-11 |
Mitjançant el programa ClustalW (EBI) realitzem l'alineament múltiple de les
seqüències proteiques escollides per tal de limitar l'alineament a la zona
conservada.
Aquest és el resultat.
Paral.lelament, hem cercat la seqüència del domini UPF0005 (Mus musculus) a la base de dades Prosite i
comprovem que realment coincideix amb la seqüència conservada deduïda a partir de l'alineament múltiple anterior (ClustalW).
FPAGHHEHFTTFSWDDQKVRRLFIRKVYTILLVQLLVTLAVVALFTFCDVVKDYVQANPGWYWASYAVFFATYLTLACCS
pfam:UPF0005 <================================================================
GPRRHFPWNLILLTIFTLSMAYLTGMLSSYYNTTSVLLCLVITALVCLSVTIFSFQTKFDFTSCQGVLFVLLMTLFFSGL
pfam:UPF0005 ================================================================================
LLAVLLPFQYVPWLHAVYAVLGAGVFTLFLAFDTQLLMGNRRHSLSPEEYIFGALNIYLDIIYIFTFFLQLYGTNRE
pfam:UPF0005 ===========================================================================>
Prenem les seqüències seleccionades prèviament per tal de relacionar-les
evolutivament en un arbre filogenètic (pel mètode de "Neighbour joining").
Els dos programes clau són: ClustalW (Pasteur) i TreeView. L'opció "bootstraping"
per l'arbre "NJ" calcula 1000 vegades l'alineament previ a l'atzar i dóna una mesura
de robustesa (nombre de vegades que es genera el mateix arbre), en el nostre cas,
dóna exactament el mateix arbre i amb una elevada robustesa (pròxima a 1000).
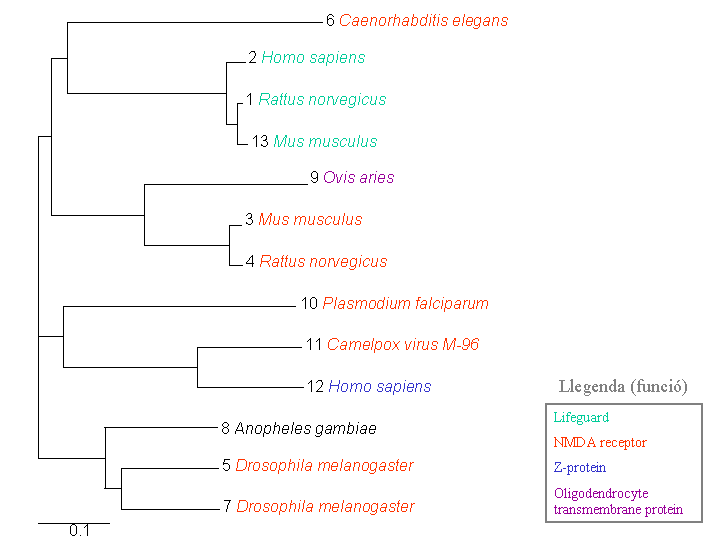
En l'arbre, observem una primera ramificació on s'agrupen d'una banda les lifeguards de
mamífer relacionades amb les NMDAs també de mamífer i de C. elegans; d'altra
banda una segona ramificació composta per Z-protein humana directament relacionada amb
Camelpox virus i secundàriament amb P. falciparum,
espècies que infecten i parasiten l'home respectivament; i un tercer cluster protagonitzat
per proteïnes d'insectes (NMDAs de Drosophila sp. i agCP6292 d'Anopheles
gambiae).
Comentarem les funcions de les diferents proteïnes. Com veiem a l'arbre,
les proteïnes a distància filogenètica més propera són les lifeguards (de
Rattus norvegicus, Homo sapiens i Mus musculus).
La proteïna lifeguard pot unir-se al receptor de Fas però no regula l'expressió
de Fas ni interfereix amb la unió d'un agonista. Lifeguard és una molècula
anti-apoptòtica (protegeix les cèl.lules de l'apoptosi induïda per Fas) (1).
La maquinària intracel.lular responsable de l'apoptosis és similar en totes les
cèl.lules animals. Aquesta maquinària depèn d'una família de proteases que
hidrolitza les proteïnes diana en el seu costat carboxil dels àcids aspàrtics
(d'aquí el seu nom, c-caspasa). Les caspases són sintetitzades com a precursors
inactius (procaspases) que poden activar-se per l'esmentada ruptura. Així,
un cop desencadenada l'activació, té lloc una cascada de caspases (la qual
provoca la degradació de proteïnes estructurals, de factors de transcripció,
etc, acabant amb la mort cel.lular). L'activació de les procaspases pot ser
induïda des de l'exterior de la cèl.lula mitjançant l'activació de receptors
mortals presents a la superfície cel.lular. El lligand de Fas unit al
receptor Fas activa la cascada (2). Lifeguard és present a
les sinapsis de l'adult del Sistema Nerviós Central.
En humà, lifeguard (KIAA0950) s'expressa
en tots els teixits excepte melsa i placenta (1).
Els receptors NMDA
(N-metil-D-aspartat) són els típics receptors ionotròpics (que formen un
canal iònic) activats per l'aminoàcid glutamat. El glutamat és el
neurotransmissor excitador més important en el SNC. Quan el receptor NMDA és activat,
augmenta la permeabilitat de la membrana plasmàtica pel Calci. L'entrada de Calci
a l'interior cel.lular comporta innumerables efectes postsinàptics. En repòs aquest
receptor està bloquejat pel Magnesi (normalment present al medi extracel.lular).
La suma d'ambdues propietats (permeabilitat al Calci i bloqueig pel Magnesi) el
fan responsable de fenòmens de naturalesa plàstica implicats en els processos
d'aprenentatge i memòria (3). El fet que Camelpox virus tingui
aquest receptor suggereix que l'ha adquirit per infecció d'una cèl.lula hoste,
possiblement d'Homo sapiens, per la seva proximitat evolutiva (reflectida
a l'arbre filogenètic).
Z-protein és una proteïna eucariota integral de membrana relacionada amb GBP
(Glutamate Binding Protein), és a dir amb NMDA. Té 7 dominis transmembrana,
que pertanyen a la família BI1 (supressor apoptòtic de mamífers). La base de
dades GO prediu la seva funció: regulador de l'apoptosi.
La proteïna transmembrana d'oligodendròcit (OTMP) també és una GBP. És una proteïna
integral de quatre hèlix amb un loop extern i amb les regions N i C-terminals
localitzades dins la cèl.lula. Es suggereix que OTMP és un receptor de
senyalització amb un paper en la mielinogènesi (4).
La proteïna d'Anopheles gambiae és de funció desconeguda.
Tant les proteïnes lifeguard com els receptors NMDA contenen el domini BI1
(Bax inhibitor-1). El domini BI1 és similar a la família de proteïnes
intracel.lulars Bcl-2 (que contribueix a regular l'activació de les
procaspases). Alguns membres d'aquesta família (com el mateix Bcl-2 o
Bcl-XL) inhibeixen l'apoptosi bloquejant l'alliberament del citocrom C
(transportador d'electrons) del mitocondri (2).
Existeix una homologia significativa en la meitat C-terminal entre les proteïnes:
- Neural Membrane Protein 35 (NMP) de Rattus sp.
- Glutamate Binding Protein de Rattus sp.
- Drosophila NMDA receptor-associated protein
- dues proteïnes de funció desconeguda de C. elegans
demostrada per un article [PubMed]
[Free Full Text]
(1) trobat a la base de dades PubMed. Alhora, el contingut d'aquest article reflecteix
una correlació entre NMDA i l'expressió de Fas i el seu lligand (FasL), i
un important paper protector anti-apoptòtic de LFG implicat en aquesta correlació. S'ha
observat expressió de Fas i FasL després d'injectar NMDA a l'hipocamp de ratolí (5).
Malalties com leucèmia i Alzheimer (amb un component apoptòtic i el cromosoma 12q13 humà
implicat) l'expressió i modulació de LFG pot tenir un important ús terapèutic quan
la protecció davant de Fas és requerida.
Hem ampliat la informació d'aquest arbre amb altres proteïnes d'interès (que
apareixen en el tercer bloc) i, a més a més, amb una seqüència d'un herpes
virus humà (relacionat amb Lifeguard Q8K1F6 en l'article: (6)).
En aquest article hi ha evidències d'homologia entre Lifeguard humana i la proteïna HCMVA
(pertanyent a la família US12) de l'herpesvirus 5 humà, suggerint un paper anti-apoptòtic.
Aquesta proteïna és integral de membrana, interacciona amb FtsH, i conté el domini
UPF0005 amb subdomini BI1.
En l'alineament múltiple
de l'article citat es reflecteix el domini conservat UPF0005 entre membres de la família
US12 d'Herpesvirus, lifeguard humana, i dues proteïnes humanes addicionals.
Mitjançant el programa ClustalW (EBI) realitzem un alineament múltiple de totes aquestes
seqüències i una posterior representació de les relacions evolutives entre les diferents
espècies implicades:
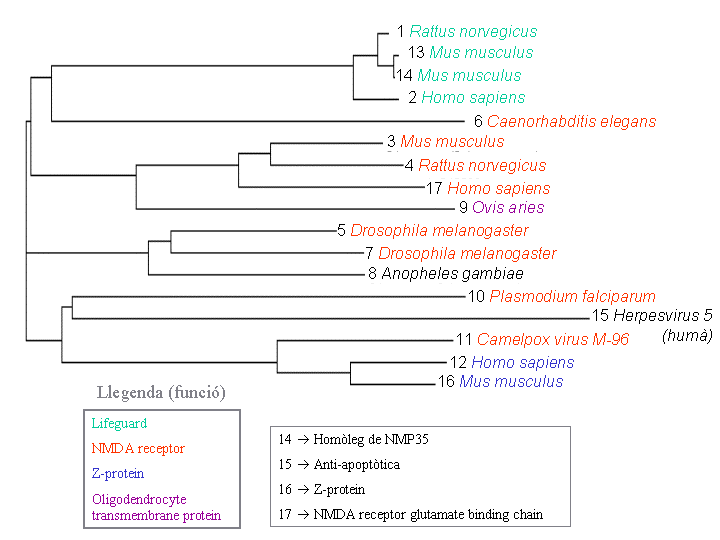
En aquest segon arbre, es manté la mateixa agrupació d'espècies i funcions plasmada en el
primer arbre. Les quatre proteïnes (l'homòleg de NMP35 (14) i la Z-protein (16) de Mus musculus,
NMDA receptor glutamate-binding chain (17) de Homo sapiens, i una proteïna relacionada
d'Herpesvirus (15) humà) afegides s'integren en l'arbre sense desestructurar aquesta distribució.
D'aquesta manera, la proteïna número 14 es relaciona directament amb la lifeguard (13)
de la mateixa espècie (Mus musculus); la número 16 directament amb la seva homòloga
Z-protein de Homo sapiens; la número 17 amb NMDA de rosegadors; i finalment, la
número 15 dins el mateix grup que P. falciparum i Camelpox virus
reflectint la possible adquisició de gens d'hostes humans, entre altres espècies, al
llarg de l'evolució.
Entrant a l'ENSEMBL la seqüència FASTA de la nostra proteïna de partida
(Q8K1F6) per tal de córrer un BLASTP (córrer la proteïna contra la base de dades
de l'ENSEMBL). El resultat és que el millor alineament no és suficientment
significatiu per constatar que ENSEMBL conté la proteïna. Per tant, no podem
mostrar l'estructura exònica de Q8K1F6.
Ja que no tenim un representant de
Lifeguard en Mus musculus, partim de la Lifeguard d'Homo
sapiens (KIAA0950). Prenem la seqüència humana en format FASTA i correm un
BLASTP contra les proteïnes de Mus musculus d'ENSEMBL. Així trobem la
seva proteïna homòloga en Mus musculus: Lifeguard Q9D6K4.
De la
mateixa manera, a partir de la seqüència FASTA de la proteïna NMDA receptor
glutamate-binding chain (Lag protein) de Mus musculus, trobem la seva
homòloga humana: NMDA receptor glutamate-binding chain (GRINA).
Segons la base de dades Pfam, totes les proteïnes de la taula anterior
comparteixen el domini conservat UPF0005. Els membres de la família UPF0005 es preveu
que contenen 7 dominis transmembrana i que comparteixen la regió compresa entre el
principi del 3er domini fins meitat del 4rt (1). La cerca per ENSEMBL de les
proteïnes de Mus musculus mostra el domini conservat BI-1 (Bax inhibitor-1),
mentre que per les proteïes de Homo sapiens mostra el domini conservat
UPF0005. La base de dades INTERPRO caracteritza BI-1 com a subdomini del domini
conservat UPF0005.
Segons la bibliografia (7), BI-1 és una proteïna integral de membrana
evolutivament conservada en mamífers, implicada en la supressió de l'apoptosi.
Conté múltiples segments transmembrana, com podem veure en totes les proteïnes de
la taula.
Trobem evidència d'Splicing Alternatiu en Z-protein i en Lifeguard
d'Homo sapiens. El primer cas, quatre transcrits generen quatre isoformes
diferents de la Z-protein. En el segon cas, es tradueixen dues isoformes de la
Lifeguard a partir de dos transcrits.
4.Referències
(1) Somia NV, Schmitt MJ, Vetter DE, Antwerp DV, Heinemann SF, Verma IM.
LFG: An anti-apoptotic gene that provides protection from Fas-mediated cell death.
Proc. Natl. Acad. Sci. USA. 1999 October 26;96(22):12667–12672.
(2) Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P.
Molecular Biology of the Cell. New York: Garland Science, 2002.
(3) Lerma J, Mora J, Prieto JS. Sinapsis monoaminérgicas y
peptidérgicas. In: Delgado JM, Ferrús A, Mora F, Rubia FJ. Manual de Neurociencia.
Madrid: Editorial Síntesis, 1998;233-234.
(4) Szuchet S, Plachetzki DC, Eaton KS.
Oligodendrocyte transmembrane protein: a novel member of the glutamate-binding protein subfamily.
Biochem Biophys Res Commun 2001 May 18;283(4):900-7.
(5) Shin SW, Park JW, Suh MH, Suh SI, Choe BK.
Persistent expression of Fas/FasL mRNA in the mouse hippocampus after a single
NMDA injection. Journal of Neurochemistry 1998;71,1773-1776.
(6) Holzerlandt R, Orengo C, Kellam P, Albà MM.
Identification of New Herpesvirus Gene Homologs in the Human
Genome. Genome Res 2002 Nov;12(11):1739-48.
(7) Xu Q, Reed JC.
Bax inhibitor-1, a mammalian apoptosis suppressor identified by
functional screening in yeast. Mol Cell
1998 Feb;1(3):337-46.
5.Autores
Marta Beltran Domingo (marta.beltran01@campus.upf.es)
Ariadna Echenique Vergés (ariadna.echenique01@campus.upf.es)



