Les selenoproteïnes són aquelles proteïnes que contenen com a mínim un aminoàcid de selenocisteïna (Sec/U) en la seva seqüència. Aquests aminoàcids contenen seleni en la seva estructura, un micronutrient esencial per a tots els éssers vius. Les selenocisteïnes estan codificades pel codó UGA, que en condicions normals és un codó STOP.
Com que la major part d’aquestes proteïnes es troben incorrectament anotades, el principal objectiu és predir, identificar i analitzar les selenoproteïnes que pertanyen a l’espècie Atractosteus spatula. Per a assolir el nostre objectiu hem utilitzat el peix zebra (Danio rerio) com a organisme de referència, ja que és una espècie més estudiada i coneguda.
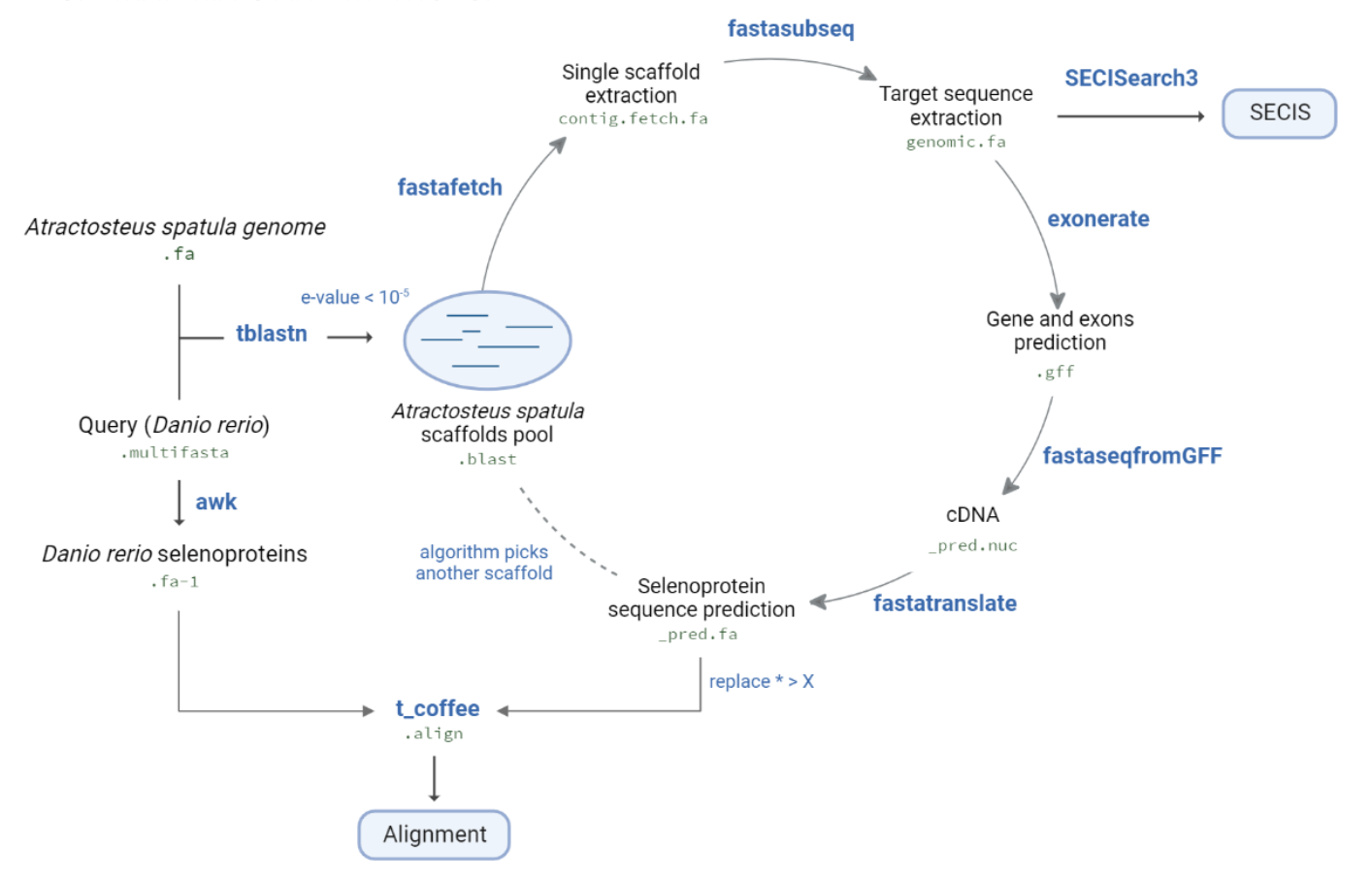
Per tal de fer-ho, hem creat un programa automatitzat per fer l’alineament entre la seqüència coneguda de cada proteïna del Zebrafish i el nostre organisme. Així, hem emprat les següents eines informàtiques: tBLASTn, fastafetch, fastasubseq, exonerate, fastaseqfromGFF, fastatranslate, t-coffee i SECISSearch 2.0.
Amb el script desenvolupat, s’han determinat 39 selenoproteïnes, 10 proteïnes homòlogues que contenen l’aminoàcid Cys i 9 proteïnes que corresponen a la maquinària de transcripció de les selenoproteïnes, la qual cosa permet entendre de forma acurada i precisa el selenoproteoma d’Atractosteus spatula.
El seleni (Se) és un nutrient essencial per vertebrats, altres animals i, fins i tot, microorganismes. Tenir uns nivells equilibrats d’aquest metall és fonamental per molts processos o sistemes cel·lulars com el desenvolupament o el sistema immunitari. Una deficiència de Sel s’associa amb malalties cardíaques, com la malaltia de Keshan, o càncer.
Les selenoproteïnes són proteïnes que tenen un aminoàcid especial, la selenocisteïna (Sec, U). S’anomena també com a aminoàcid 21, i es diferencia de la cisteïna pel canvi de l'àtom de sofre per un àtom de seleni, és per això que la cisteïna i la selenocisteïna tenen propietats químiques similars. Tot i això, a nivell molecular la Sec és molt més cara que la Cys, però això es veu compensar perquè és més reactiva que la Cys i més resistent davant una oxidació permanent. Al crear un enzim amb Cys en comptes de Sec, té una vida mitja inferior i s’ha de produir un de nou.

Fig. 1. Cisteïna i Selenocisteïna
La major part del seleni del nostre cos es troba en forma de selenoproteïnes, en el cos humà en tenim 25, però en cada organisme el nombre és molt diferent. Cadascuna d’aquestes selenoproteïnes té funció pròpia i diferent de les altres, però totes actuen com enzims catalitzadors de reaccions oxido reductores, i funcionen en el procés de l’homeòstasi redox com a defensa antioxidant. Aquestes proteïnes actuen com a reductors defensen a les cèl·lules quan hi entren oxidants que roben electrons a les biomolècules cel·lulars.
El codó codificant per la selenocisteïna no és propi, és el codó UGA, el qual sempre té funció d’STOP pero degut a la presència de gens codificants, les selenoproteïnes aconsegueixen canviar el significat d’aquest codó i inserir una molècula de Sec.
La maquinària de traducció detecta aquests codons com a diferents per la presència de la regió SECIS present a l’extrem 3’ UTR, que es plega d’una manera específica per tal de ser detectada i informar que s’ha de produir una recodificació del senyal STOP a Sec.

Un cop el senyal SECIS ha indicat que és necessari fer una recodificació, hi ha tot un pathway constituït per múltiples proteïnes que s’encarreguen de la síntesi i incorporació de la Sec. Cal destacar que la síntesi es fa directament sobre el tRNAsec, únic d’aquest aminoàcid.

Taula 1. Proteïnes de síntesi i incorporació de Sec
Trobem selenoproteïnes en els tres dominis de la vida (eucariotes, bacteris i arqueobacteris), però el seu nombre varia entre espècies. Utilitzant els genomes seqüenciats podem traçar l’evolució de les selenoproteïnes reconstruint quines estan presents en cada organisme i com van evolucionant entre espècies.
Al mirar la història antiga d’eucariotes, trobem que hi ha genomes, com els de vertebrats, que tenen un alt nombre de selenoproteïnes, però hi ha d’altres , com els insectes, que les han perdut totes, per exemple Drosophila willistoni havia perdut la maquinària involucrada en el metabolisme de les selenoproteïnes (tRNA-Sec, SPS2, EFSec, SecS), i en conseqüència va anar perdent la resta progressivament. També observem la desaparició de les selenoproteïnes en fongs, tot i que encara hi ha alguns que en conserven. Aquesta doble evolució dels fongs i els insectes cap a la desaparició de les selenoproteïnes ha estat convergent.
La causa de la desaparició de les selenoproteïnes pot ser diversa, l’evolució és dinàmica i els selenoproteomes responen a diverses forces evolutives, per exemple la disponibilitat de Sec: si un organisme depenent passa a viure en un lloc on no hi ha seleni, serà negatiu. D’altre banda, segons l’estil de vida de cada organisme serà més o menys necessària la defensa antioxidant que donen les selenoproteïnes i, per tant, les forces evolutives actuar d’una manera o d’un altre, en conseqüència. Per últim, la Sec està sempre en competició a nivell molecular amb el codó STOP, fet que propicia també la seva desaparició.
Per tal d’identificar les Sec en el genoma d’un organisme no podem només mirar el seu codó, ja que com ja hem esmentat no en té un específic perquè el comparteix amb el codó STOP.
Podem identificar-les a partir de la predicció de les SECIS i buscant una Sec propera. A partir de l’estudi d’aquests elements s’han fet diversos models mitjançant diferents mètodes:
Pattern-based: Es basa en mirar patrons en la seqüència.
Covariance model: Es basa en selenoproteïnes conegudes de les quals se sap que contenen elements hèlix, a partir d’aquestes fan una predicció en altres proteïnes. Un servidor de predicció basat en covariància és Seblastian.
A partir de la predicció de novo de seqüències codificants també les podem identificar, mirant la probabilitat que sigui codificant a partir de l’estructura gènica: codó d’inici, seqüències donadores i acceptores d’splicing, codó STOP, i element SECIS en la regió 3’UTR.
Per últim, podem identificar-les amb la predicció de seqüències codificants homology-based, que consisteix en aprofitar el fet que els gens estan conservats entre espècies: si tenim una seqüència codificant per selenoproteïnes en humà, és probable que també la tinguin els ratolins. La manera més senzilla per fer això és a partir d’un mapping genètic i una predicció amb les de les selenoproteïnes que es troben en altres espècies.
Per tal d'identificar les selenoproteïnes de Atractosteus spatula hem utilitzat la predicció de seqüències basada en homologia a partir del genoma de Danio rerio i posteriorment hem fet una predicció de SECIS.
El principal objectiu d’aquest estudi era predir les proteïnes que contenien l’aminoàcid Sec, les proteïnes necessàries per a la seva síntesi i les encarregades de la incorporació de Sec en Atractosteus spatula. Per a poder aconseguir el nostre objectiu, hem dut a terme la comparació de totes les 47 proteïnes de Zebrafish trobades a SelenoDB 2.0 (que contenen o estan relacionades amb Sec). De les 47 proteïnes, 16 eren homologues (que contenien Cys) o bé eren proteïnes relacionades (sense contenir Sec); mentres les 31 restants contenien l’aminoàcid Sec. Hem decidit escollir el Zebrafish com a organisme de referència perquè era l’organisme filogenèticament més proper al nostre organisme. Hem de tenir en compte que els peixos ossis van patir una duplicació del genoma sencer (coneguda com a Ts3R).
Deiodinasa d’hormones tiroidees (DIO): En mamífers en trobem 3 (DI1, DI2 i DI3) i estan involucrades en la deionidació reductiva. Són essencials per a la regulació de l’activitat de les hormones tiroidees. No són específiques d’eucariotes, sinó que també es troben en altres regnes com en els bacteris. Aquestes proteïnes tenen un “thioredoxin fold”, un plegament característic en enzims que catalitzen la formació i isomerització dels ponts disulfurs. D. rerio presenta 4 proteïnes DIO, que són: DIO1, DIO2, DIO3a i DIO3b. DIO1 es troba present en les membranes cel·lulars i és l’encarregada de la metabolització de la hormona T4 a T3 en teixits perifèrics, cosa que també pot produir DIO2 (que es localitza en el reticle endoplasmàtic). DIO3 només pot metabolitzar T4 a rT3 (reverse triiodothyronine), que no és biològicament activa.
Glutatió peroxidasa (GPx): Aquestes proteïnes juguen un rol en moltes funcions fisiològiques com ara la senyalització del peròxid d’hidrogen (H2O2), detoxificació de hidroperoxidases i el mantiment de l’homeòstasi redox cel·lular. En mamífers en trobem 8 i de totes aquestes, 5 (GPx1, GPx2, GPx3, GPx4 i GPx6) contenen un residu Sec en el seu centre actiu. En les altres 3 (GPx5, GPx7 i GPx8), aquesta Sec ha estat substituida per una Cys. D. rerio conté 9 proteïnes GPx: GPx1a, GPx1b, GPx2, GPx3a, GPx3b, GPx4a, GPx4b, GPx7 i GPx8. De totes aquestes, el Sec del centre actiu s’ha substituït per una Cys en GPx7 i GPx8.
Tioredoxina reductasa (TRs): Són oxidoreductases que, juntament amb la tioredoxina (Trx), composen el principal sistema de reducció de disulfurs de la cèl·lula. En aquest cas, totes les proteïnes tant en mamífers com en Zebrafish contenen Sec. Juguen un paper molt important en el manteniment de l'homeòstasi redox mitjançant la reducció de les tioredoxines. En Zebrafish trobem només 2 d’aquestes proteïnes, mentres que en la resta de vertebrats en trobem 3.
Methionine sulfoxide reductases A (MsrA): Aquesta proteïna es troba molt conservada, la seva funció principal consisteix en catalitzar la reducció enzimàtica de l’aminoàcid metionina-S-sulfòxid mitjançant la tioredoxina.
Selenoproteïna R (família MSRB): Aquesta selenoproteïna, que conté zinc, també catalitza la reducció enzimàtica de l’aminoàcid metionina-R-sulfòxid mitjançant la tioredoxina. Malgrat això, aquesta família és estructuralment diferent i no presenta similaritats en les seqüències amb la família anterior (MrsA), només comparteixen la funció i el nom. MSRB1 és la principal proteïna d’aquesta família en mamífers i es localitza al citosol i al nucli. Trobem també dues proteïnes homòlogues (MSRB2 i MSRB3), amb una eficiència catalítica similar, però uns nivells d’expressió inferiors. MSRB2 es troba als mitocondris, mentres MSRB3 es troba al reticle endoplasmàtic. En Zebrafish trobem MSRB1a, MSRB1b, MSRB2 i MSRB3.
15-kDa selenoprotein (Sel15): S’anomena així pel seu pes molecular. En mamífers s’anomena Sep15. Aquesta forma un complex juntament amb una xaperona resident del reticle endoplasmàtic (UGGT), que és essencial per a la regulació del cicle de la calnexina. Aquest cicle és essencial per al correcte plegament d’algunes glicoproteïnes en el RE i la seva expressió s’indueix per la presència de proteïnes mal plegades al RE.
Selenoproteïna E (SELENOE): Aquesta proteïna només s’ha descrit en peixos. Probablement, el gen que codifica per aquesta proteïna va sorgir degut a una duplicació genètica, però la seva funció encara no es coneix.
Selenoproteïna H (SELENOH): És una selenoproteïna relacionada amb l’estrès. En mamífers, s’ha vist que protegeix a les neurones del dany UVB, promou la biogenesis de les mitocòndries i suprimeix la senescència cel·lular.
Selenoproteïna I (SELENOI): Aquesta és una de les selenoproteïnes que ha evolucionat de forma més recent i només es troba en vertebrats. És una proteïna transmembrana que s’encarrega de produir fosfatidiletanolamines, necessari per a la formació i el manteniment de les membranes vesiculars. També es troba involucrada en el metabolisme lipídic i en el plegament proteic.
Selenoproteïna J (SELENOJ): Aquesta també és una de les selenoproteïnes específiques de vertebrats i només es troba en peixos actinopterigis, i els eriçons de mar. Pot tenir un rol en la embriogènesi, expressant-se en els ulls. En Zebrafish se n’han descrit dues: SELENOJ1 i SELENOJ2.
Selenoproteïna K (SELENOK): Relacionada amb SelS. És una proteïna transmembrana localitzada al reticle endoplasmàtic. Es troba involucrada en mecanismes de ERAD, degradació de proteïnes associada al reticle endoplasmàtic. Funciona en proteïnes que es troben glicosilades i que es troben mal plegades. Així, protegeix les cèl·lules contra l’apoptosi deguda a estrès del reticle.
Selenoproteïna L (SELENOL): Pertany a la mateixa superfamília que les tioredoxines. Només es troba en organismes acuàtics, per tant en el cas dels vertebrats només es troba en els peixos. El motiu UxxU substitueix el motiu catalític CxxC de les tioredoxines, però la seva estructura és similar cosa que suggereix una funció redox. SELENOL conté dos residus Sec.
Selenoproteïna M (SELENOM): És un homòleg distant de Sel15. Es troba present en vertebrats, però està altament conservada en mamífers. La seva funció segueix sent desconeguda, però pot estar involucrada en mantenir l'homeòstasi redox.
Selenoproteïna N (SELENON): Glicoproteïna que es localitza al reticle endoplasmàtic. Participa en la regulació de l’homeòstasi del Calci i en la protecció cel·lular davant l'estrès oxidatiu. Presenta un paper important en la organització dels músculs, en el desenvolupament primerenc de Zebrafish.
Selenoproteïna O (SELENOO): Aquesta proteïna es troba localitzada en els mitocondris i és la selenoproteïna més llarga en mamífers. En D. rerio trobem dos homòlegs, SELENOO1 i SELENOO2.
Selenoproteïna P (SELENOP): Presenta una elevada expressió i secreció al plasma (ella sola constitueix el 50% de Sec total al plasma). Zebrafish presenta dos homòlegs, SELENOP1 i SELENOP2. És única perquè conté 17 residus Sec, i és un antioxidant extracel·lular.
Selenoproteïna S (SELENOS): Aquesta és una proteïna transmembrana que es troba localitzada al reticle endoplasmàtic. Es troba involucrada en la degradació de proteïnes mal plegades i es pensa que juga un paper en el control de la inflamació en eucariotes superiors.
Selenoproteïna T (SELENOT): També localitzen al reticle endoplasmàtic. Pertanyen a la familia Rdx juntament amb SelW, SelH i SelV. Com el seu nom indica, presenta un motiu CxxU conservat. En Zebrafish trobem tres homòlegs: SELENOT1a, SELENOT1b, SELENOT2.
Selenoproteïna U (SELENOU): Conté un motiu UxxC on els seus homòlegs tenen CxxU. En Zebrafish se n’han descrit 3: SELENOU1a, SELENOU1b i SELENOU3.
Selenoproteïna W (SELENOW): En Zebrafish, es troba expressada en els òrgans sensorials.
Per a poder incorporar el seleni com a Sec en una selenoproteïna, es necessita un mecanisme complicat per a decodificar el codó UGA (que generalment correspon a un STOP codon) en el mRNA. Per tant, és necessari el reconeixement del codó UGA com a codificant i que la Sec s’introdueixi a la seqüència proteica mitjançant un tRNA-Sec. Primer de tot, una serina s’ajunta amb el tRNASec mitjançant una seril-tRNA sintetasa, coneguda també com a SerRS. Posteriorment, la PSTK (una quinasa), fosforila la Ser-tRNASec. El següent enzim involucrat en l’obtenció del producte final és la Sec Sintasa (SecS), i aquest procés requereix un gast energètic mitjançant ATP. Per a que actuï el tRNASec, necessitem la presència d’una selenocysteine insertion sequence (SECIS). Aquest element es localitza en la regió 3’UTR en eucariotes, però en bacteris generalment es troben a prop del codó UGA. Aquesta seqüència conté 60 nucleòtids que adopta una estructura secundària en forma de stem-loop. Així, aquesta estructura actua com a un element en cis que reconeix factors en posicions trans i els dirigeix cap als ribosomes.
En eucariotes, necessitem almenys dos factors en trans per a interpretar que el codó UGA codifica per a una Sec, i són el SECIS binding protein 2 (SBP2) i el Sec-specific elongation factor (eEFSec). SBP2 es troba associat als ribosomes i conté un domini d’unió al RNA que interacciona amb aquests elements SECIS amb una elevada afinitat i especificitat. Per altra banda, també presenta un domini d’unió amb eEFSec, que s’encarrega de reclutar el tRNA i facilita la incorporació de la Sec en la seqüència del pèptid.
Finalment, és important parlar d’una altra proteïna coneguda com a SECp43 (tRNA Sec 1 associated protein 1), que juga un paper important en la síntesis de les selenoproteïnes mitjançant un complexe multiproteic. Cal mencionar que ninguna d’aquestes proteïnes contenen una Sec en la seva seqüència d’aminoàcids. Totes són essencials per al correcte funcionament fisiològic de les cèl·lules, ja que mutacions en aquestes porten a patologia. En el seu genoma, Danio rerio conté 2 homòlegs per a SBP2 i 2 altres per a SECp43.
Per més informació, consultar Wikipedia
Obtenció del genoma d’Atractosteus spatula: Aquest genoma ens ha estat facilitat pels professors de l'assignatura accedint a /mnt/NFS_UPF/soft/genomes/2021/Atractosteus_spatula/genome.fa
Obtenció de les seqüències de selenoproteïnes d’una espècie propera:
Primer de tot, hem de determinar quina és l’espècie més propera (o prou propera) per a obtenir uns bons resultats. En aquest cas l’espècie d’interès és Atractosteus spatula, un peix. Per tant, hem considerat que l’organisme amb el qual té més sentit comparar-lo és el Zebrafish (Danio rerio).
Per a obtenir les seqüències relacionades amb les selenocisteïnes de l’organisme que ens interessa, hem utilitzat la base de dades SelenoDB 2.0. Copiem totes les seqüències en un arxiu multifasta que hem anomenat selzebra.multifasta. Les proteïnes que hem obtingut de la base de dades són: Sel15, eEFsec, SELENOE, GPx1a, GPx1b, GPx2, GPx3a, GPx3b, GPx4a, GPx4b, GPx7, GPx8, DIO1, DIO2, DIO3a, DIO3b, MsrA, PSTK, SBP2, SecS, SEPHS, SEPHS2, SELENOH, SELENOI, SELENOJ1, SELENOK, SELENOL, SELENOM, SELENON, SELENOO1, SELENOO2, SELENOP, SELENOP2, MSRB1a, MSRB1b, MSRB2, MSRB3, SELENOS, SELENOT1, SELENOT1b, SELENOT2, SELENOU1a, SELENOU2, SELENOU3, SELENOW, TXNRD2, TXNRD3, SECp43a, SECp43b.
Abans de començar amb l’anàlisi, s’ha de dur a terme una modificació del fitxer multifasta, que en aquest cas l’hem feta manual. Totes les selenocisteïnes (U) que detectem en les seqüències s’han de substituir per “X” per tal de poder realitzar la predicció. En descarregar-nos les seqüències proteiques de la base de dades SelenoDB 2.0, pot ser que apareguin entre les seqüències alguns caràcters especials (@, #, *, per exemple). Els programes utilitzats no detecten aquests caràcters, de manera que s’han d’eliminar si es troben al final de la seqüència o substituir per una “X” si estan intercalats amb els aminoàcids. Amb aquest fitxer multifasta modificat, ja podem començar a realitzar l’anàlisi.
tBLASTn: Aquest programa utilitza un algorisme per a comparar una seqüència problema (query) contra una gran quantitat de seqüències que es troben en una base de dades. En aquest cas, hem utilitzat un tipus de BLAST anomenat tBLASTn, on la query és la seqüència d'aminoàcids de la selenoproteïna que coneixem en el genoma de Danio rerio i la comparem contra una seqüència de nucleòtids traduïda en tots els possibles marcs de lectura (el genoma problema Atractosteus spatula). tBLASTn permet comprovar si les diferents selenoproteïnes de Danio rerio també es troben al genoma d’Atractosteus spatula.
Un cop hem fet aquest pas obtenim una taula que conté: el contig en el qual es produeix el hit, el percentatge de similaritat, la posició de start del hit, la posició end i el e-value, que és un indicador de la significació del hit. Com més petit sigui aquest valor, més significatiu és l'alineament que en resulta. S’ha determinat que, per a seleccionar un scaffold, e-value ha de ser menor que 10^-5.
Fastafetch: Un cop seleccionat els millors hits, volem seleccionar on es troba el hit del nostre scaffold per no haver de treballar amb tot el genoma i ser més eficients. Fastafetch ens permet emmagatzemar la seqüència del scaffold del hit (o hits) escollits en un arxiu anomenat contig.fetch.fa. Destacar que se’n genera un per cada scaffold analitzat i es registra a la carpeta en qüestió.
Fastasubseq Amb fastasubseq acotarem la regió del scaffold on es troba el hit, és a dir, guardarem en un arxiu la regió d’interès on es troba la selenoproteïna. Primer de tot, el programa agafa aquests dos valors, i interpreta si la cadena és forward o reverse. A partir de la posició de la seqüència, obtinguda a través del BLAST, augmentarem la llargada del nostre hit 100.000 nucleòtids downstream i 100.000 upstream per tal d'assegurar-nos que la nostra selenoproteïna es troba en aquesta regió. Obtenim un arxiu per cada scaffold anomenat genomic.fa que guardarà la seqüència del scaffold.
Exonerate: Aquest programa ens permet obtenir un alineament acurat i predir l'estructura exònica de la seqüència problema. Ens permet assegurar que el nostre hit es troba dins l'exó i que, per tant, codifica per una proteïna. El que estem fent és l’alineament del fragment de DNA que hipotitzem és una selenoproteïna i hem extret amb fastasubseq amb la seqüència de DNA de la proteïna inicial. Ara obtenim el nostre arxiu .gff, en el qual li assignem el nom de la seva proteïna. En aquest trobarem una representació de l’alineament entre el query i la regió que hem extret amb el fastasubseq. El que farem serà obtenir un fitxer en format .gff perquè ens doni les seqüències que es troben incloses en els exons.
FastaseqfromGFF: Seguidament, el que volem és crear un arxiu .fasta amb la seqüència de cDNA. Per tal d'aconseguir això, utilitzarem un programa Perl anomenat fastaseqfromGFF.pl Aquest programa ens dona la seqüència del cDNA dels exons predits en el pas anterior, i emmagatzemem aquests valors en un arxiu que acaba en pred.nuc.
Fastatranslate: El programa ara transformarà la seqüència de nucleòtids a aminoàcids. Obtenim un arxiu que acaba en _pred.fa.
t_coffee: Utilitzem aquest programa per a fer l’alineament de les nostres seqüències d’aminoàcids predites (l’arxiu pred.fa) amb la seqüència en format fasta de la query.
Per tal de guanyar eficiència, s’ha automatitzat el procés d’anàlisi executant els programes descrits anteriorment en un mateix codi en llenguatge python. D’aquesta manera, amb un sol algoritme podem analitzar cada scaffold d’interès. Per consultar el script del programa feu click aquí.
Un cop tenim l’alineació entre la selenoproteina de Zebrafish de la base de dades i la predita del genoma d’interès, s’ha de fer la predicció dels elements SECIS, característics de les selenoproteïnes i que es troben a l’extrem 3’ de la regió codificant d’aquestes. Per realitzar-ho, s’ha utilitzat el SECISSearch 2.0.
Nota: les proteïnes sombrejades en blau corresponen a la maquinària de les selenoproteïnes.
Per la realització del projecte s’han emprat els processos bioinformàtics mencionats anteriorment, obtenint una sèrie de dades representades a la taula de resultats. Durant l’anàlisi de dades, han sorgit un seguit d’inconvenients que cal destacar a continuació.
El procés d’automatització al principi era molt bàsic, però a mesura que avancem en el procés d’obtenció dels resultats és molt més complex. De fet, al començament realitzàvem un canvi manual dels caràcters que no ens interessen (com canviar les U per X). Un cop passada aquesta barrera, tota la resta ja es podia fer amb el nostre script. Per exemple, podem detectar si el nostre hit es troba en la cadena forward o reverse, i depenent d’això duem a terme una acció o una altra.
També cal comentar que el script ens guardava la informació necessària per a poder calcular les posicions dels exons en els scaffolds, a partir de les quals es pot obtenir la posició dels gens de les selenoproteïnes al genoma. La idea original era realitzar un mapa visual dels gens que contenen o estan relacionats amb les selenoproteïnes, però al final no s’ha pogut dur a terme.
Un altre dels principals aspectes a tenir en compte en el nostre projecte, és que el nostre organisme d’interès Atractosteus spatula es tracta d’un peix ossi. Com s’ha explicat abans, aquests organismes van patir una duplicació del genoma sencer (Ts3R). Això ha afectat al screening, ja que a part de la presència d’un gran nombre de selenoproteïnes del nostre organisme de referència, Danio rerio, també s’havia de tenir en compte totes les scaffolds de l’organisme d’interès. Com que hi ha hagut tantes duplicacions, els hits d’unes proteïnes s’alineaven amb les seves proteïnes paràlogues, provocant una gran quantitat d’informació que va provocar “soroll” a les dades realment importants.
Arran d’aquestes duplicacions que presenten les espècies de peixos ossis, vam pensar que la forma més factible d’assignar de forma correcta cada scaffold amb cada selenoproteïna era a partir de la realització d’arbres filogenètics, sobretot de les famílies de selenoproteïnes més extenses, com les GPx. Un cop realitzats els arbres, vam comprovar les associacions que s’havien establert i vam percebre algun error en l’alineament. Per tal de solucionar-ho, vam acabar elaborant l'anàlisi de forma manual, comparant cada scaffold amb la seqüència proteica i veure el nivell de concordança entre ambdues seqüències.
A continuació, per redactar la discussió de forma detallada, aquesta ha estat dividida en apartats segons la proteïna d’interès estudiada:
Iodothyronine deiodinase
En el nostre genoma de referència, en Danio rerio (Zebrafish), hi ha un total de 4 proteïnes; SPP00000611_2_0_Di1, SPP00000612_2_0_Di, SPP00000613_2_0_Di i SPP00000614_2_0_Di. Quan ho comparem amb el nostre organisme d’interès, Atractosteus spatula, observem que només hi ha un total de 3 scaffolds que actuïn com a hits per aquestes proteïnes. Aquests són JAAWVO010011080.1, JAAWVO010016830.1 i JAAWVO010044538.1. Quan observem els t-coffee veiem que les interaccions són les representades a les taules. Concretament, volem destacar que tant la proteïna SPP00000612_2_0_Di, com la SPP00000613_2_0_Di de l’organisme de referència s’alineen amb la scaffold JAAWVO010016830.1.
Per tant, podem hipotetitzar que quan l’organisme de referència i el nostre organisme es van separar, només hi havia 3 proteïnes. Tanmateix, hi ha hagut una pèrdua d’una proteïna en el nostre organisme d’interès, ja sigui perquè l’organisme de referència ha patit una duplicació funcional d’una de les proteïnes (la 613 de la 612 o viceversa) o perquè hi ha hagut una pèrdua de la seva respectiva scaffold. Considerem que és funcional, ja que presenta tots els exons i mida dels altres gens i manté la selenocisteïna.
15 kDa selenoprotein-like protein (Fep15)
En aquesta proteïna, es manté la selenocisteïna de l’organisme de referència i la major part de la proteïna. L’única regió modificada és una petita deleció a la regió central de la proteïna. Tanmateix, sembla no afectar a l’estructura tridimensional de la proteïna, ja que la predicció del seu SECIS (SEleno Cysteine Insertion Sequences) segueix predient una estructura en el nostre organisme.
Methionine sulfoxide reductase A (MsrA)
Tant en l’organisme de referència com en el nostre organisme només trobem 2 proteïnes. Per tant, podem determinar que la duplicació es va fer en l’organisme de referència abans que ambdues espècies se separessin (ancestre comú). Aquesta situació és força interessant, ja que en ambdós casos en el nostre organisme tenim la proteïna incompleta amb delecions a la regió inicial de la proteïna. A més a més, tampoc presenten estructura SECIS predita. Per tant, hom podria considerar que el nostre organisme d’interès, l’Atractosteus spatula, ha perdut aquestes proteïnes. És un cas estrany, ja que com hem mencionat a la introducció el gen MsrA està altament conservat, per tant, caldria més informació per confirmar la seva disfuncionalitat.
Selenoprotein R (MsrB family)
La Selenoproteina R, també anomenada Methionine-R-sulfoxide reductase 1 (MSRB1), forma part de la família de les MsrB i, com hem mencionat, té una gran proximitat amb la família anterior, ja que encara que no provinguin de la mateixa seqüència ni siguin similars, tenen funcions complementàries. Cal destacar, que en aquest cas ha succeït el cas contrari. Mentre que en Danio rerio hi ha un total de 4 proteïnes, hem trobat un total de 9 scaffolds. Quan hem fet una inspecció manual de cadascun d’ells, hem trobat que 3 dels scaffolds JAAWVO010014277.1, JAAWVO010067529.1 i JAAWVO010055741.1 són duplicacions parcials de la regió inicial i es poden considerar com a proteïnes incompletes. Tanmateix, les altres 6 scaffolds sí que presenten tota l’estructura de la proteïna, i en 5 d’elles es pot predir l’estructura SECIS. Tenint en compte els T-coffees, podríem hipotetitzar que l’organisme original presentava 4 proteïnes en el moment en el qual el nostre organisme de referència i d’interès es van separar. És més, com es pot observar a la taula es pot determinar quina proteïna correspon a cada scaffold.
El que encara és més interessant, és el cas de la proteína SPP00000645_2.0_SelR1 amb la qual s’uneixen tres scaffolds JAAWVO010037148.1, JAAWVO010011014.1 i JAAWVO010078268.1. És destacable i podem considerar que totes provenen de la mateixa proteïna perquè les 3 scaffolds presenten un guany d’una selenocisteïna. Això és determinant, perquè ens indica que la duplicació de la selenoproteïna es va realitzar posteriorment a l’aparició de la selenocisteïna (seria molt difícil que les tres seqüències haguessin portat a una duplicació en el mateix punt). Les altres tres proteïnes s’uneixen cadascuna a una scaffold en concret. Cal destacar, que hi ha una major similitud entre la SelR i la SelR1 i també entre la SelR2 i la SelR3, la qual cosa podria estar relacionada amb el seu origen, aquesta relació encara queda més recolzada per la presència de selenocisteines en la SelR i SelR1, però presència de cisteïnes en la SelR2 i SelR3. Cal destacar que l’ordre de la triple separació es desconeix, per tant, qualsevol de les 3 haver estat l’original.
Per tant, potser el fet que les MsrA en Atractosteus spatula puguin haver deixat de fer funcionals, podria estar relacionat amb aquest guany de proteïnes de MsrB. En realitzar funcions complementàries, potser una adaptació va portar a les MsrB a prendre la funció principal com a reductors enzimàtics catalítics de la Methionine-R-sulfoxide. Tanmateix, això no explicaria la reducció de la methionine-S-sulfoxide (MsrA). Més estudis sobre si les porteïnes MsrA han deixat de ser funcionals en Atractosteus spatula i en si les proteínes MsrB són capaç de reduir la methionine-S-sulfoxide podria donar-nos més informació sobre aquest fenomen que observem en el nostre organisme.
15-kDa selenoprotein (Sel15)
La Sel15 s’ha conservat molt entre l’organisme de referència i el de la nostra espècie, destacant com a únic canvi la pèrdua d’alguns aminoàcids en la part inicial de la proteïna. És interessant recalcar que aquesta proteïna comparteix el mateix scaffold amb la DI1, indicant la proximitat d’ambdues proteïnes en el genoma de l’organisme d’interès. Presenta també un SECIS i 4 exons ben determinats. Tota aquesta informació apunta al bon funcionament d’aquesta proteïna així com a la seva importància en l’organisme per la necessitat de ser preservada.
Hi ha una bona conservació de tota la proteïna, també presenta la selenocisteïna, igual que en l’organisme de referència i podem observar una clara predicció amb SECIS. No hi ha cap factor que sembli que pugui afectar a la seva funció.
Selenoprotein H (SELENOH)
La regió central i terminal de la proteïna estan ben conservades entre ambdós organismes. Tanmateix, una de les principals diferències és una falta dels primers aminoàcids de la regió inicial. Entre ells, també destaca la pèrdua de la metionina (el nou aminoàcid inicial és una G). Encara que l’inici és diferent, podem observar la predicció per SECIS (vegeu taula de resultats). En tractar-se d’una sola proteïna sense duplicacions no sembla gaire factible que es tracti d’una pèrdua de funció.
Selenoprotein I (SELENOI)
Com heu pogut observar en la taula de resultats mostrada anteriorment, aquesta proteïna presenta un total de 4 duplicacions respecte a una única proteïna original en l’organisme de referència. Queda força clar que les scaffolds JAAWVO010070734.1 i JAAWVO010057697.1 són duplicacions no funcionals de la regió inicial. La part interessant es troba en les scaffolds JAAWVO010042823.1 i JAAWVO010000708.1. La JAAWVO010042823.1 es troba en la cadena positiva i presenta un total de 10 exons. En canvi, la JAAWVO010000708.1 es troba en la cadena negativa i presenta un total de 8 exons. Hom podria pensar que, en haver perdut 2 exons, es tracta d’una proteïna no funcional. Tanmateix, cal destacar que en aquest segon scaffold hi ha hagut un canvi de selenocisteïna a cisteïna. Aquest canvi podria ser degut a la pèrdua de funció mencionada anteriorment (ja que una selenocisteïna és un aminoàcid rar i no valdria la pena l’energia necessària per al seu funcionament). Però també podria implicar un canvi de funció en aquesta nova proteïna. A més a més, com hi ha hagut aquest canvi d’aminoàcid podem determinar amb força seguretat que la scaffold JAAWVO010000708.1 és una duplicació de la JAAWVO010042823.1, perquè si no implicaria un doble canvi d’aminoàcids.
Selenoprotein J (SELENOJ)
Només observem una seqüència en ambdós organismes. Tot i la seva longitud, en un total de 8 exons, destaca per mantenir una bona conservació respecte de l’organisme de referència. El principal canvi és la deleció de la regió terminal, la qual no sembla afectar ni a la predicció de la SECIS, ni a la presència d’una selenocisteina ni, per tant, en la seva funció.
Selenoprotein L (SELENOL)
La selenoproteïna L presenta una pèrdua important de la regió inicial de la proteïna. Resulta força estrany que tant el Seblastian com la predicció de SECIS determinin que és una selenoproteïna. Encara que presenti unes bones prediccions no estem segures de com és de probable que mantingui la seva funció, en tot cas, potser aquesta pèrdua pot haver provocat un canvi de funció, d’eficàcia o de target.
Selenoprotein M (SELENOM)
En aquest cas és remarcable la manca d’una metionina inicial tant en l’espècie de referència com en Atractosteus spatula. També hi ha una deleció inicial de 6 aminoàcids. La predicció de SECIS és bona i sembla que la selenocisteïna s’ha mantingut entre ambdues espècies. Podem confirmar que es tracta d’una selenoproteïna funcional.
Selenoprotein N (SELENON)
La selenoproteïna N presenta 2 possibles scaffolds. A la taula només s’ha marcat la JAAWVO010054850.1, ja que l’altra scaffold es tracta només d’una duplicació de la regió terminal de la proteïna. Pel que fa a la scaffold d’interès, cal remarcar que hi ha una pèrdua de la regió inicial, incloent-hi la metionina inicial que passa a ser substituïda per una fenilalanina. Un altre punt que cal remarcar, és que en el nostre estudi hi apareix una segona selenocisteïna. Per poder tenir altres referències hem anat al genoma humà, ja que és el més conegut, i hem comparat la nostra Selenoprotein N. Els resultats han confirmat que en el genoma humà també presenta dues selenocisteïnes. Per tant, podem suposar que sí que ha patit el guany d’una selenocisteïna i es tracta d’una selenoproteïna funcional.
Selenoprotein O (SELENOO)
Mentre que l’organisme de referència té dues selenoproteïnes, el nostre organisme d’interès presenta una única scaffold. Cal destacar que en ambdós casos l’alineament és molt acurat. En l’alineament amb SPP00000641_2.0_SelO veiem una pèrdua en la regió central de la proteïna, mentre que en l’alineament amb SPP00000640_2.0_SelO presenta una pèrdua en la regió inicial de la proteïna. En ambdós casos es fa la predicció del SECIS i és positiva. Per tant, podem determinar que, en primer lloc, es va separar l’ancestre comú d’ambdues espècies quan només hi havia una selenoproteïna i, un cop separats, va patir una duplicació en Danio rerio. També es podria haver donat una pèrdua de la proteïna a Atractosteus spatula.
Selenoprotein S (SELENOS)
La selenoproteina S està clarament truncada en la regió inicial. Les prediccions SECIS segueixen considerant l’estructura de la proteïna funcional per a generar la selenoproteïna. S’haurien de fer més proves per comprovar si hi ha hagut una pèrdua de funció. Tanmateix, en tractar-se d’una proteïna única no ho creiem probable tot i la manca de la regió inicial.
Selenoproteins T (SELENOT)
Les selenoproteines T presenten, en l’organisme de referència, un total de 3 proteïnes. Tanmateix, en el nostre organisme d’interès només hi ha dues scaffold completes, i una scaffold que només presenta la regió terminal de la SPP00000650_2.0_SelT. Llavors, tenint en compte que en l’organisme de referència SPP00000649_2.0_SelT i SPP00000651_2.0_SelT s’alineen amb JAAWVO010058105.1 i que la scaffold JAAWVO010009937.1 s’alinea amb SPP00000649_2.0_SelT, podem hipotetitzar que després de la primera duplicació en l’ancestre comú es van separar, amb la presència de dues proteïnes en cada organisme. Després., en l’organisme de referència hi va haver una segona duplicació, a partir de la proteïna que s’unís a la scaffold JAAWVO010058105.1. Per últim, en l’organisme d’interès hi va haver una duplicació incompleta de la regió terminal de la scaffold JAAWVO010009937.1. Podem saber que es duplica d’aquesta perquè presenta alineament amb el seu homòleg. Per tant, podem determinar que en el nostre organisme d’interès hi ha dues selenoproteïnes funcionals.
Glutathione peroxidases (GPx)
La família de les GPx és la família de selenoproteïnes més extensa, per tant, el procés d’hipotetitzar els seus gens homòlegs i paràlegs és complex i llarg. Durant la realització del treball es van provar de fer arbres filogenètics, amb aplicacions com phylogeny.fr. Tanmateix, comparant els resultats obtinguts en l’arbre amb els resultats obtinguts al t-coffee veiem que hi havia errors i alineaments que no eren correctes. Per tant, vam decidir fer nosaltres el nostre propi sistema per separar-los. La majoria de gens homòlegs han estat determinats, tanmateix, en un cas ens hem trobat amb una situació que no tenim clar com s’ha originat evolutivament. Per tant, hem determinat una hipòtesi que comentarem a continuació. En primer lloc, totes les proteïnes que contenien selenocisteïnes en l’organisme de referència en tenen també en el nostre organisme d’interès. Passa el mateix en el cas dels gens amb cisteïnes, es mantenen conservades en ambdós genomes. Com s’observa a la taula, es veu que ja l’organisme de referència presentava una gran quantitat de gens ortòlegs que es van obtenir per duplicació de genoma. Hi ha dos casos en concret on la duplicació es va dur a terme després de la separació de l’ancestre comú. Per tant, no sabem quina de les dues és l’original. Per altra banda, el problema principal es troba entre SPP00000619_2.0_GPx7 i SPP00000620_2.0_GPx8, ja que ambdues presenten alineament amb la scaffold JAAWVO010027071.1. Tanmateix, una de les proteïnes inicials ja té un homòleg específic (SPP00000620_2.0_GPx8). Per tant, suposaríem que la proteïna homòloga seria SPP00000619_2.0_GPx7, no obstant això, aquesta proteïna presenta un exó menys, la qual cosa implica que no podria haver sorgit d’aquesta (perquè hauria aparegut un exó idèntic al de l’altra proteïna de referència la qual cosa és impossible). Per tant, hem acabat considerant que l’escenari més probable és que es tracti d’una duplicació en la qual és el gen de referència el que ha patit una deleció en la regió central del gen. Això explicaria per què hi ha similitud amb els 3 exons de la proteïna SPP00000620_2.0_GPx8, però seria en realitat la SPP00000619_2.0_GPx7.
Selenoproteins U (SELENOU)
La família de Selenoproteines U presenta una situació molt similar a la de les iodotironina deiodinases. Ens trobem davant de 4 proteïnes en l’organisme de referència i 3 scaffolds en l’organisme d’interès. En aquest cas com sabem que la scaffold JAAWVO010057056.1 s’alinea amb SPP00000654_2.0_SelU i SPP00000652_2.0_SelU podem determinar que, en primer lloc, hi va haver una separació de l’ancestre comú quan només hi havia 3 proteïnes i, més tard, el Danio rerio va patir una duplicació de la SelU, l’altra opció seria que hi hagués hagut una pèrdua de la selenoproteïna en algun moment en l’organisme de referència. Tant SPP00000656_2.0_SelU3 com SPP00000655_2.0_SelU2 presenten una cisteïna, mentre que les dues SelU (SPP00000654_2.0_SelU i SPP00000652_2.0_SelU) són selenoproteïnes.
Selenoproteins W (SELENOW)
En aquest cas, les proteïnes de Danio rerio SPP00000659_2.0_SelW, SPP00000657_2.0_SelW i SPP00000658_2.0_SelW estan alineades amb JAAWVO010009874.1 i JAAWVO010004104.1. Com que només hi ha dues scaffolds podem dir que hi ha una pèrdua en el nostre organisme d’interès. Aquesta pèrdua podria ser per una duplicació posterior a separació dels organismes d’interès o una pèrdua un cop aquests ja s’havien separat. Cal destacar, que les tres proteïnes de l’organisme de referència tenen selenocisteïnes, tanmateix, en el scaffold JAAWVO010009874.1 hi ha una deleció en la regió inicial que porta a l’eliminació de la selenocisteïna. Per tant, podem considerar que hem perdut una de les selenoproteïnes en el nostre organisme d’interès.
Thioredoxin reductases (TR)
Hi ha certes contradiccions en aquesta selenoproteïna. Es tracta d’una de les selenoproteïnes més ben conservada. Tant en l’organisme de referència com en el d’interès contenen dues proteïnes. Tanmateix, quan es fa la predicció amb SECIS i amb Seblastian una d’elles, SPP00000661_2.0_TR3, apareix com a proteïna sense cap mena de selenoproteïna. Es va tornar a realitzar el procés per comprovar que no fos un error humà (escollint la proteïna) o de l’ordinador (en el moment de la predicció) però els resultats van ser negatius. Considerant que la proteïna és tan similar a la de l’organisme de referència, encara que Seblastian no pugui predir la selenoproteïna no podem descartar-la com a possible positiu. Més estudis més detallats s’haurien de fer per comprovar la situació real d’aquest cas.
Maquinària de les Selenoproteïnes
La maquinària també juga un paper fonamental en la formació de selenoproteïnes i en la correcta traducció del codó UGA (Stop) a selenocisteïna. Com podem observar a la taula de resultats comptem amb totes les proteïnes les quals tenen un paper en la maquinària de les selenoproteïnes. Aquestes no són selenoproteïnes, per tant, té sentit que quan es realitzen proves de fer una predicció de Seblastian i SECIS el valor obtingut sigui negatiu. Tanmateix, cal destacar que la proteïna SECIS Binding Protein 2 (SBP2) presenta en el scaffold JAAWVO010060449.1 una molt bona predicció de SECIS, amb presència d’una selenocisteïna tant en l’organisme d’interès com en el de referència.
Els recursos obtinguts durant les pràctiques de l’assignatura de Bioinformàtica, destacant l’aprenentatge de tot el procés d’automatització, ha permès la realització del projecte de la forma més senzilla, mecanitzada i homogènia, evitant el treball manual d’obtenció de dades per cada proteïna. Les eines de predicció com Seblastian o SECIS són un clar indicador de la proteïna esperada i serveixen per reforçar i donar robustesa a suposicions i hipòtesis plantejades. Per fer els resultats més sòlids, es podrien haver comparat les proteïnes predites de l’organisme d’interès (Atractosteus spatula) amb altres organismes de referència, a banda del Zebrafish.
Un dels principals inconvenients ha estat l’estudi filogenètic, el qual no va funcionar correctament i es va haver de realitzar cada comparació manualment. Aquest hàndicap ens ha servit per aprofundir i comprendre de forma més clara conceptes com gens ortòlegs/paràlegs i les principals formes de modificacions presents en les nostres proteïnes, com serien les duplicacions, delecions, pèrdues o regions incompletes.
Tot això ha portat a la determinació de 39 selenoproteïnes al genoma d’Atractosteus spatula, a banda de 10 proteïnes homòlogues i 9 proteïnes de la maquinària de transcripció de les selenoproteïnes. A més a més, també ens ha permès reflexionar sobre fenòmens que requereixen més estudi com serien els gens MsrA i MsrB, o la duplicació de selenocisteïnes en les MsrB. Més estudis combinant eines informàtiques amb coneixements biològics podran permetre un millor coneixement de les selenoproteïnes en organismes complexos com Atractosteus spatula.
Papp LV, Lu J, Holmgren A, Khanna KK. From Selenium to Selenoproteins: Synthesis, Identity, and Their Role in Hu-man Health. Antioxid Redox Signal. 2007; 9 (7): 775–806.
Lorin T, Brunet FG, Laudet V, Volff JN. Teleost fish-specific preferential retention of pigmentation gene-containing families after whole genome duplications in vertebrates. G3 - Genes Genom Genet. 2018; 8 (5): 1795-1806.
Labunskyy V, Hatfield D, Gladyshev V. Selenoproteins: Molecular Pathways and Physiological Roles. Physiol Rev. 2014; 94 (3): 739-777.
Mariotti M, Ridge P, Zhang Y, Lobanov A, Pringle T, Guigo R et al. Composition and Evolution of the Vertebrate and Mammalian Selenoproteomes. PLoS ONE. 2012;7(3):e33066.
Kryukov G, Gladyshev V. Selenium metabolism in zebrafish: multiplicity of selenoprotein genes and expression of a protein containing 17 selenocysteine residues. Genes Cells. 2000;5(12):1049-1060.
Sunde RA. Molecular Biology of Selenoproteins. Annu Rev Nutr. 1990;10(1):451–74.
Xu X, Mix H, Carlson B, Grabowski P, Gladyshev V, Berry M et al. Evidence for Direct Roles of Two Additional Factors, SECp43 and Soluble Liver Antigen, in the Selenoprotein Synthesis Machinery. J Biol Chem. 2005;280(50):41568-41575.
Tujebajeva RM, Ransom DG, Harney JW, Berry MJ. Expression and characterization of nonmammalian selenoprotein P in the zebrafish, Danio rerio. Genes Cells. 2000;5(11):897-903.
Santesmasses D, Mariotti M, Gladyshev VN. Bioinformatics of Selenoproteins. Antioxid Redox Signal. 2020;33(7):525-536.
Gladyshev VN, Arnér ES, Berry MJ, Brigelius-Flohé R, Bruford EA, Burk RF, Carlson BA, Castellano S, Chavatte L, Conrad M, Copeland PR, Diamond AM, Driscoll DM, Ferreiro A, Flohé L, Green FR, et al. Selenoprotein Gene Nomenclature. J Biol Chem. 2016;291(46):24036-24040.
Mariotti M, Lobanov AV, Guigo R, Gladyshev VN. SECISearch3 and Seblastian: new tools for prediction of SECIS elements and selenoproteins. Nucleic Acids Res. 2013;41(15):e149.
Mariotti M, Ridge PG, Zhang Y, Lobanov AV, Pringle TH, Guigo R, Hatfield DL, Gladyshev VN. Composition and evolution of the vertebrate and mammalian selenoproteomes. PLoS One. 2012;7(3):e33066.