
|
| Emiliania huxleyi | Selenoproteínas | Antecedentes de selenoproteínas en Emiliania huxleyi |
El selenio es un nutriente esencial en un gran número de especies para las cuales su clasificación abarca desde el reino Procariota al Eucariota. Mientras que la deficiencia de éste elemento en el organismo puede comprometer la salud del individuo, su exceso puede resultar tóxico. A nivel celular, la fuente predominante de selenio proviene de las selenoproteínas, proteínas que incorporan el aminoácido selenocisteína en su cadena peptídica. La selenocisteína es un aminoácido análogo a la cisteína que contiene selenio en lugar de azufre. Para que la síntesis de éste aminoácido tenga lugar es necesaria una maquinaria de síntesis específica ya que la selenocisteína es un aminoácido codificado por un solo codón (UGA) que codifica normalmente para una señal de STOP. El principal elemento implicado en descodificar de forma alternativa éste codón es el elemento SECIS, siempre presente en selenoproteínas. Los elementos SECIS vienen definidos por una secuencia característica en la región 3' del gen de la selenoproteína que otorga una estructura secundaria muy característica.
El principal problema a la hora de intentar buscar selenoproteínas en nuevos genomas reside en la diversidad existente a nivel de secuencia entre diferentes selenoproteínas y la importancia de similitud en cuanto a la estructura 3D de los elementos SECIS. Para la mayoría de programas de predicción de genes, intentar discernir qué codones UGA van a poder ser descodificados de forma alternativa resulta imposible ya que éste codón aparecerá siempre como una señal de STOP.
En éste trabajo recurriremos a métodos bioinformáticos para rastrear el genoma recientemente secuenciado del organismo procariota Emiliania Huxleyi, en busca de nuevas selenoproteínas, maquinaria de síntesis así cómo selenoproteínas que ya hayan podido ser descritas en otros organismos cuyos genomas ya han sido secuenciados y rastreados.
Emiliania huxleyi

|
 .
.
Emiliania huxleyi es una de las 5000 especies de fitoplancton que podemos encontrar en las distintas capas del océano. El fitoplancton es el equivalente oceánico a las plantes terrestres, formando las bases de los ecosistemas marinos. Estos microorganismos fotosintetizadores son de vital importancia para regular el clima del planeta.
Como hemos visto en el árbol filogenético, E. Huxleyi pertenece al orden de los Cocolíferos, los cuáles poseen cubiertas celulares de carbonata cálcico. Dentro de este orden, E. Huxleyi es la especie más abundante y globalmente extendida a excepción de los océanos polares. Cuando las condiciones del agua son favorables, es capaz de proliferar de forma masiva, a veces más de 100.000 km2 (el tamaño de Inglaterra). Durante estas proliferaciones masivas las células se acompañan de un gran número de cocolítos (cubiertas de carbonato cálcico), muchos de ellos pegados a las células pero también otros muchos flotando por separado en el agua. Esto se cree que se debe a que cuando realizan la división celular la cubierta se rompe y desprende. Este hecho es único entre los cocoliforados.
E. Huxleyi son cocolitoforados, lo que significa que producen una concha a modo de exoesqueleto que contiene placas llamadas cocolítos. Los cocolítos se forman en el interior de la célula a través de un proceso llamado cocolitogénesis. Cuando cada cocolíto esta completamente formado se transfiere al exterior celular gracias a un transporte vesicular.
El género Emiliania, como muchos otros tipos de fitoplácton, es autótrofo. Produce su propia energía a través de la fotosíntesis, por lo tanto necesita cloroplastos. Estos, son fruto de una endosimbiosis secundaria que consitió en el engullimiento de una alga que ya contenía cloroplastos de un ancestro tipo cyanobacteria.
Emiliania es un cocolífero maríno que vive en aguas frías a unas profundidades de entre 10 y 20 metros. Esto sugiere que intensidades de luz fuertes pueden ser un factor que cause la proliferación masiva de estos organismos. Estas proliferaciones se conocen como "Blooms" y pueden desencadenarse también por niveles de fosfato y hierro bajos. E. Huxleyi generalmente prefiere aguas frías y pobres en nutrientes, concretamente bajas concentraciones de CO2 ya que ayudan al proceso de calcificación.
Como todas las especies, E.Huxleyi forma parte de nichos ecológicos, son la comída principal de determinados tipos de bacteria y zoopláncton, los cuales se alimentan durante los "Blooms".

E.Huxleyi juega un papel importante en la atmósfera terreste. Los "Blooms" que forman estos cocolítos, lejos de absorber la luz, la reflejan, provocando por un lado que el agua adquiera un color turquesa que incluso se puede detectar desde un satélite y por otro lado causan que el calor de los rayos solares se reflejen hacia la atmósfera en vez de hacia el mar. Además, al fabricar sus esqueletos producen una liberación de CO2 que reduce la cantidad total de captación de CO2 atmosférico por parte del agua del mar. Por lo tanto se puede afirmar que al perturbar el ciclo de carbono en el océano provocando diferencias en la cantidad final de CO2 que puede ser almacenada en la atmósfera contribuyendo así al efecto invernadero.
La Estación Biológica de Roscoff en Bretaña (Francia) ha propuesto que el sexo surgió en el agua, resultado de la interacción entre la alga Emiliania huxleyi y el virus Eh V, un virus que infecta tal alga.
La investigación demostró que la interacción entre ambos fue precursora de la célula haploide, esencial en la reproducción sexual, puesto que el alga en presencia de virus pasa a un estado de camuflaje volviéndose haploide, como los espermatozoides o los óvulos. De este modo el alga evita la amenaza del virus.
El biólogo Colomban de Vargas, coordinador de la investigación, ha llamado a esta técnica estrategia del Gato de Cheshire, el gato de la novela "Alicia en el país de las maravillas" de Lewis Carrollquien quien desaparecía para evitar ser decapitado por la Reina de Corazones.
Tal estrategia, publicada en PNAS, es responsable del aumento de la población de la colonia, haciéndola visible incluso desde el espacio. Las algas camufladas, haploides, como si de óvulos o espermatozoides se trataran, pueden fusionarse para dar origen a nuevas algas, células diploides.
Selenoproteínas
El selenio es un micronutriente de vital importancia tanto para los hombres como para los animales. El rol del selenio ha sido atribuido básicamente a su presencia en selenoproteínas. Estas proteínas contienen el aminoácido número 21, la selenocisteína (Sec, U). Sec son aminoácidos codificados por el codón UGA, que en las proteínas normales corresponde a un codón de STOP. Para incorporar la Sec sin finalizar la proteína hay un mecanismo único y especializado que identifica este codón como selenocisteína y no como un codón de STOP. Este proceso requiere de elementos como la secuencia de inserción de selenocisteínas (SECIS) y muchos factores proteicos incluyendo un factor de elongación específico de EFSec y la proteína de unión a SECIS 2.
En el genoma humano, se han identificado 25 genes para las selenoproteínas. Selenoproteínas como la glutatión peroxidada, la tioredoxina reductasa y la iodotironina deiodinasa están involucradas en reacciones redox, y las Sec se encuentran en el residuo activo esencial para la actividad catalítica. Las selenoproteínas tienen funciones biológicas en oxidoreducciones, señalización redox, defensa antioxidante, metabolismo de la hormona tiroidea y en respuestas inmunes. Por lo tanto ellas tienen una fuerte relación con enfermedades humanas como el cáncer, la enfermedad de Keshan, infecciones víricas, infertilidad masculina y anomalías en las respuestas inmunes y en la hormona tiroidea.
La síntesis de selenoproteínas es un proceso que se ha conservado a lo largo de la evolución, aunque haya diferencias entre procariotas, eucariotas y archaea. La característica común a todos los organismos es el codón UGA que codifica para Sec, el tRNA específico, el elemento SECIS y algunos factores proteicos (binding proteins).
El elemento SECIS se trata de una secuencia cis situada en el extremo 3'UTR del RNA mensajero con una estructura stem-loop, y que permite insertar una selenocisteína en presencia de un codón UGA cuando es necesario, en el momento de la traducción (sin elemento SECIS, el codón UGA atura la traducción). El elemento SECIS está definido por unas secuencias características (nucleótidos en sitios concretos), y por una estructura secundaria característica. Esta estructura se deriva del apareamiento de bases del RNA, que normalmente presenta un apareamiento de bases no canónicos A-G, críticos para la función de éste. Además, normalmente presenta dos hélices, un loop interno y un loop apical. En bacterias, la región esencial del elemento SECIS incluye un sitio de unión para un factor de elongación especializado (eEFsec), que recluta a un tRNA específico que permite la incorporación del Sec en la secuencia aminoacídica.
La síntesis de Sec i tRNAsec resulta de la metabolización del seryl-tRNA. En este proceso intervienen las siguientes proteínas:
PSTK: Phosphoseryl tRNA kinase.
SPS1 y SPS2: Selenofosfato sintetasa.
SLA/LP: Selenocisteína Sintasa.
SECP43: Selenocysteine associated protein.
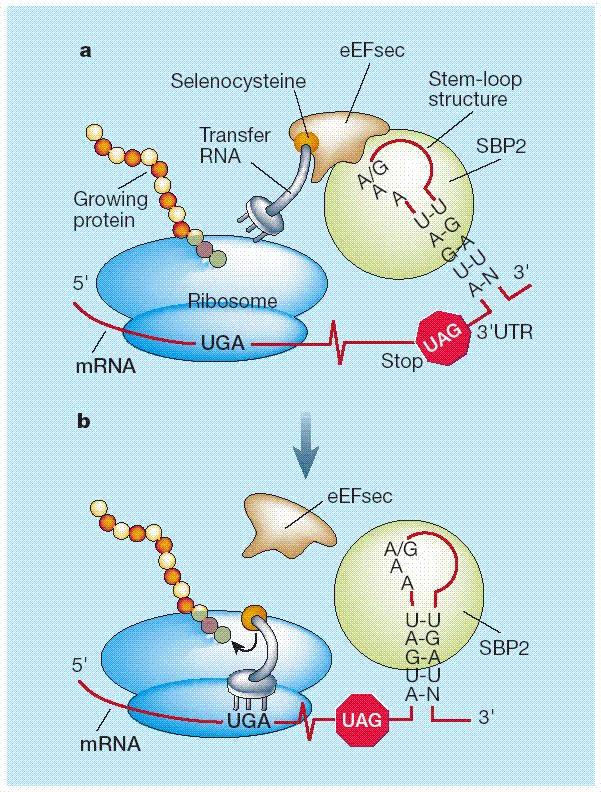
Para la recodificación del codón de terminación UGA como codón para selenocisteína es necesaria la presencia de un elemento SECIS (Selenocystein Insertion Sequence Element) en el extremo 3'UTR del gen.
Esta estructura tridimensional recluta a SBP2 (Secis Binding Protein), que une eEFsec (un factor de elongación eucariótico específico de selenoproteínas) y acerca el tRNA de selenocisteína (tRNAsec) al codón UGA del mRNA determinando la traducción a selenocisteína. La traducción de la proteína continua hasta el siguiente codón stop.
Antecedentes de selenoproteínas en Emiliania huxleyi
Anteriormente al trabajo que presentamos a continuación, ya se había estudiado el selenoproteoma de E.huxleyi.
En 2006 un grupo de investigación publicó un artículo donde se describía una selenoproteína encontrada en esta alga. (Obata, Shiraiwa, 2005: A novel eukaryotic selenoprotein in the Haptophyte Alga Emiliania huxleyi). Este artículo aportó nueva información, hecho interesante ya que aún no se conocen muy bien las bases del requerimiento de Selenio en organismos fotosintéticos. Este grupo describe cómo encontró 6 proteínas que contienen Selenio mediante ensayos con Selenio marcado radiactivamente. Una de estas 6 proteínas encontradas (de 27-kDa) fue aislada, y su cDNA fue clonado. Se puede observar como la proteína codificada por esta secuencia es homóloga a la proteína disulfito isomerasa (PDI), y contiene un dominio tioredoxin muy conservado (por lo que se puede deducir una función similar). La secuencia nucleotídica contiene un TGA in frame que codifica por una selenocisteína en la posición que corresponde al aminoácido cisteína en en el centro activo de PDI. A pesar de esto, no se encontraron elementos SECIS parecidos a los ya hay descritos en la secuencia de cDNA. Esto sugerió que existe un sistema distinto para insertar Sec asociado a este gen. De todos modos, se vio que esta proteína es necesaria para el crecimiento de E.huxleyi y que los niveles de su mRNA están estrechamente relacionados con la abundancia de selenio.
Más recientemente, en octubre de 2008, Araie H, Suzuki I, Shiraiwa Y. publicaron un artículo, "Identification and characterization of a selenoprotein, thioredoxin reductase, in a unicellular marine haptophyte alga, Emiliania huxleyi." En él se estudia la segunda selenoproteína más abundante en E.huxleyi, EhSEP1. Se caracterizó la proteína, viendo que se codificaba a partir de una secuencia de 1950 pares de bases, y que resultaba en un péptido de 495 aminoácidos y 52,2 kDa. Esta proteína muestra una similitud muy grande a la enzima Tioredoxin reductasa (TR). Además, se vio que contiene residuos de cisteína con actividad redox en el lugar de unión de FAD del dominio "oxidoreducción disulfito de nucleótios del tipo piridínico", que contiene también un dominio de dimerización, y que tiene una región C-terminal Gly-Cys-Sec-Gly que se sabe que actua como un centro redox adicional. En la región 3' UTR se encontró un elemento SECIS, similar a los encontrados previamente en animales. Esto sugirió que E.huxleyi contiene tanto elementos SECIS parecidos a los descritos anteriormente, como también elementos de inserción de Sec distintos (como se comenta en los resultados del primer artículo). Finalmente se vio que el tránscrito del gen era igual tanto en condiciones de exceso de Selenio como de déficit de éste, pero que la proteína traducida era mucho menor en el primer caso, de modo que el control en la síntesi de la proteína se da a nivel de traducción.