SelK
SelK és una selenoproteïna que té una funció antioxidant, es troba sobretot en els cardiomiocits.
Per buscar si la nostra espècie, L. Braziliensis, conté la selenoproteïna K, hem realitzat una comparació del seu genoma contra les selenoproteïnes d'Homo sapiens, Mus musculus, Drosophila melanogaster i Trypanosoma brucei.
Així, primerament hem fet un tBLASTn per veure si hi ha algun hit prou bo en l'alineament de la nostra espècie contra alguna de les quatre anteriors.
Observant els resultats, veiem com l'alineament de L. Braziliensis amb Trypanosoma brucei és l'únic que té un bon hit i un bon E-value:

Veiem com l'E-value no és gaire baix, però entra dins les premisses predites: acceptem E-values que siguin menors que 10-4. A més, veiem com en l'alineament ens alinea la selenocisteïna de T. brucei amb un codó Stop de L. braziliensis, tots dos representats amb un asterisc (*). Aquest fet ens pot fer pensar que aquest teòric codó stop és, en realitat, una selenocisteïna.
Cal dir que SelK de T. brucei és una selenoproteïna curta. Quan tenim una selenoproteïna amb aquestes característiques és difícil que surti un bon E-value, ja que hi ha poca seqüència a alinear. A més, veiem com en aquest cas l'últim aminoàcid que ens alinea és la possible selenoproteïna. Per veure si realment ho és o, per contra, es tracta d'un codó stop, el que fem és allargar l'alineament fins el següent codó stop. Només podem allargar l'alineament dos aminoàcids més, ja que ens trobem seguidament amb un altre asterisc (RS*). Això ens fa pensar que el primer asterisc alineat es tracta d'una selenoproteïna i, aquest segon asterisc és un codó stop.
Agafem doncs el còntig d'aquest alineament per poder treure la seqüència genòmica d'interès: gi|154346825|ref|NC_009327.1|
A partir del programa genewise, determinem l'estructura gènica d'aquesta regió d'interès, és a dir, obtenim els exons i els introns d'aquesta subseqüència. Els resultats ens mostren que en la seqüència predita només hi ha un exó.
En aquest cas, abans d'executar el programa genewise, cal executar la comanda fastarevcomp, ja que aquesta seqüència està en sentit revers.
A partir del genewise obtenim només un exó. Agafem el cDNA que ens ha predit el programa genewise i fem un fastatranslate per obtenir els 6 ORFs possibles. Per determinar quin dels 6 ORFs correspon a la possible seqüència codificant de la selenoproteïna, realitzem un blastp dels 6 ORFs contra la SelK de T. brucei. Dels resultats obtinguts, ens quedem amb la seqüència proteica que té el millor hit:
>gi|154346825|ref|NC_009327.1|:subseq(2352826,6353).[3001:3351].sp [translate(1)]
MAYIHNGRVVEKKPFTVRGFLLAFWGMLSLFARTLFTTQPMTQVVEEYQHPAPRAANAPANDGGILGSLRAMLNRRGGGGQ
ALGGGGNSGWAAAVNRRGGNVHTLPKAPISGGCASG
Sospitem doncs que SelK es troba en el genoma de L. Braziliensis. Per acabar-ho de confirmar realitzem un SECISearch. El resultat ens mostra dos possibles elements SECIS, però cap d'ells suficientment convincent.
Utilitzo el programa que ens han proporcionat els professors de l'assignatura a través de la terminal i del disc 8. La comanda per executar-lo és: $ /disc8/bin/SECISearch.pl -vp t a. On "a" és el nom que li hem posat a l'arxiu que conté la seqüència ja revers. Per mirar els resultats la comanda és: $ eog a.twil_png/*
Trobem en aquest cas un element SECIS que, per la posició on es troba i per la seva estructura, sembla un bon resultat.
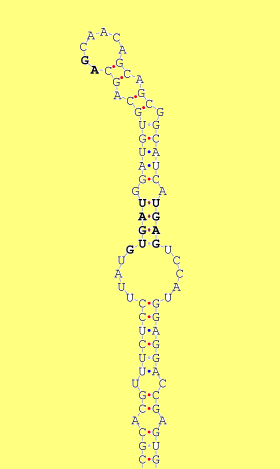
Hem realitzat també, per mitjà del programa InterPro, la cerca de dominis proteics i com a resultat ens ha donat:
Finalment hem realitzat un alineament múltiple amb les diferents espècies que presenten la SelK per veure si hi ha conservació entre elles després de la cisteïna.


