Selenoproteïnes en Plasmodium vivax
Clara Conill, Estel Enreig, Albert Gil, Carlos González, Jordina Guillén
Facultat de Ciències de la Salut i de la Vida
Universitat Pompeu Fabra
SEL 3
Sel 3 és una selenoproteïna que trobem en Plasmodium falciparum i que no es troba present en el genoma humà. És la tercera de les selenoproteïnes específiques trobades en P.falciparum.
RECERCA DE SEL 3 EN Plasmodium vivax
En aquest cas el TBLASTN tampoc el vam fer H.Sapien vs. P.vivax sinó que partírem de la seqüència d'aminoàcids de P.falciparum i ho blastejàrem contra tot el genoma de P.vivax.
El TBLASTN en el genoma de P.vivax usant Sel 3 de P.falciparum com a query va donar el següent resultat:
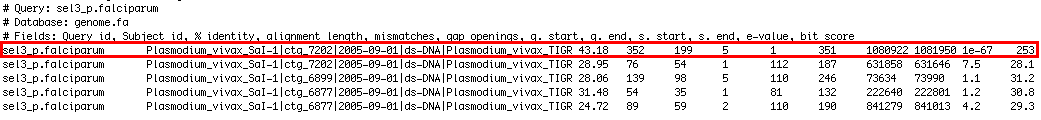 |
| Figura 1. Resultats TBLASTN |
| Descarrega't l'arxiu |
En aquest cas el contig amb l'e-value més petit (ctg_7202) era el que més bons resultats mostrava a nivell de % d'identitat, nombre de gaps, etc.
En la seqüència de Sel 3 que podem veure a PlasmoDB vàrem detectar que hi havia un error. Normalment senyalitzen la presència del TGA o selenocisteïna amb un * . En aquest cas, hi havia un * però també hi havia una U al costat. Això ens va fer dubtar, i al fer el BLAST vam poder veure com allà fallava quelcom. Vam analitzar la seqüència detingudament i vam trobar l'error. Al senyalitzar on era la selenocisteïna van posar una U on realment hi ha una G. Per confirmar-ho vàrem fer un BLAST de la seqüència modificada contra els mRNA de P.falciparum i aquests contenien una G en el lloc de la U.
>sel3_PlasmoDB_falciparum MILKKVYILIILILLSILSRTVDSU*SKQLHIKLPDEDDDYLAKLIGVFNDICTYSINNK EKIAKILSTSAVSVYTITSLYNSGITFKKNPHYSFFLPSQKYILKIINNNVNIQNNDIKK IDNLKDIPIFERTFTNKHNIQMSKTYILNNFINFINFLPYKLKKESLYNFSKYKEFDGLK YVENPFTQVQVLNHEDNIIVYQAKTKKHYWIHLPFKYNIIKISDDNTTSYILMFIPLYKY YSNYIIQMKITSNNNNQNVTFSSCIKQEKTEHIQNNSFHYDIIKNISKHITYDIINAIDN NINILYMRNVKPGKYNFINTTNALQKKTKNYKKKKFTLPSFKSLPFKFKRT >sel3_real_falciparum MILKKVYILIILILLSILSRTVDSG*SKQLHIKLPDEDDDYLAKLIGVFNDICTYSINNK EKIAKILSTSAVSVYTITSLYNSGITFKKNPHYSFFLPSQKYILKIINNNVNIQNNDIKK IDNLKDIPIFERTFTNKHNIQMSKTYILNNFINFINFLPYKLKKESLYNFSKYKEFDGLK YVENPFTQVQVLNHEDNIIVYQAKTKKHYWIHLPFKYNIIKISDDNTTSYILMFIPLYKY YSNYIIQMKITSNNNNQNVTFSSCIKQEKTEHIQNNSFHYDIIKNISKHITYDIINAIDN NINILYMRNVKPGKYNFINTTNALQKKTKNYKKKKFTLPSFKSLPFKFKRT
Així doncs, vam utilitzar la seqüència modificada per tal de realitzar tots els anàlisis següents.
Per una banda, amb els resultats del GeneWise (aquí) vam poder predir l'estructura del nostre gen i veure on es trobava el TGA que codifica per selenocisteïna i si aquest es trobava alineat amb el TGA de P.falciparum.
Aquí podem veure el tros de l'alineament on s'observa dit TGA:
sel3_p.falcipar 1 MILKKVYILIILILLSILSRTVDSG-SKQLHIKLPDEDDDYLAKLIGVF
M+L KVY+L IL+L + + V++G SK+LHIKLP+EDDDYL KLI +
MVLNKVYLLTILVLFYVSTLCVEAGXSKKLHIKLPNEDDDYLGKLINIS
subseq(1080850, 72 agtaagtccaacgcttgtactggggttaatcaaccaggggttgataaat
tttaatattctttttatcctgtacggcaatatatcaaaaaatgattatc
gtgtggctcccgggtctccatgaagacagacaggacgtctcgcaacctg
sel3_p.falcipar 49 NDICTYSINNKEKIAKILSTSAVSVYTITSLYNSGITFKKNPHYSFFLP
I Y+ NNK KIAK+L+TSA+S Y++ +Y SG+T++++PHYS+F+P
KKITQYARNNKGKIAKVLATSALSAYSLNWVYQSGVTLQRDPHYSLFVP
subseq(1080850, 219 aaaactgcaaagaagagtgatgctgttcatgtctggaccagccttttgc
aatcaacgaaagatcattcccctccactagtaacgtctagacaactttc
ggaaacgactggatcggataattaccactgacgcccagggcacccgtcc
Es pot veure com realment el GeneWise ens alinea el TGA de P.falciparum amb el TGA de P. vivax i com en ambdós casos, no es tracta d'un codó stop sinó que la traducció de la proteïna segueix.
A més a més, també vam fer un Exonerate per tal de poder assegurar-nos que l'estructura era correcta i així doncs, obtenir també els resultats en format gff. D'aquesta manera vam poder saber exactament quins eren els llocs d'splicing, l'inici i final del gen, etc.
Com a resum de l'estructura del gen:
# seqname source feature start end score strand frame attributes Plasmodium_vivax_SaI-1|ctg_7202|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome gene 1080922 1081953 1800 + .
gene_id 1 ; sequence Sel3_plasmo ; gene_orientation . Plasmodium_vivax_SaI-1|ctg_7202|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome cds 1080922 1081953 . + . Plasmodium_vivax_SaI-1|ctg_7202|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome exon 1080922 1081953 . + .
insertions 0 ; deletions 0 Plasmodium_vivax_SaI-1|ctg_7202|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome similarity 1080922 1081953 1800 +
. alignment_id 1 ; Query Sel3_plasmo ; Align 1080922 1 1032
| Figura 2. Resultats Exonerate (.gff) |
D'altra banda, vam fer un altre TBLASTN però en aquest cas de la nostra proteïna predita contra la base de dades d'EST i mRNA de P.vivax que podem trobar a l'NCBI. D'aquesta manera podem saber si la proteïna hipotètica s'expressa o no.
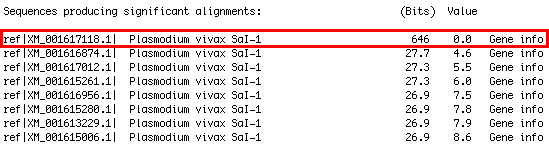 |
| Figura 3. Resultats TBLASTN mRNA |
| Descarrega't l'arxiu |
Però tota selenoproteïna ha de tenir també un element SECIS. Per tant, calia trobar-ne un en la seqüència genòmica adjacent al nostre gen. Com bé diu el protocol, gràcies al SECISearch vàrem poder predir l'estructura de l'element SECIS de la Sel 3.
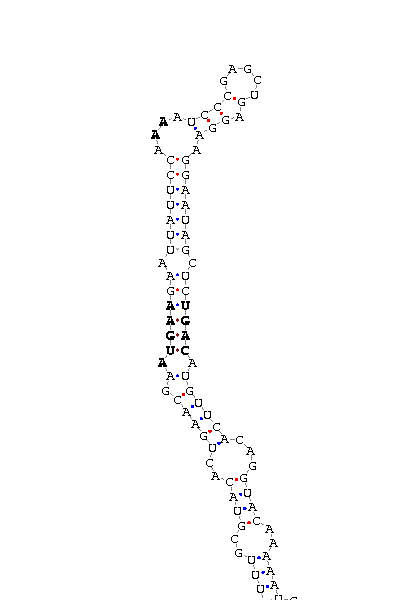 |
| Figura 4. Element SECIS de Sel 3 |
Per tal d'acabar de corroborar que la proteïna predita és homòloga a la proteïna de P. falciparum, i que té els dominis necessaris per a la seva funció, analitzàrem la seqüència amb l'InterProScan i vam veure que tant la nostra proteïna com la de P.falciparum tenen un pèptid senyal.
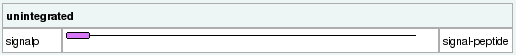 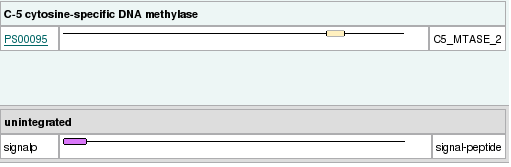 |
| Figura 5.Dominis EFsec P.falciparum (esquerra) i P.vivax (dreta) |
Podem dir doncs, que hem identificat Sel 3 en P.vivax i no només on es troba sinó també quina estructura té el propi gen i el seu element SECIS:
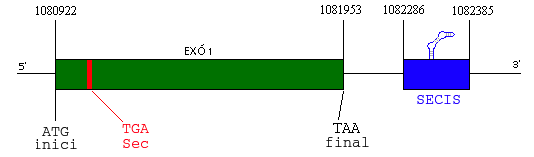
|
| Figura 6. Esquema Sel 3 |