Selenoproteïnes en Plasmodium vivax
Clara Conill, Estel Enreig, Albert Gil, Carlos González, Jordina Guillén
Facultat de Ciències de la Salut i de la Vida
Universitat Pompeu Fabra
SEL 1
Sel 1 és una selenoproteïna, és a dir, el que normalment seria un TGA com a codó stop, en aquest cas aquest codó codifica per l'aminoàcid selenocisteïna. Sel 1 està present en el genoma de P.falciparum però no en l'humà.
RECERCA DE SEL 1 EN Plasmodium vivax
En aquest cas el TBLASTN no el vam fer H.Sapien vs. P.vivax sinó que vam partir de la seqüència d'aminoàcids de P.falciparum i ho blastejàrem contra tot el genoma de P.vivax.
El TBLASTN amb el genoma de P.vivax usant Sel 1 de P.falciparum com a query va donar el següent resultat:
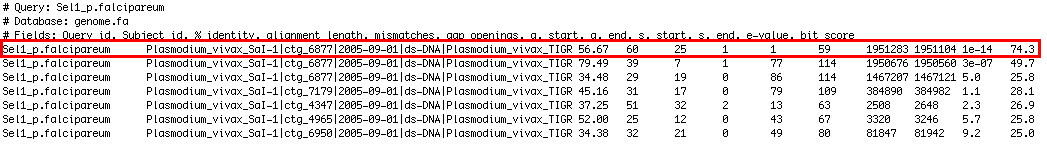 |
| Figura 1. Resultats TBLASTN |
| Descarrega't l'arxiu |
En aquest cas el contig amb l'e-value més petit (ctg_6877) era el que més bons resultats mostrava a nivell de % d'identitat, nombre de gaps, etc. Vam haver de buscar la seqüència complementària (mitjançant la comanda fastarevcomp des del Shell) ja que GeneWise no és capaç de reconèixer que es tracta d'una seqüència reverse.
Observàrem que hi ha un error d'annotació en PlasmoDB ja que la seqüència de Sel 1 que hi ha en aquesta base de dades no conté cap selenocisteïna. Semblava estrany que una proteïna catalogada com a selenoproteïna no contingués cap U, però al mirar en detall la regió genòmica on es trobava el gen vam observar que just després de l'aminoàcid que PlasmoDB considerava l'últim hi havia un TGA, que com era de suposar, no es tractava del codó sinó del TGA que codifica per selenocisteïna. A continuació hi havia una glicina i just després ja trobàvem el codó stop (TAA en aquest cas).
>Sel1_PlasmoDB MDDRKENKKNADYEIFLRDRKLKYEKIRKRNALHNKFILPIINFIKGIINFIKTVINILA IFFKTLLGQSNTSQSSNGKNNDDDDGFFKKKRTGGLPKSRIMELKNLGTCLGST >Sel1_real MDDRKENKKNADYEIFLRDRKLKYEKIRKRNALHNKFILPIINFIKGIINFIKTVINILA IFFKTLLGQSNTSQSSNGKNNDDDDGFFKKKRTGGLPKSRIMELKNLGTCLGSTUG
Així doncs, la seqüència que nosaltres considerem vàlida és la que té annotada la U
Per una banda, amb els resultats del GeneWise (aquí) vam poder predir l'estructura del nostre gen tot i que, per algun motiu, no ens alineava bé el TGA que hauria de codificar per selenocisteïna.
Aquí podem veure el tros on hi ha la U (surt com a X) de P.falciparum alineada amb un gap:
Sel1_falciparum 75 SSNGKNNDDDDGFF-KKKRTGGLPKS
+ +G+NND DD FF KKK+TG L KS
TLSGRNNDGDDDFFNKKKKTGNLQKS
subseq(1947500, 668 GTAACCC Intron 2 TAGacagcaagggggttaaaaaagatcaa
<0-----[668 : 818]-0>ctgggaaagaaattaaaaacgataag
ggcacctcatctcctggaagctgagc
Sel1_falciparum 100 RIMELKNLGTCLGSTXG
RIMELKNLGTCLGS
RIMELKNLGTCLGS--T
subseq(1947500, 897 aaagcaatgattga a
gttataatgcgtgg c
aagagacgcctgcc c
Però com que en la seqüència genòmica de P.vivax vam veure que just si que hi havia un TGA el que vam fer va ser recòrre al programa Exonerate per tal de veure com ens alineava la proteïna predita pel GeneWise amb la seqüència genòmica.
En aquest cas, l'alineament que obtenim és ja el correcte perquè podem veure que el TGA correspon a la X, la glicina que abans ens alineava amb una treonina ara s'alinea amb una altra glicina i just després ja apareix el codó stop. Aquí podem veure la part que conté tant el TGA com el codó d'stop.
94 : sLysThrGlyAsnLeuGlnLysSerArgIleMetGluLeuLysAsnLeuGlyThrC : 112
||||||||||||||||||||||||||||||||||||||||||||||||||||||||
sLysThrGlyAsnLeuGlnLysSerArgIleMetGluLeuLysAsnLeuGlyThrC
1950630 : AAAAACGGGCAATTTGCAAAAGAGCAGAATAATGGAACTGAAAAACTTGGGCACCT : 1950574
113 : ysLeuGlySerThr***Gly*** : 119
|||||||||||||||||||||||
ysLeuGlySerThr***Gly***
1950573 : GTTTGGGCAGCACCTGAGGTTAA : 1950551
L'error que comet GeneWise creiem que segurament es deu al fet que el TGA que codifica per la selenocisteïna es troba en l'antepenúltim codó i per tant, li és més difícil fer un bon alineament d'aquests tres últims codons.
L'esquema del gen obtingut gràcies a l'Exonerate és doncs:
# seqname source feature start end score strand frame attributes Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome gene 1950551 1951283 559 - .
gene_id 1 ; sequence sel1_plasmo ; gene_orientation + Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome cds 1951107 1951283 . - . Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome exon 1951107 1951283 . - .
insertions 0 ; deletions 0 Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome splice5 1951105 1951106 . - .
intron_id 1 ; splice_site "GT" Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome intron 1950882 1951106 . - .
intron_id 1 Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome splice3 1950882 1950883 . - .
intron_id 0 ; splice_site "AG" Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome cds 1950834 1950881 . - . Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome exon 1950834 1950881 . - .
insertions 0 ; deletions 0 Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome splice5 1950832 1950833 . - .
intron_id 2 ; splice_site "GT" Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome intron 1950683 1950833 . - .
intron_id 2 Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome splice3 1950683 1950684 . - .
intron_id 1 ; splice_site "AG" Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome cds 1950551 1950682 . - . Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome exon 1950551 1950682 . - .
insertions 0 ; deletions 0 Plasmodium_vivax_SaI-1|ctg_6877|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome similarity 1950551 1951283 559 -
. alignment_id 1 ; Query sel1_plasmo ; Align 1951284 1 177 ; Align 1950882 60 48 ; Align 1950683 76 132
| Figura 2. Resultats Exonerate (.gff) |
Un dels anàlisis més concloents a l'hora de dir si una proteïna està present o no en una espècie és veure si hi ha expressió del mRNA del gen en qüestió. Per aquest motiu, també vam realitzar un TBLASTN de la nostra proteïna predita contra la base de dades d'mRNA de P.vivax que hi ha a l'NCBI. En el cas de Sel 1 els resultats foren afirmatius, és a dir, P.vivax té expressió del mRNA de Sel 1.
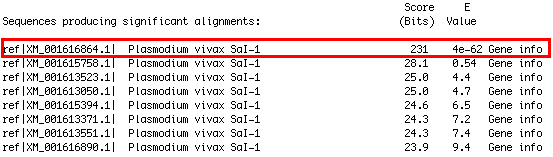 |
| Figura 3. Resultats TBLASTN mRNA |
| Descarrega't l'arxiu |
Però tota selenoproteïna ha de tenir també un element SECIS. Per tant, calia trobar-ne un en la seqüència genòmica adjacent al nostre gen. Com bé diu el protocol, gràcies al SECISearch vam predir l'estructura de l'element SECIS de la Sel 1.
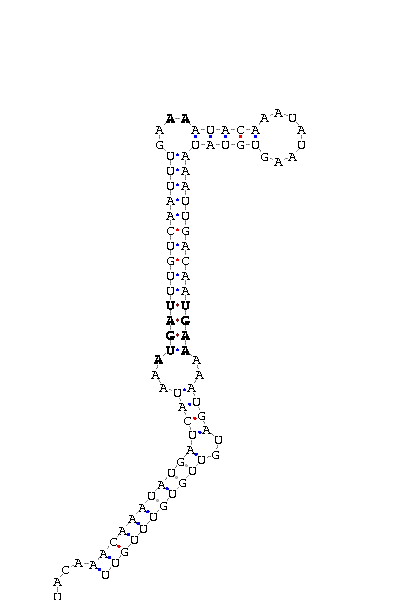 |
| Figura 4. Element SECIS de sel 1 |
L'anàlisis amb InterProScan aquest cop no ens ha portat a resutats interessants ja que no ha trobat cap domini conegut en Sel 1. Val a dir que no només no hi ha cap domini conegut en la Sel 1 de P.vivax sinó que tampoc se'n coneix cap per la Sel 1 de P.falciparum.
Després de tota la recerca, hem identificat Sel 1 en P.vivax i no només on es troba sinó també quina estructura té el propi gen i el seu element SECIS.
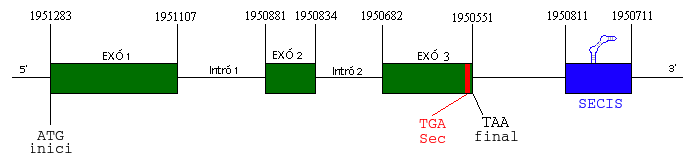
|
| Figura 5. Esquema Sel 1 |