Selenoproteïnes en Plasmodium vivax
Clara Conill, Estel Enreig, Albert Gil, Carlos González, Jordina Guillén
Facultat de Ciències de la Salut i de la Vida
Universitat Pompeu Fabra
CERCA DE NOVES SELENOPROTEÏNES
Un cop vàrem comprovar l'existència de totes les selenoproteïnes que ja havien estat descrites en el nostre organisme en estudis anteriors, vam intentar caracteritzar-ne de noves amb l'ajuda del programa SECISearch, que permet trobar seqüències putatives de SECIS amb un input en format FASTA. En el nostre cas el que vam fer es introduïr tot el genoma de P.vivax. Aquest programa et dóna un output on s'inclouen possibles seqüències que continguin elements SECIS amb un dibuix de l'estructura secundària predita d'aquests. També, et dóna un valor d'score per aquesta predicció i un valor mínim recomanat d'score per a considerar aquella seqüència com a un veritable lloc putatiu per a l'existència d'un element SECIS.
Vam obtenir un gran nombre de possibles elements SECIS però vam poder descartar-ne la gran majoria per presentar valors d'score inferiors a 15. Dels que presentaven valors superiors a 15 vam trobar els elements SECIS de les quatre selenoproteïnes esmentades en aquest treball i d'altres estudis anteriors (Sel 1, Sel 2, Sel 3, Sel 4). Però a més a més, vam trobar un element SECIS amb un score de 16,62:
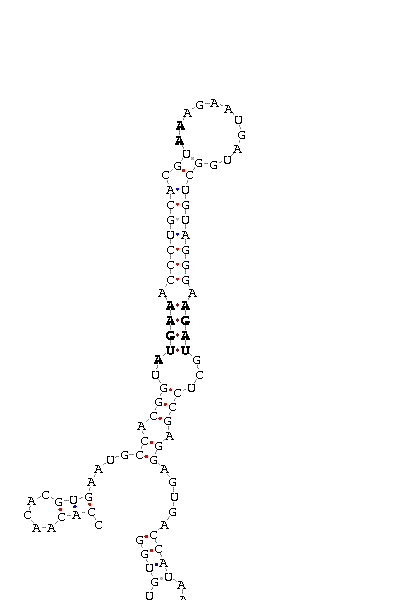 |
| Figura 1.Element SECIS |
El mateix programa ens indica a quin contig es troba l'element SECIS predit (ctg_6977). Per tant, localitzàrem aquest element SECIS en el nostre genoma (SECIS en blau).
...GGAATTTTCCCACCCCAAAAAAAAGGCGCCATAAAAATGGAACACACCCCTAAGA AGTGAGCACAACAGATAGGTAGTGTGTTCCACAAAGTTTTACGGACCATTCCACAA CACGTGAATGCCACGGTATGAAACCCTGCACGTAAAGAATGATGGCTGTAGGGAAGATGCTCCGAGGAGTGACCA TAAAATGTGGAAACGTGACAAACGCGGTGAGAGTTCCTAGCCGATTTATAAGCATGGCCCCTACGCGG...
Després, vam extreure d'aquell contig un fragment anterior al nostre element SECIS que podria contenir la seqüència d'alguna possible selenoproteïna. Amb aquesta seqüència vam realitzar un TBLASTX (resultat TBLASTX) contra una base de dades que conté tots els transcrits processats (PlasmoDB) en Plasmodium vivax.
Aquí podem veure la part de la seqüència de la putativa selenoproteïna que conté els TGA (tant el de la hipotètica selenocisteïna com el de l'stop que nosaltres predim):
...CCC AGG GAT TGA GCA GAT AAG GGG GCA TCA CAC ACA GGG CAG CGC GCC TTC GCA CCG GTC CTT TTG CTG ACG AAG GCT CAA AAA CGG GTG AAC AAA CTT TGG AGT TCA TTT TTT TGG GCG CAA AAA AAG CGG ACC ACA ACC ATT GGT TGT AAT TTT TTT TTT TTT TCC TTT TCA TTA GAC AAT GTG CCC CCC ACA CAC GCA TAC AAA TTT GTT AAG CGG CAC TCA CAATTC ATT TTT ACA ATT CAT TTT TTT TTA ATA TTT CTT CTT TCT TTA TGA TTTTTTAGGATCCTT...Descarrega't l'arxiu
A partir de la seqüència proteica existent a la base de dades i sabent quins són els últims aminoàcids vam poder determinar a la seqüència genòmica quin és el primer codó stop (marcat en verd a la seqüència). Aquest codó stop és un TGA, fet que podria ser indicatiu de que aquesta proteïna fos en realitat una selenoproteïna mal anotada. Just abans d'aquest codó stop trobem l'últim triplet que ens va alinear el TBLASTX (marcat en lila a la seqüència). Aquest alineament va ser molt significatiu amb un e-value de 0 amb el transcrit processat d'una proteína ja coneguda: la subtilisin-like protease (PV09935). Això ja podria ser bastant indicatiu de que no existeixen aminoàcids conservats després del codó stop.
Per tal de poder refutar completament la hipòtesi de que la subtilisin-like protease es tracti en realitat d'una selenoproteïna traduírem els triplets existents després del codó TGA (hipotètic codó codificant per una selenocisteïna) i vàrem realitzar un TBLASTN (resultat TBLASTN) contra els transcrits processats de Plasmodium vivax. Va quedar comprovat que el TBLASTN només fou capaç de donar-nos un alineament per aquells aminoàcids que es troben abans del TGA sospitós, és a dir, no es troba homologia després del codó stop, per tant, és molt poc probable que es tracti d'una selenoproteïna.
Finalment, vam comprovar que en el transcrit de la subtilisin-like protease no existeix cap element SECIS. De fet això ja ho havíem verificat abans amb la localització de l'element SECIS en el nostre genoma, ja que aquest està molt allunyat de la regió transcrita.
Per tant, amb les proves realitzades anteriorment podem concloure que l'element SECIS predit pel programa no correspon a cap selenoproteïna i que l'obtenció d'aquest es deu exclusivament a l'atzar. Com ja vam comentar inicialment, l'score obtingut per a aquest element SECIS era molt just si ho comparàvem amb l'score obtingut pels SECIS de Sel 1, Sel 2, Sel 3 i Sel 4.