Selenoproteïnes en Plasmodium vivax
Clara Conill, Estel Enreig, Albert Gil, Carlos González, Jordina Guillén
Facultat de Ciències de la Salut i de la Vida
Universitat Pompeu Fabra
eEFSec
eEFSec és un factor de traducció necessari per a la incorporació de selenocisteïna en les proteïnes. Probablement reemplaça EF-Tu (factor d'elongació) per la inserció de la selenocisteïna degut al codó TGA. Aquesta proteïna uneix GTP i GDP ja que forma part de la família de les GTP-binding elongation factor, concretament de la subfamília SelB.
RECERCA D'eEFSec EN Plasmodium vivax
Resultats TBLASTN:
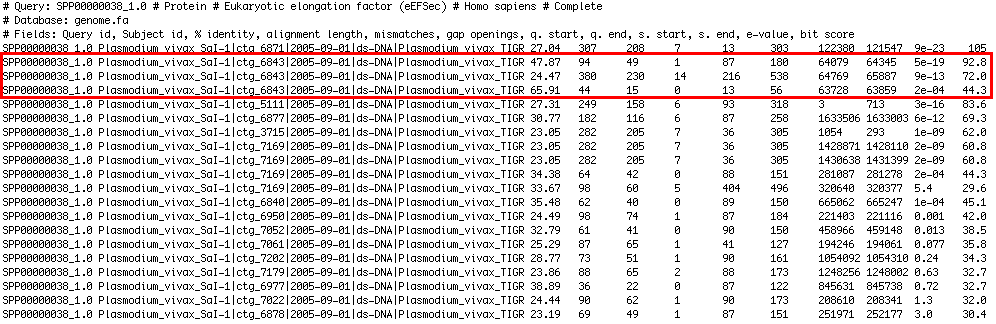 |
| Figura 1. Resultats TBLASTN |
| Descarrega't l'arxiu |
No vam escollir el contig amb l'e-value més petit ja que no era el que millor concordava amb la proteïna. La llargada era poca, molts els mismatches i poc % d'identitat. Així doncs, el contig que millor encaixava amb un possible homòleg era el ctg_6843
Després de delimitar la zona on hi havia teòricament el gen, vam analitzar-la pel GeneWise. D'aquesta manera, vam poder veure quina estructura tenia el nostre gen, i quina seqüència d'aminoàcids ens predia. Per problemes amb el GeneWise i el format .gff d'aquest programa, enlloc d'anotar els exons com a tals, els anotava com a diferents gens. Per tal motiu, el que vam decidir fer és un Exonerate mitjanant el Shell. Si en el cas del GeneWise la query era la seqüència aminoacídica de l'eEFSec humà i la seqüència de DNA era la de Plasmodium vivax, en el de l'Exonerate la query era la seqüència aminoacídica predita enfront al DNA de Plasmodium vivax.
D'aquesta manera, amb el GeneWise vam poder fer una primera aproximació (aquí) i amb l'Exonerate vàrem obtenir ja els resultats en el format .gff desitjat on es pot observar l'inici i final del gen, els exons, els llocs d'splicing, etc.
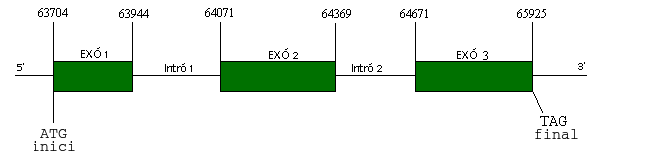
Com a resum de l'estructura del gen:
# seqname source feature start end score strand frame attributes Plasmodium_vivax_SaI-1|ctg_6843|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome gene 63704 66750 3533 + .
gene_id 1 ; sequence efsec_allargat ; gene_orientation + Plasmodium_vivax_SaI-1|ctg_6843|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome cds 63704 63944 . + . Plasmodium_vivax_SaI-1|ctg_6843|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome exon 63704 63944 . + .
insertions 0 ; deletions 0 Plasmodium_vivax_SaI-1|ctg_6843|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome splice5 63945 63946 . + .
intron_id 1 ; splice_site "GT" Plasmodium_vivax_SaI-1|ctg_6843|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome intron 63945 64070 . + .
intron_id 1 Plasmodium_vivax_SaI-1|ctg_6843|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome splice3 64069 64070 . + .
intron_id 0 ; splice_site "AG" Plasmodium_vivax_SaI-1|ctg_6843|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome cds 64071 64369 . + . Plasmodium_vivax_SaI-1|ctg_6843|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome exon 64071 64369 . + .
insertions 0 ; deletions 0 ; frameshifts 5 Plasmodium_vivax_SaI-1|ctg_6843|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome splice5 64370 64371 . + .
intron_id 2 ; splice_site "GT" Plasmodium_vivax_SaI-1|ctg_6843|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome intron 64370 64670 . + .
intron_id 2 Plasmodium_vivax_SaI-1|ctg_6843|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome splice3 64669 64670 . + .
intron_id 1 ; splice_site "AG" Plasmodium_vivax_SaI-1|ctg_6843|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome cds 64671 65925 . + . Plasmodium_vivax_SaI-1|ctg_6843|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome exon 64671 65925 . + .
insertions 0 ; deletions 0 Plasmodium_vivax_SaI-1|ctg_6843|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome splice5 65926 65927 . + .
intron_id 3 ; splice_site "GT" Plasmodium_vivax_SaI-1|ctg_6843|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome intron 65926 66476 . + .
intron_id 3 Plasmodium_vivax_SaI-1|ctg_6843|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome splice3 66475 66476 . + .
intron_id 2 ; splice_site "AG" Plasmodium_vivax_SaI-1|ctg_6843|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome cds 66477 66750 . + . Plasmodium_vivax_SaI-1|ctg_6843|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome exon 66477 66750 . + .
insertions 0 ; deletions 0 Plasmodium_vivax_SaI-1|ctg_6843|2005-09-01|ds-DNA|Plasmodium_vivax_TIGR exonerate:protein2genome similarity 63704 66750 3533
+ . alignment_id 1 ; Query efsec_allargat ; Align 63704 1 240 ; Align 64073 82 258 ; Align 64336
168 33 ; Align 64673 180 1251 ; Align 66478 598 273
| Figura 2. Resultats Exonerate (.gff) |
També vam fer un altre TBLASTN, però en aquest cas va ser de la nostra proteïna predita contra la base de dades d'EST i mRNA de P.vivax que podem trobar a l'NCBI. D'aquesta manera podem saber si la hipotètica proteïna s'expressa o no en P.vivax.
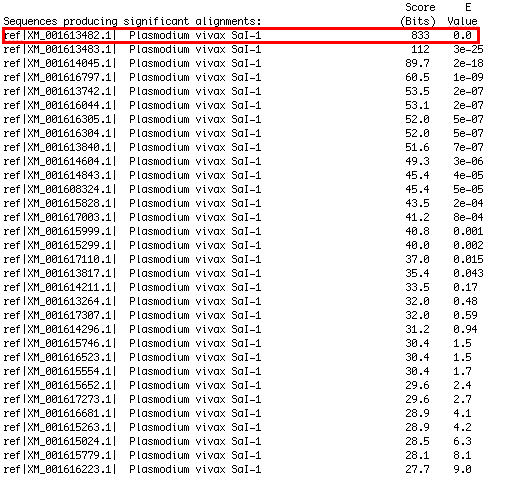 |
| Figura 3. Resultats TBLASTN mRNA |
| Descarrega't l'arxiu |
Per tal d'acabar de corroborar que la proteïna predita és homòloga a la proteïna humana, i que té els dominis necessaris per a la seva funció, analitzàrem la seqüència amb el programa InterProScan:
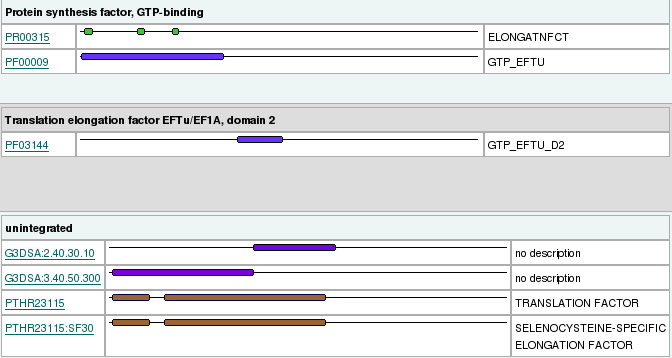 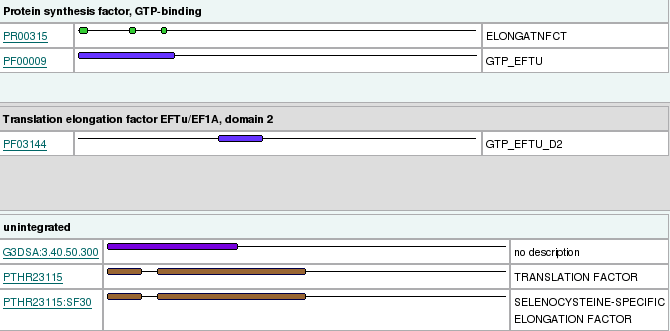 |
| Figura 4. Dominis eEFSec en H.sapiens (esquerra) i en P.vivax (dreta) |
Com era d'esperar, trobàrem en P.vivax els dominis GTPasa i de factor d'elongació específic de selenocisteïna necessaris per poder fer la seva funció.
Finalment, per resumir tota aquesta informació estructural aquí podem veure un esquema de la proteïna eEFSec de P.vivax:
|
| Figura 5. Esquema d'eEFSec |