| Selenoproteínas | Plasmodium knowlesi | El selenoproteoma de Plasmodium |
|---|
El selenio es un microelemento esencial para llevar a cabo diversas funciones, entre ellas hay que destacar la función antioxidante en animales, microorganismos y algunos eucariotas simples. Éste se incorpora en la vida a través de las selenoproteinas: proteínas en cuya secuencia existe un residuo de selonocisteína. Este tipo de proteína existe en los tres grandes dominios de la vida: archeas, eubacterias y eucariotas. En los dos primeros dominios solo se han encontrado en algunas archeas y en diversas especies de bacterias; mientras que en organismos eucariotas están presentes en animales y algas pero ausentes en plantas y hongos.
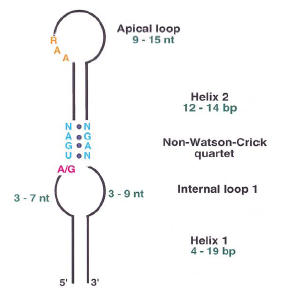
La selenocisteína es el ventiundécimo aminoácido codificado por el codón UGA (secuencia Sec). Sin embargo, este triplete se corresponde con un codón STOP, por esta razón, es necesaria la presencia de un elemento SECIS (SelenoCysteine Insertion Sequence Elements) que permita la recodificación del triplete.
 |
| Estructura de los elementos SECIS |
| Krol, 2002 |
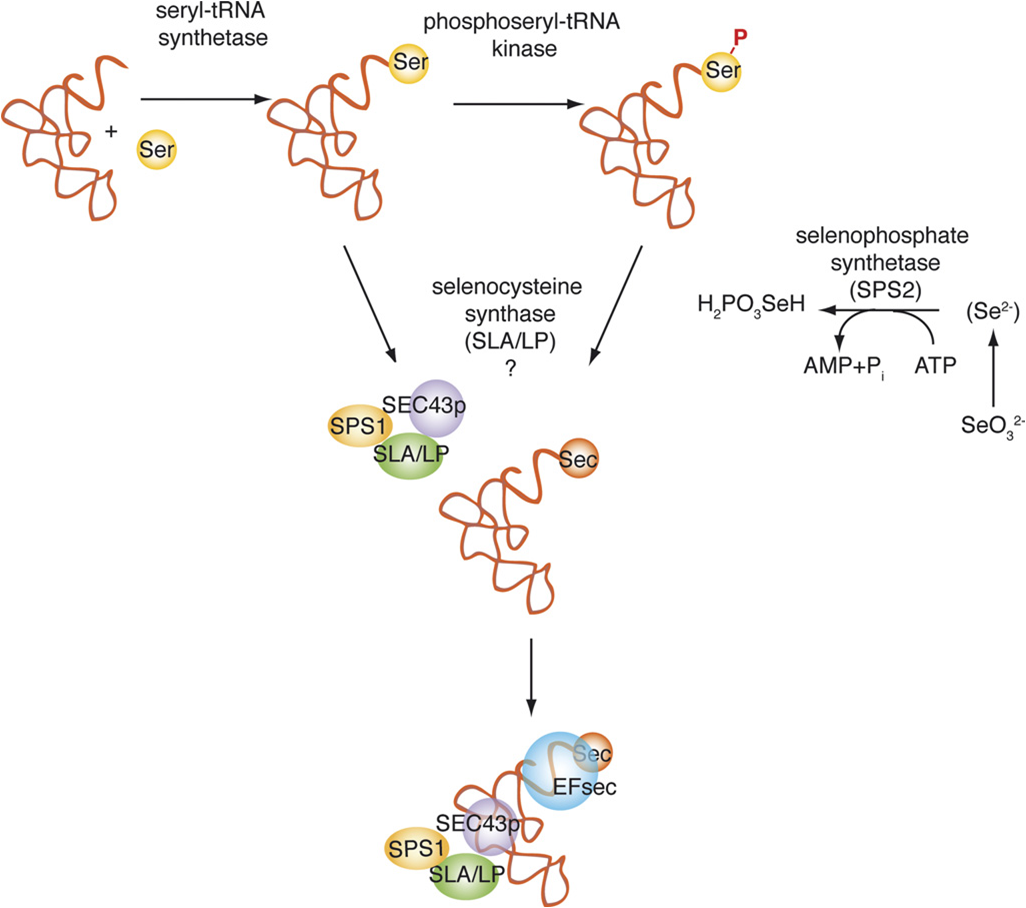
Además de los tres elementos trans (SBP2, eEFsec y tRNAsec) y el elemento cis (SECIS) es necesaria la presencia de la maquinaria que permita la síntesis del Sec-tRNASec: SPS1, SPS2, SLA/LP, Sec43p y PSTK. Actualmente, aún no se conoce muy bien la importancia de estas proteínas y su exacta función durante el proceso de síntesis. En el apartado de Resultados y Métodos de este trabajo se mencionan los artículos recientes que tratan cada una de estas proteínas. La imagen que se muestra a continuación ilustra la hipótesis aceptada hasta el momento (Allmang and Kroll, 2006).
La existencia de las selenoproteínas en los organismos ha sido un hallazgo científico relativamente reciente. Por esta razón, muchos de los genomas secuenciados hasta el momento no contemplaban la presencia de éstas (no se sabía que el triplete UGA podía codificar también para una selenocisteína). Así pues, muchos de estos genomas están actualmente mal anotados. Por este motivo, en el siguiente estudio, se pretende encontrar el selenoproteoma de Plasmodium knowlesi.
 |
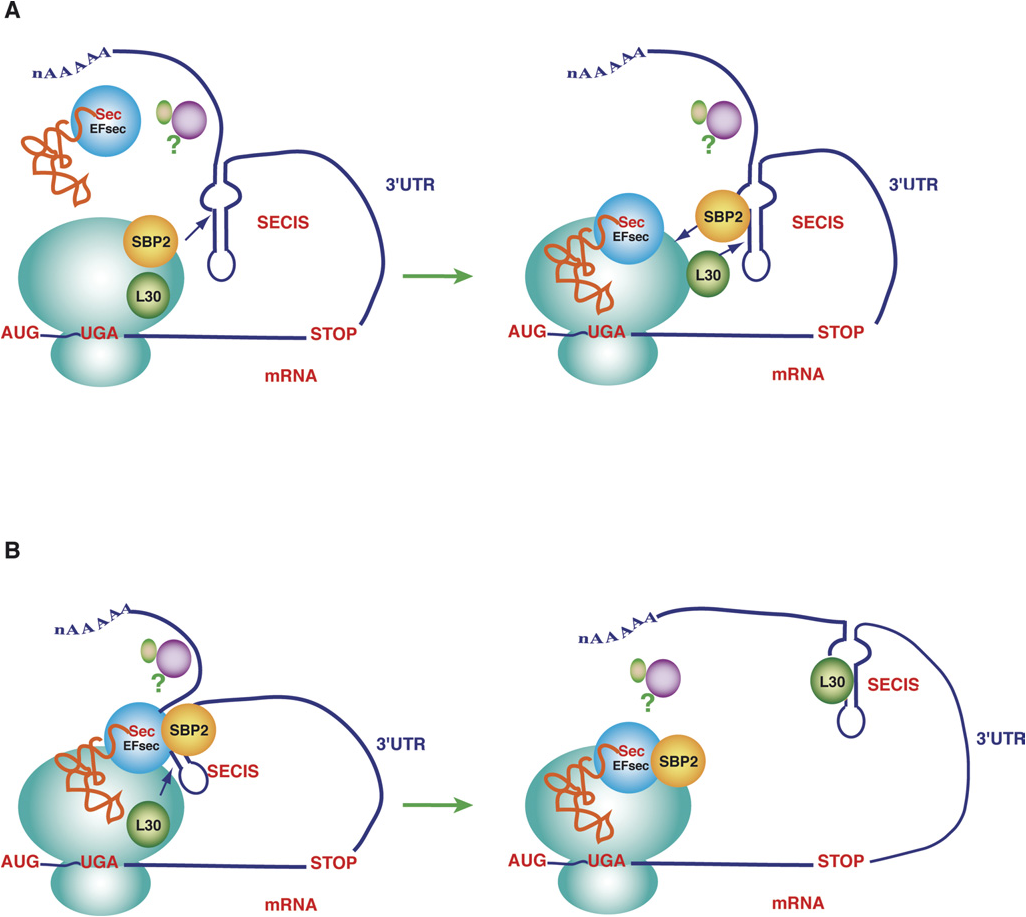 |
| Via de sintesis del Sec-tRNASec | Modelos de la via de sintesis de las selenoproteinas |
| Allmang and Krol,2006 | Allmang and Krol,2006 |
Plasmodium knowlesi es un protozoo parásito procedente del sur-este asiático que causa malaria en Macaca fascicularis y Macaca nemestrina, aunque también puede infectar a humanos. La enfermedad es transmitida por el mosquito Anopheles latens y Anopheles hackeri.
 |
 |
 |
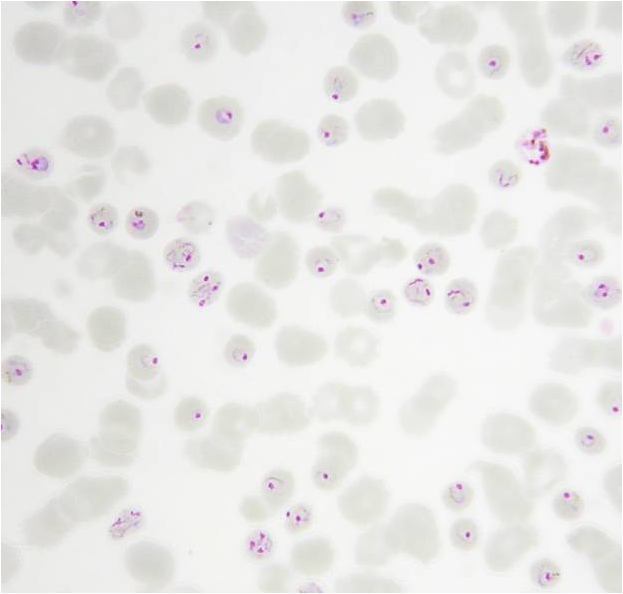
| Imagen microscópica de P.knowlesi | ||
| Cox-Singh et al, 2007 | ||
Se han detectado infecciones humanas en el sur-este asiático, especialmente en Malasia, pero también en la frontera de Thai-Burmese. En total, aproximadamente una quincena de casos han sido diagnosticados en Sarawak, Malasia y Borneo (Vythilingam I et al, 2006, Cox-Singh et al, 2007). Por un lado, el mosquito Anopheles latens infecta tanto a macacos como a humanos y se considera el principal vector de transmisión de humanos en los territorios de Kapit, Division de Sarawak y Malaysian Borneo. Por otro lado, en la población de macacos de Malasia, el mosquito Anopheles hackeri es el principal vector de transmisión. Aunque éste tiene la capacidad de transmitir el parásito a humanos no suele infectarlos, por esta razón, no se considera un vector de transmisión importante.
La enfermedad producida por Plasmodium knowlesi se diagnostica mediante exámenes parasitoscópicos de sangre (el aspecto de Plasmodium knowlesi es similar al de P.malariae) o mediante ensayos de detección molecular. Además, se usa el mismo tratamiento que para combatir las otras malarias aunque, en la actualidad, se están desarrollando muchas resistencias a estos fármacos. La imagen que se muestra a continuación ilustra el ciclo general de la malaria (Xinzhuan et al, 2007).
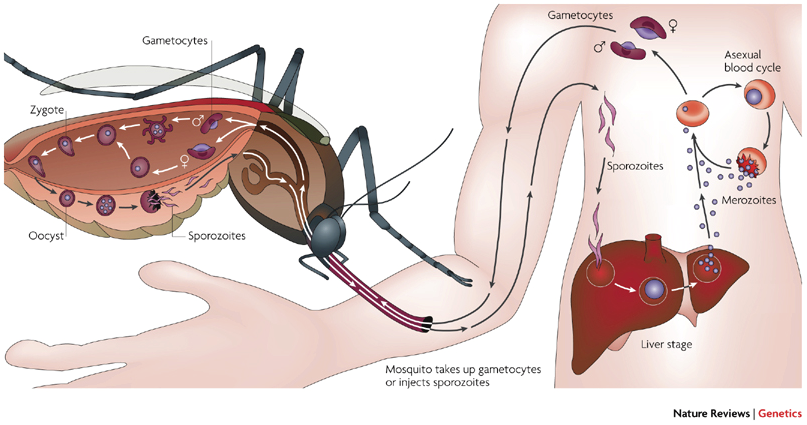 |
| Ciclo de infección de la malaria |
| Xinzhuan et al, 2007 |
El genoma de P.knowlesi ha sido secuenciado por el Sanger Institute. Así pues, con el estudio de su genoma se podrán buscar nuevas dianas farmacológicas para combatir la malaria, que permitan una solución al problema de las resistencias de los fármacos actuales (Coppel et al, 2004).
Existen cinco cepas diferentes de P.knowlesi: la cepa Malaya H (o simplemente designada como cepa H), la cepa Nuri, la cepa Philippine, la cepa Hackeri y, finalmente, la cepa Malaysian. El genoma de P. knowlesi utilizado en el siguiente estudio corresponde a la cepa H.
Recientemente en el género Plasmodium se han identificado cuatro selenoproteínas diferentes que no contienen homología con las selenoproteínas conocidas hasta el momento. Sin embargo, se encuentran muy conservadas entre las diferentes especies.
La secuencia de Sel1 está organizada en tres exones; Sel2 y Sel3 codifican para un solo exón y, finalmente, Sel4 se organiza en dos exones. En todas ellas el triplete UGA se encuentra en el último exón y el elemento SECIS en la región 3'-UTR, aunque separados por diferentes distancias. En Sel1 y Sel4, la Sec se encuentra en el penúltimo residuo del extremo C-terminal. Esta localización es característica de algunas selenoproteínas como las TRs y las Sel K, O y S. Además, estudios de estructura secundaria revelan similaridades entre la Sel1 y la Sel4 de Plasmodium con la SelK y la SelS de mamíferos, sugiriendo un posible origen común. Sel2 tiene la secuencia Sec en el extremo N-terminal como motivo CxxU (una cisteína, dos aminoácidos cualesquiera y la secuencia Sec). Este motivo se encuentra en diferentes selenoproteínas animales como Sel M, T, V y W. Además, en dos de éstas proteínas, junto con la Sel2 de Plasmodium, se les predice una helice α después del motivo. De esta manera, se sugiere que Sel2 podría tener una función redox en la célula. Finalmente, Sel3 contiene la secuencia Sec en el extremo N-terminal aunque, de momento, no se le conoce ningún motivo (Lobanov et al, 2006).
El hallazgo de estas cuatro selenoproteínas únicas hasta el mometo, sugiere una posible estrategia para el desarrollo de nuevos fármacos contra la malaria. Por este motivo, en el siguiente estudio hemos procedido, mediante análisis bioinformáticos, a la identificación del selenoproteoma de P. knowlesi, así como de la maquinaria necesaria tanto para la recodificación del triplete UGA como para la síntesis de selenocisteína.