Alba Ruiz de Andrés (alba.ruiz01@upf.edu.es) i Aitana Vidal Yago (aitana.vidal01@upf.edu.es)
Facultat de Ciències de la Salut i de la Vida
Universitat Pompeu Fabra

La proteïna Kua-UEV és un híbrid resultant de la fusió de 2 proteïnes anomenades Kua i UEV. Les proteïnes UEV són unes variants enzimàticament inactives dels enzims E2 conjugats amb ubiquitina. Regulen l'elongació no canònica de les cadenes d'ubiquitina. Presenten una forta conservació en molts organismes pel què fa a la seva estructura exònica i intrònica. Concretament, en humans la variant UEV1 ve codificada per un gen del cromosoma 20 anomenat UBE2V1,i és capaç de transcriure's independentment o bé de fusionar-se amb el gen recentment descobert que es troba en la regió contigua i que codifica per la proteïna Kua. El resultat de la fusió és un transcrit híbrid amb 2 dominis proteics.
Les proteïnes Kua constitueixen per elles soles una nova classe de proteïnes conservades, amb motius rics en l'aminoàcid histidina, i que acostumen a localitzar-se en estructures citoplasmàtiques de la cèl·lula. Alguns experiments han demostrat que Kua té la capacitat de localitzar en posició citoplasmàtica a la proteïna de fusió Kua-UEV, tenint en compte que UEV1 es localitza en el nucli cel·lular. A més, Kua dota de noves propietats biològiques a aquest coefector de la poliubiquitinació.
| -Inici- |
A continuació es mostren els resultats de la recerca. La informació ha estat estructurada per tal que distingim entre la informació del gen Kua, del gen UBE2V1 i aquella informació corresponent a la proteïna de fusió. Aquest estudi a tres nivells s'ha realitzat perquè hem considerat important demostrar les diferències que hi ha entre les 2 proteïnes independents i el resultat de la seva fusió, sobretot a nivell d'estructura genòmica, de conservació en espècies i d'expressió en teixits.
Kua forma part d'una família nova de proteïnes conservades. Té una posició cromosòmica dins el cromosoma 20, en la regió 20q13.13 i comprèn les bases 48,173,717 fins la 48,203,667. Amb una grandària de 29951 nucleòtids, aquesta proteïna té 6 exons que codifiquen per una seqüència nucleotídica de 270 aminoàcids.
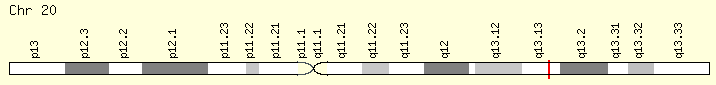
Imatge 1:Posició cromosòmica de Kua
Treballem amb la informació que ens proporciona la base de dades del RefSeq, ja que en fer l'alineament de la seqüència proteica de Kua-UEV amb la versió Kua els resultats obtinguts són millors. (veure alineaments). La base de dades de l'Ensembl no té informació de la proteïna independent, ja que només reconeix Kua com una regió de la proteïna de fusió, per tant queda descartada per a l'estudi de l'estructura genòmica de Kua.
Així doncs, considerem que el millor alineament és el següent:

Veiem que Kua ocupa la regió 5' de la proteïna de fusió. En color grana apareix la regió de Kua que formarà part de l'híbrid Kua-UEV, mentre que els aminoàcids encerclats en color lila formen part de la Kua independent, però es perden quan es fa la fusió amb UEV, ja que aquesta es col·loca en la regió 3', exactament quan acaba la regió colorejada en grana.
Obtenim la seqüència aminoacídica de Kua, que conté 270 aminoàcids

Kua té 6 exons. El primer exó conté una regió UTR en l'extrem 5' i una regió codificant. De la mateixa manera, l'exó 6 també té una regió UTR en 3' que ocupa 1268 parells de bases des del codó STOP que forma part de la regió codificant d'aquest mateix exó, fins la senyal de poliadenilació. La resta d'exons (2-5) són completament codificants.
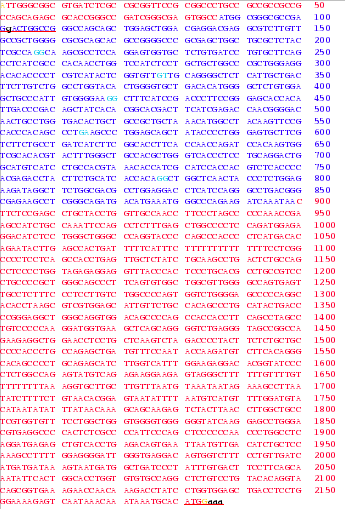
A continuació es mostra la seqüència nucleotídica del cDNA de Kua extreta de la base de dades RefSeq, des d'on s'ha fet l'estudi dels exons:
|
Els nucleòtids blaus són seqüència codificant, mentre que la part vermella són UTR (exons no codificants). Això ho sabem perquè a l'inici de la part codificant hi ha un codó ATG i al final hi ha un TAA. Cada inici i final d'exó es mostra amb blau clar. Observem que efectivament hi ha 2 exons flanquejants que tenen una regió codificant i una regió no codificant (al principi i al final de la seqüència), i 4 exons codificants. Per estudiar les zones de canvi entre exó i intró i comprovar si hi ha regions susceptibles de patir splicing, cal analitzar la regió genòmica sencera. Si observem la seqüència nucleotídica que ens proporciona RefSeq, veiem que efectivament tots els introns comencen amb gt i acaben amb g i que poden ser sotmesos a splicing. |
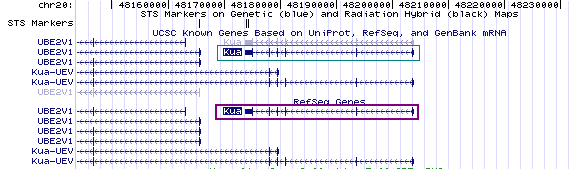
Com s'observa a la imatge, tant el transcrit de UCSC com el de RefSeq són iguals, per tant deduïm que Kua només té un transcrit possible.
En quant a isoformes, com es pot observar en la imatge anterior, Kua només presenta una isoforma, resultant del transcrit descrit anteriorment.
La proteïna UEV (variant d'ubiquitina conjugada amb l'enzim E2) és una proteïna que pertany a una subfamília dins de la família de proteïnes E2.
La seva seqüència és similar a altres enzims E2 conjugats amb ubiquitines, però els falta un residu de cisteïna, que és crític per l'activitat catalítica d'E2.
La proteïna codificada per aquest gen es troba localitzada al nucli i causa activació transcripcional del protooncogen humà c-FOS. Està involucrada en el control de la diferenciació, alterant el caràcter del cicle cel·lular (transició G2-M).
Després de fer varis alineaments amb diferents transcrits de la proteïna UEV contra la seqüència aminoacídica completa de la proteïna de fusió Kua-UEV, (veure alineaments), hem decidit treballar amb el següent transcrit (NM_001032288.1) perquè és el que alinea millor amb la nostra proteïna de fusió.
El resultat de l'alineament és el següent:

La UEV correspon a la part terminal de la proteïna Kua-UEV, a la regió més 3'. La major part dels nucleòtids alinien perfectament amb la proteïna de fusió, el que ens indica que gairebé tota ella formarà part de la proteïna final Kua-UEV. A més a més, l'inici d'aquest alineament encaixa amb el final de l'alineament de la proteïna Kua.
La seqüència aminoacídica de UEV conté 147 aminoàcids

Es troba al cromosoma 20, en la posició 20q13.2, específicament comprèn les bases de la posició 48163117 a la 48132712.

Està formada per 4 exons, dels quals 2 són codificants i els altres 2 són parcialment codificants, ja que contenen regions UTR en un dels seus extrems (a 5' i 3' pel primer i l'últim exó, respectivament).
Tot seguit es pot observar la seqüència nucleotídica del cDNA que codifica per UEV (obtinguda del refSeq: cDNA NM_001032288).

|
- El color blau representa la regió codificant: en tots els casos comença per un triplet ATG (codó d'inici, codificant per Metionina) i acaba amb una TAA (codó stop). - El color blau clar indica el final d'un exó i l'inici del següent. S'ha analitzat tota la seqüència genòmica , amb exons i introns, i s'ha vist que al final de cada exó hi ha els nt gt (que ens indica inici d'intró, lloc típic d'splicing) i al final de cada intró hi ha una g (també lloc típic d'splicing). - El color vermell representa la regió no codificant, els UTR. |
|
|
S'observa que en un dels transcrits es deu haver produït splicing alternatiu perquè desapareix un exó central. Els altres dos transcrits semblen iguals entre ells, i es diferencien del nostre perquè són lleugerament més llargs en la regió 5'. |
La següent taula mostra els 4 transcrits de la proteïna UEV, amb els seus exons corresponents:
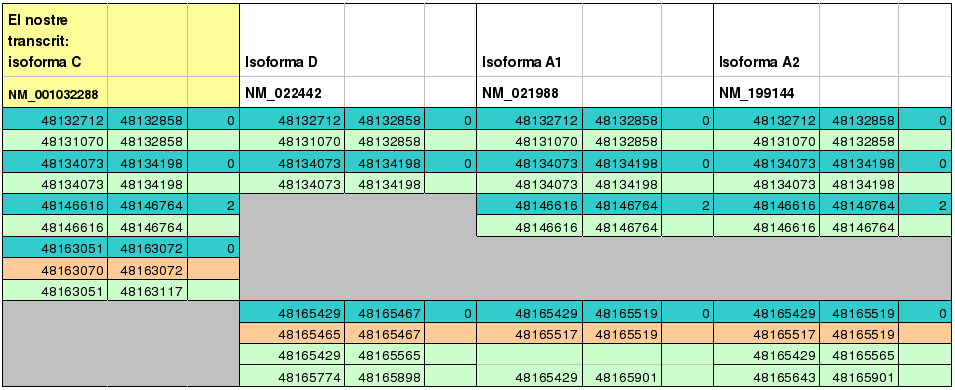
| _____ | Exon |
| _____ | CDS |
| _____ | Start Codon |
Taula 1:Posició dels exons, cDNA i codó start de cada isoforma de UEV. Reverse strand.
El comentari de la taula es farà seguint l'ordre de transcripció dels exons, com que està en reverse strand, s'iniciarà per les últimes caselles i es seguirà l'explicació en ordre ascendent.
Isoforma D
alineament de la isoforma D contra el nostre transcrit (isoforma C)
Aquesta isoforma conté 4 exons dels quals 1 és tot ell no codificant.(veure seqüència cDNA)
Iniciant la transcripció per 5'i en reverse strand:
- El primer exó que trobem és no codificant.
- El segon exó és parcialment codificant: es troba comprès entre les posicions 48.165.565 i 48.165.429; el codó start el trobem a la posició 48.165.467 (fins a la 48.165.465). Tot ell es troba en una regió upstream respecte el 1er exó del nostre transcrit (isoforma c).
- Seguidament es produeix un splicing alternatiu on es perden els 2 primers exons del transcrit de referència. Tot i així no s'ha produït canvi en la pauta de lectura perquè l'inici del següent exó que es tradueix coincideix amb l'exó corresponent del transcrit de referència.
- El tercer i el quart exó són idèntics als del nostre transcrit, fins i tot en l'extensió de les regions UTR .
Isoforma A1
alineament de la isoforma A1 contra el nostre transcrit (isoforma C)
Aquesta isoforma conté 4 exons.(veure seqüència cDNA)
Iniciant la transcripció per 5' i en reverse strand:
- El primer exó es troba en una regió upstream (5') respecte el nostre transcrit.
Destaquem que correspon a la mateixa regió on es troba el segon exó de la isoforma d (el 1er exó codificant que observàvem).
Aquest exó és parcialment codificant, conté una regió UTR bastant llarga. El codó start s'inicia a la posició 48.165.519.
El marc de lectura no ha canviat.
- Seguidament veiem que s'ha produït splicing alternatiu i s'ha saltat el 1er exó del nostre transcrit.
- Els 3 exons restants són idèntics als del nostre transcrit, per tant, el marc de lectura en aquest lloc no ha canviat.
Isoforma A2
alineament de la isoforma A1 contra el nostre transcrit (isoforma C)
Aquesta isoforma conté 5 exons(veure seqüència cDNA)
Veiem que és la mateixa isoforma que l'anterior, ja que hi ha dos transcrits que transcriuen pel mateix producte proteic, quedant així la mateixa isoforma.
Iniciant la transcripció per 5' i en reverse strand:
- El primer exó és tot ell no codificant i es troba comprès entre les posicions 48.165.901 i 48.165.643.
- El segon exó té una regió UTR més curta que el primer exó parcialment codificant de la isoforma 1A.
Es pot observar que aquests dos exons són un sol exó en la isoforma anterior. Aquesta és la causa de que la isoforma A1 i la A2 siguin dos transcrits diferents però la mateixa isoforma final.
- La resta d'exons són idèntics a la isoforma anterior (1A) i al nostre transcrit (isoforma c).
De la fusió dels gens Kua i UBE2V1 en resulta un transcrit que pot traduir-se de forma natural encara que és poc freqüent. La transcripció i traducció d'aquests gens fusionats dona lloc a la proteïna de fusió Kua-UEV.
Kua-UEV és una variant de la família d'enzims E2 conjugats amb ubiquitina, amb la diferència que els falten certs residus de cisteïna que resulten fonamentals per l'activitat catalítica d'aquests d'enzims.
Se situa en posició citoplasmàtica, i la seva importància a nivell biològic encara no està del tot determinada.
És codificada des de la posició 20q13.3 del genoma humà, ocupant una regió genòmica de 72598 nucleòtids que van des de la base 48131070 fins la 48203667, generant una proteïna de 370 aminoàcids.
A continuació es mostra la seqüència nucleotídica de Kua-UEV, diferenciant la part que correspon a Kua (lila) i a UEV (verd):

Per estimar el número d'exons que té la proteïna Kua-UEV mirem la seva seqüència de cDNA.
Podem observar que té 8 exons, dels quals el primer i l'últim contenen part de seqüència codificant i una part no codificant corresponent a regió UTR.
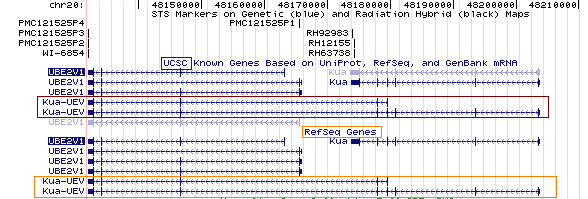
D'acord amb la informació de la base de dades del UCSC i el Ref Seq, veiem que Kua-UEV té 2 transcrits, l'un més llarg que l'altre. Si observem la imatge, veiem que el transcrit llarg és el resultat de la fusió dels gens UEV i Kua sencers, mentre que el transcrit curt comprèn tot el gen de UEV però només agafa una petita porció del gen Kua. Per aquest motiu hem treballat amb el transcrit llarg.
S'ha realitzat l'alineament dels 2 transcrits de Kua-UEV amb la seqüència aminoacídica de Kua per veure exactament la regió que ocupa Kua dins els dos híbrids diferents (a l'esquerra el transcrit llarg, a la dreta el transcrit curt).
 |
 |
Efectivament, podem comprovar que la proteïna de fusió més curta no alinia bé amb tota la seqüència de Kua ja que només en conserva una regió i per tant l'alineament resultant no surt correctament.
Desde les bases de dades de UCSC i RefSeq apareixen 2 isoformes de la proteïna de fusió Kua-UEV, que corresponen als 2 transcrits diferents.
| -Inici- |
| Espècies | Gen | Protein Identity | Dna Identity | Quocient Ka/Ks | Gen | Protein Identity | Dna Identity | Quocient Ka/Ks | |
 |
H.sapiens-P.troglodytes | Kua | - | - | - | UBE2V1 | 98.6 | 99.1 | 0.284 |
 |
H.sapiens-C.familiaris | Kua | 98.3 | 96.4 | 0.045 | UBE2V1 | 100.0 | 98.4 | 0 |
 |
H.sapiens-M.musculus | Kua | 93.3 | 88.5 | 0.080 | UBE2V1 | 93.9 | 87.1 | 0.038 |
 |
H.sapiens-R.norvegicus | Kua | 92.3 | 88.0 | 0.090 | UBE2V1 | 93.9 | 87.3 | 0.036 |
 |
H.sapiens-G.gallus | Kua | 76.5 | 69.0 | undef | UBE2V1 | 98.6 | 91.6 | 0.012 |
 |
H.sapiens-D.melanogaster | Kua | 59.8 | 61.3 | 0.122 | UBE2V1 | 66.9 | 59.3 | undef |
 |
H.sapiens-A.gambiae | Kua | 63.0 | 63.0 | 0.168 | UBE2V1 | 70.4 | 63.0 | undef |
 |
H.sapiens-A.thaliana | Kua | - | - | - | UBE2V1 | 53.7 | 56.0 | undef |
En aquesta taula podem observar els organismes pels quals tenim la proteïna ortòloga, tant pel què fa a Kua com respecte UEV.
D'acord amb la hipòtesi del rellotge molecular de l'evolució, que planteja una separació entre diferents llinatges evolutius tot utilitzant l’acumulació de diferències genètiques o mutacions al llarg del temps, el percentatge d'identitat esperat tant pel què fa a seqüència nucleotídica com de DNA hauria de ser més elevat per a espècies més properes (mamífers i aus) i hauria de disminuïr en relació amb espècies més llunyanes (insectes i vegetals).
Tanmateix, quan observem els ortòlegs per a Kua observem que en el cas de Gallus gallus no es compleix la hipòtesi del rellotge molecular, ja que presenta una identitat inferior a la que mantenen alguns insectes respecte l'humà, tot i ser una espècie molt més propera.
En el cas dels ortòlegs per a UEV ocorre un cas igualment desconcertant d'acord amb les prediccions que havíem fet inicialment: existeix una incongruència respecte el rellotge molecular en la relació entre Homo sapiens, Pan troglodytes i Canis familiaris, que s'estudia a continuació.
A nivell de DNA, i coincidint amb els resultats esperats, es pot observar que el percentatge d’identitat és més elevat entre Homo sapiens i Pan troglodytes que no pas entre Homo sapiens i Canis familiaris; en canvi, el percentatge d’identitat en la seqüència de proteïna és d’un 100% entre Homo sapiens i Canis familiaris i d’un 98,6% entre Homo sapiens i Pan troglodytes. Veiem per tant que, contràriament a la hipòtesis del rellotge molecular, la proteïna de l'espècie més llunyana (Canis familiaris) ha estat més conservada en l’evolució que la de l'espècie més propera (Pan troglodytes). En aquest cas, la proteïna en el llinatge que ha donat lloc a Pan troglodytes és molt probable que hagi sofert alguna modificació en algun aminoàcid que fa que sigui lleugerament diferent.
Per tal de poder veure si la selecció ha actuat de manera diferent en aquests llinatges, en la taula també es mostra el ratio Ka/Ks, que és la relació entre les taxes de substitucions no sinònimes (Ka) i sinònimes (Ks). Aquesta mesura ens permet saber si la selecció que ha actuat sobre un gen determinat ha estat positiva (si el valor és superior a 1), neutre (si el valor és semblant a 1) o negativa (si el valor és inferior a 1).
Un excés de canvis no sinònims (en els que hi ha canvis d’aminoàcids) respecte els canvis sinònims seria indicatiu d’una pressió de selecció positiva que afavoriria un canvi en la proteïna. En aquest cas, la relació Ka/Ks seria major que 1. Per contra, un excés de canvis sinònims indicaria una pressió de la selecció purificadora, on els canvis d’aminoàcid són eliminats, i per tant la proteïna es manté pràcticament inalterada al llarg de l’evolució (llavors tindríem una relació Ka/Ks inferior a 1).
Si observem a la taula els valors de Ka/Ks pels ortòlegs de UEV, podem veure que són molt inferiors a 1 (excepte per Pan troglodytes, cas que es comenta més endavant). Per tant, podem dir que UEV ha estat sotmesa a la selecció purificadora al llarg de la seva evolució, indicant el seu important paper funcional pels organismes.
Puntualment, en comparar el ratio Ka/Ks entre Homo sapiens i Canis familiaris podem veure que la selecció que ha actuat sobre la proteïna ha estat molt negativa, degut a què la proteïna té una identitat del 100% amb l’Homo sapiens, i efectivament la relació Ka/Ks es manté en 0.
De forma excepcional, veiem que el ratio Ka/Ks en Pan troglodytes té un valor sorprenentment alt comparat amb la resta de valors. Això s'hauria produït per una relaxació de la selecció purificadora, durant la qual s’han permès alguns canvis en aquesta espècie.
En pensar possibles causes d'aquesta modificació de UEV en Pan troglodytes, se'ns fa imprescindible observar què ocorre en el cas de Kua. En les taules superiors s'indica que no existeix homologia entre Homo sapiens i Pan troglodytes pel gen Kua.
Per tal d'estudiar més profundament el cas s’ha realitzat un arbre estimant les taxes evolutives sinònimes i no-sinònimes per branca de la seqüència codificant de la proteïna UEV, per veure si realment es produïa algun canvi interessant en l’evolució, especialment en els llinatges que han donat lloc a Homo sapiens i a Pan troglodytes.
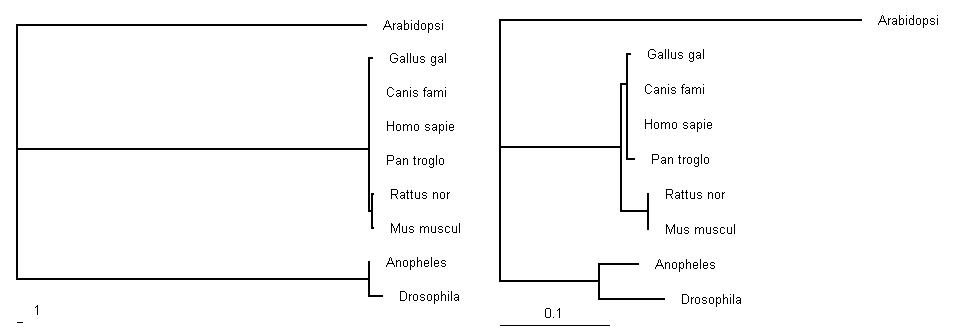
Imatge 2 esquerra:Arbre basat en Ka de UEV Imatge 2 dreta:Arbre basat en Ks de UEV
Imatge 3:Arbre no escalat amb el ratio Ka/Ks de les branques terminals
La topologia dels arbres permet diferenciar clarament les plantes, els insectes i els mamífers. Els dos arbres inicials estan escalats segons les distàncies evolutives. En el cas del primer arbre s'observen les taxes de substitucions no sinònimes (Ka), on es pot observar que les branques són molt curtes, degut a que els canvis no sinònims afecten a la proteïna codificada, per tant, si la proteïna és altament funcional tendeix a ser conservada, i per tant els canvis no són tolerats per la selecció i no es mantenen. L'única excepció és la planta Arabidopsi thaliana, que sí ha anat acumulant grans canvis (observem que la seva branca és molt més llarga que les altres).
En el segon arbre s’observen les taxes de substitucions sinònimes (Ks), on les branques internes són molt més llargues, el que ens indica que s’han produït molts canvis al llarg de l'evolució. Això és un resultat esperable, ja que els canvis sinònims no afecten a la proteina codificada, per tant, no són eliminats per selecció i es poden acumular de manera neutra.
Al tercer arbre les branques no són proporcionals a la distància evolutiva, però al costat de cada espècie podem observar el ratio Ka/Ks de les branques terminals. Aquest ratio mostra la pressió selectiva que ha sofert la proteïna a cada espècie des de que es va separar de l’espècie més propera. Podem observar que la selecció ha estat fortament negativa en gairebé tots els casos (la majoria de valors són molt menors que 1), és a dir, hi ha hagut una forta pressió purificadora per tal de conservar la proteïna al llarg de l’evolució recent.
Sorprenentment (i confirmant les sospites anteriors) s’observa que la ratio de Ka/Ks de la proteina UEV al llinatge de Pan troglodytes és superior a 1, el que ens indica que podria haver patit un període de selecció positiva en la seva evolució més recent. Aquesta data és sorprenent, ja que essent les espècies molt properes entre elles, esperaríem que també estiguessin conservades (igual que la resta de l'arbre), i en canvi, veiem que hi han hagut canvis importants per la biologia d'aquestes espècies.
A més a més, la branca interna que dona lloc a l'ancestre comú d'humans i ximpancés també té un valor de Ka/Ks molt alt, essent de 999.000. òbviament, aquest és el valor màxim de PAML quan no pot estimar el ratio Ka/Ks (pensem que hi ha hagut canvis d'aminoàcids i no hi ha hagut gairebé cap canvi sinònim, per tant el quocient Ka/Ks és un número molt gran que per defecte s'expressa com 999.000). Aquests valors ens indiquen que en aquestes dues branques s'han produït canvis no sinònims que han estat afavorits per la selecció, i que s'han acumulat en Pan troglodytes.
En conclusió, Pan troglodytes ha patit modificacions en el gen UEV, i ha acumulat substitucions que han estat mantingudes, per tant, la proteïna resultant varia respecte la versió de la proteïna ancestral. Aquest resultat coincideix amb els valors obtinguts anteriorment en el ratio Ka/Ks, on observàvem que UEV havia sofert variacions.
| Espècies | Protein Identity | Espècies | Protein Identity | ||||||||||||||||||||||
 |
H.sapiens-P.troglodytes | 99% |  |
H.sapiens-M.mulatta | 98% | ||||||||||||||||||||
| H.sapiens-T.belangeri | 81% |  |
H.sapiens-B.taurus | 96% | |||||||||||||||||||||
| H.sapiens-F.catus | 93% |  |
H.sapiens-G.gallus | 78% | |||||||||||||||||||||
 |
H.sapiens-D.novemcinctus | 92% |  |
H.sapiens-E.telfairi | 59% | ||||||||||||||||||||
| H.sapiens-E.europaeus | 84% |  |
H.sapiens-D.rerio | 86% | |||||||||||||||||||||
| H.sapiens-O.latipes | 72% |  |
H.sapiens-X.tropicalis | 95% | |||||||||||||||||||||
| H.sapiens-M.domestica | 79% |  |
H.sapiens-S.cerevisiae | 49% |
| -Inici- |
Kua s'expressa en varis teixits però amb poca abudància. En la següent taula es destaquen aquells on hi ha més expressió.
Taula 2: Expressió de Kua en teixits rellevants Mètode: GNF Expression Atlas 2 Data from U133A and GNF1H Chips |
A la següent taula es pot observar que Kua es localitza sobretot al teixit immunitari. Es pot trobar en menys expressió a teixits del sistema respitarori, neuronal i a la sang. Per apreciar la caracterització de Kua en els teixits que no apareixen en la següent taula, clicar el següent enllaç: microarray_Kua |
UEV s'expressa en molts teixits, dels quals destaquem els següents:
Taula 3:Expressió de UEV en teixits rellevants. Mètodes: GNF Expression Atlas 2 Data from U133A and GNF1H Chips |
Tal i com es pot obervar en aquesta taula superior, UEV s'expressa en alguns teixits on s'expressava Kua i a molts altres teixits diferents (veure els microarrays complets). La seva expressió és més àmplia que la de la proteïna Kua. Per apreciar la caracterització de UEV en els teixits que no apareixen en la següent taula, clicar el següent enllaç:microarray_UEV |
Kua-UEV s'expressa en varis teixits, dels quals destaquem els següents, ja que són els teixits on hi ha més expressió.
Taula 4:Expressió de Kua-UEV en teixits rellevants. Mètode: GNF Expression Atlas 2 Data from U133A and GNF1H Chips |
Es pot observar que la proteïna de fusió Kua-UEV s'expressa bàsicament als mateixos teixits on s'expressa Kua, i no als teixits on s'expressa únicament UEV. Per apreciar la caracterització de Kua-UEV en els teixits que no apareixen en la següent taula, clicar el següent enllaç:microarray_Kua-UEV |
| -Inici- |
Kua
Després d'obtenir la seqüència promotora de Kua localitzada en la regió 1kb upstream del lloc de començament de la transcripció (TSS) i 100bp downstream del TSS, creem una llista a partir del programa PROMO de tots els factors de transcripció que en principi podrien unir-se a aquesta regió promotora. (Resultat PROMO). Els factors de transcripció que tenen més probabilitat d'estar regulant l'expressió del gen de Kua seran els que tinguin un valor de RE equally menor, i en segon lloc aquesta habilitat dels factors de transcripció vindrà deteminada pel paràmetre de dissimilaritat (com més petit, més semblança amb la regió promotora i per tant més afinitat amb aquesta).
Aquest és el criteri per crear la llista dels millors factors de transcripció, seleccionant aquells que tinguin valors de RE equally menor a 0,1 i que mantinguin un valor de dissimilaritat petit (fins a 9%).
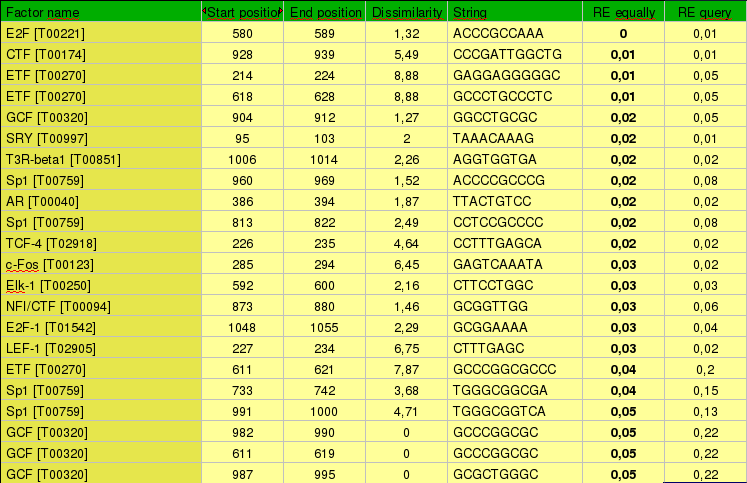
Taula 5:Factors de transcripció que tenen més possibilitat de regular l'expressió de Kua.
UEV
De la mateixa forma que Kua, cal seleccionar els factors de transcripció que poden unir-se a la seqüència promotora de UEV amb més afinitat. Del resultat que proporciona PROMO, a continuació es mostren els factors seleccionats per la seva capacitat de regular l'expressió del gen UBE2V1. El criteri de selecció per crear el llistat següent és el mateix que el realitzat per la proteïna Kua.
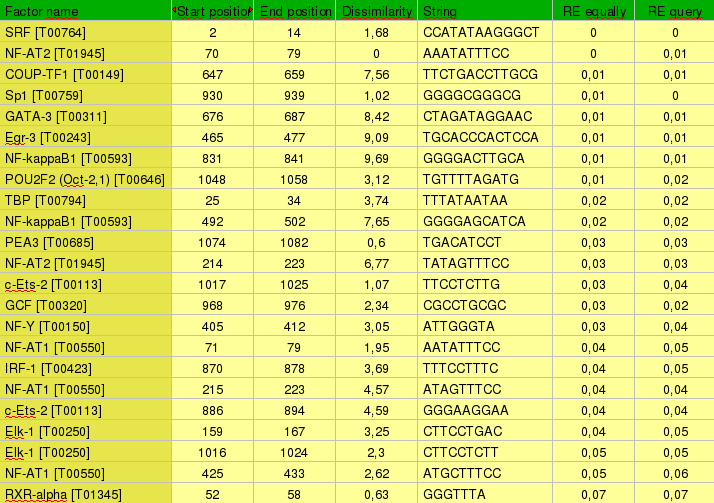
Taula 6:Factors de transcripció que tenen més possibilitat de regular l'expressió de UEV.
Kua-UEV
Finalment, fem el mateix procediment per Kua-UEV, obtenint en primer lloc la seqüència promotora de Kua-UEV i a continuació la llista dels millors factors de transcripció, obtinguts del resultat del programa PROMO:
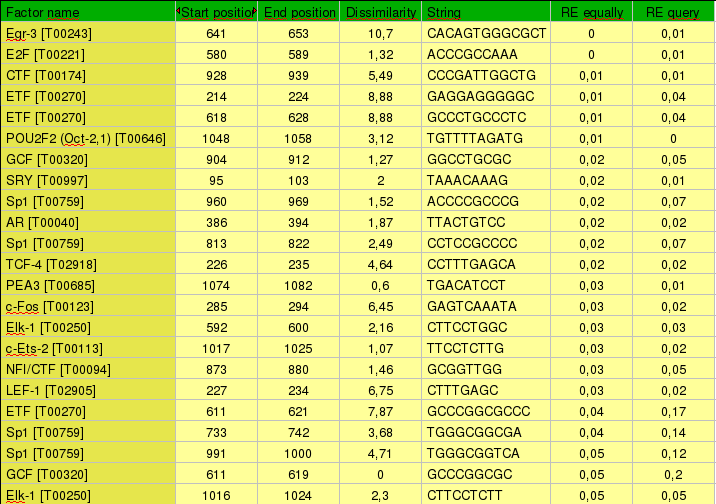
Taula 7:Factors de transcripció que tenen més possibilitat de regular l'expressió de Kua-UEV.
Programa
La implementació en Perl d'aquest algorisme la podeu trobar al següent enllaç: Reconeixement de factors de transcripció en una seqüència promotora
Interpretació
A continuació es poden observar els resultats de l'anàlisi de la unió de varis factors de transcripció en la regió promotora de les proteïnes Kua, UEV i Kua-UEV mitjançant el programa en Perl dissenyat específicament per aquesta finalitat.
S'ha estudiat la possibilitat d'unió de 13 factors de transcripció diferents a les regions promotores abans esmentades, per veure si algun d'aquests podia estar implicat en la regulació de l'expressió del gen corresponent.
Kua
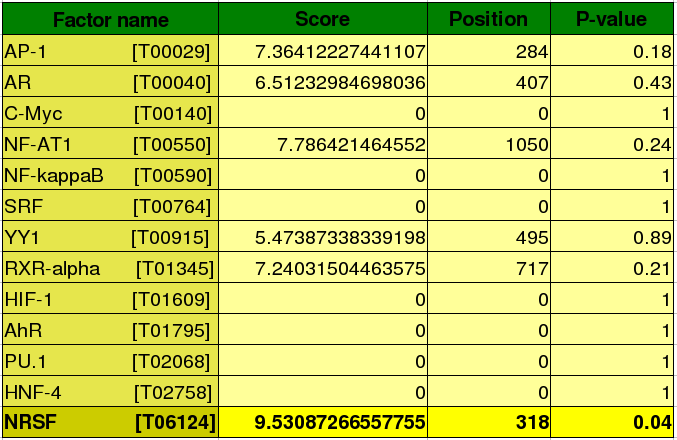
Taula 8: p-value dels 13 FT amb la seqüència promotora de Kua.
Tal i com es pot observar en la taula superior, tan sols un dels 13 factors de transcripció analitzats s'uneix a la seqüència promotora de Kua (tenint en compte que s'ha utilitzat un p-value de 0.09 com a llindar de tolerància).
Aquest factor és l'NRSF: el p-value obtingut és de 0.04, un valor força significatiu, per tant, podem dir que l'NRSF és un factor modulador de l'expressió del nostre gen.
UEV
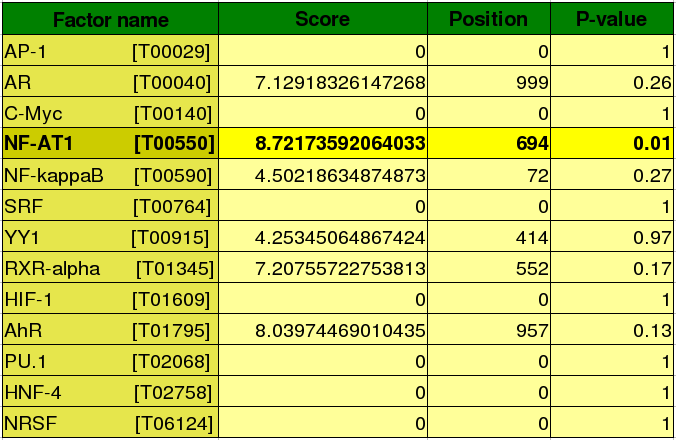
Taula 9: p-value dels 13 FT amb la seqüència promotora de UEV
Dels factors de transcripció anteriors, només el NF-AT1 s'uneix a la seqüència promotora de UEV, exactament en la posició 694. El valor de p-value obtingut és de 0.01, resultat molt significatiu.
Kua-UEV
En fer l'anàlisis dels possibles factors de transcripció que s'uneixen a la regió promotora de la proteïna de fusió, prèviament ens plantegem la hipòtesi de què haurien d'aparèixer els mateixos factors que per la proteïna Kua, ja que aquesta constitueix la primera part que es transcriu de la proteïna de fusió, per tant hauria de tenir la mateixa regió promotora.
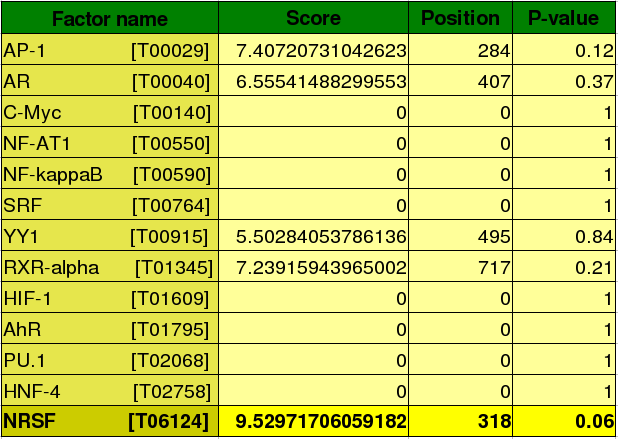
Taula 10: p-value dels 13 FT amb la seqüència promotora de Kua-UEV
Efectivament, es pot observar que l'únic factor que s'uneix a la seqüència és l'NRSF, igual que en la seqüència promotora de Kua. El p-value és lleugerament superior, de 0.06.
Cal dir que a la resta de FT, tot i que no els considerem com a vàlids, s'han obtingut uns p-values i uns scores molt semlbants als de Kua. Les posicions de la seqüència promotora on s'unirien també són les mateixes en ambdós casos. Per tant, confirmem que el promotor de Kua és el mateix que el de la proteïna de fusió Kua-UEV.
A continuació es mostren aquells factors de transcripció que coincideixen amb els resultats obtinguts a partir de PROMO i el programa en Perl. Hem considerat que els factors que tenen més probabilitat d'induïr l'expressió dels gens són aquells que mantenen un p-value i RE equally inferior a 0,1, i una dissimilaritat inferior al 9%. Tot factor de transcripció que presenti valors més elevats dels que hem seleccionat com a llindar creiem que no seran tan bons per unir-se a la regió promotora de cadascun dels gens i activar la seva transcripció.
| Factor name | p-value | RE equally | Dissimilarity |
| AP1 | 0,18 | 0,05 | 5,5 |
| AR | 0,26 | 0,13 | 6,97 |
| NRSF | 0,04 | - | - |
D'acord amb el programa realitzat amb llenguatge Perl el factor de transcripció amb un p-value menor que podria induïr l'expressió de Kua d'una forma més eficient és el NRSF, que precisament no apareix en la llista dels factors seleccionats pel programa PROMO. Segurament això és degut a què la matriu de pesos que utilitzen els dos programes és diferent.
Per altra banda, els factors de transcripció AP1 i AR apareixen tant en els resultats de PROMO com en el programa, i tot i que en el PROMO tenen un resultat significatiu, el valor de p-value que dona el programa és massa grans per a considerar-los significatius.
| Factor name | p-value | RE equally | Dissimilarity |
| AR | 0,26 | 0,13 | 6,97 |
| NF-AT1 | 0,01 | 0,04 | 1,95 |
| NF-KappaB | 0,27 | 0,01 | 9,69 |
| RXR-alpha | 0,17 | 0,13 | 6,54 |
D'acord amb el programa realitzat amb llenguatge Perl el factor de transcripció amb un p-value menor que podria induïr l'expressió de UEV d'una forma més eficient és el NF-AT1, que també apareix en la llista dels factors seleccionats pel programa PROMO, amb uns valors de RE equally i dissimilaritat acceptables d'acord amb el llindar que havíem considerat.
Per altra banda considerem que els factors de transcripció AR, NF-KappaB i RXR-alpha podrien ser bons candidats per induïr l'expressió de UEV d'acord amb PROMO, però presenten valors de p-value massa elevats per considerar-los bons.
| Factor name | p-value | RE equally | Dissimilarity |
| AP1 | 0,37 | 0,05 | 5,5 |
| AR | 0,12 | 0,02 | 1,87 |
| NRSF | 0,06 | - | - |
De la mateixa manera que amb Kua, el factor de transcripció que podria induïr l'expressió de Kua-UEV d'una forma més eficient segons el programa de Perl és el NRSF, tot i que en la llista de PROMO no existeixi. Per altra banda, els factors AP1 i AR apareixen tant en el PROMO com en les matrius analitzades segons el programa, però tampoc els considerem com a bons perquè tenen valors de p-value poc significatius.
| -Inici- |
La proteïna UEV és una proteïna localitzada al nucli que pertany a una subfamília dels enzims E2 conjugats a ubiquitina.
Les proteïnes de la família E2 són un dels 3 enzims essencials que participen al procés d'ubiquitinació. Específicament catalitzen la següent reacció:
ATP + ubiquitin + protein lysine = AMP + diphosphate + protein N-ubiquityllysine.
Hi ha moltes classes d'enzims E2 (uns 30 en humans), la majoria tenen un residu actiu de cisteïna al domini catalític per poder desenvolupar la seva acció. UEV pertany a una subfamília de proteïnes E2 que no contenen aquest residu de cisteïna, el que les fa catalíticament inactives, però tot i així, sembla que també actuen en la formació de la cadena de poliubiquitinació, específicament en l'elongació no canònica, el que pot provocar complicacions no esperades en ubiquitinació.
|
Es creu que les UEV poden dirigir i modificar accions de proteïnes com les UBC en processos biològics. L'UEV forma un complexe específic amb Ubc13 (necessària per l'ensamblatge de les cadenes de poliubiquitina) a través de la Lys 63 ( i no a través de la Lys 48 com és usat generalment en els processos canònics d''ubiquitinació). Aquesta unió, contràriament als processos normals, fa que el complexe no sigui reconegut pel proteosoma , per tant les proteïnes a les que s'uneix no contenen el targget per destruir-se. Es coneixen bastantes vies metabòliques que utilitzen aquesta variant de poliubiquitinació. Per exemple destaquem la via en la que intervé com a efector final l'NF-kB, on no es produeix la destrucció d'una proteïna sinó que hi ha modulació de senyals de transcripció (s'acaba alliberant el factor NfkB-Rel, que entra al nucli i regula la transcripció de varis gens). Les UEV estan implicades en incrementar la selectivitat i diversitat en les conjugacions d'ubiquitina, a més a més, aquesta nova via d'ubiquitinació que utilitzen pot ser una nova forma de regulació de varis processos cèl·lulars i bioquímics. |
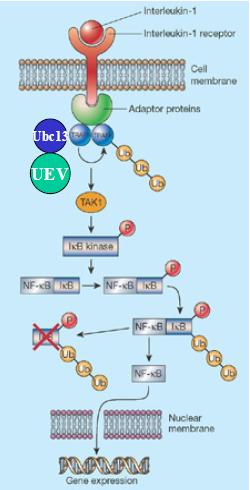 Imatge 4 Una de les vies metabòliques on intervé UEV. Imatge 4 Una de les vies metabòliques on intervé UEV.
|
També se sap que les UEV poden actuar en la regulació del cicle cel·lular, alterant-ne la distribució cel·lular (específicament acumulant cèl·lules en la fase G2-M). Per tant, sembla que les UEV estan involucrades en el control de la diferenciació i semblen tenir un rol important en la regulació fisiològica de les transicions del cicle cel·lular.
A més a més s'ha vist que les UEV poden modular la transcripció de l'oncogen c-FOS, el que pot provocar, en certs casos, càncer de colon.
Clicar als links de gene ontology per a saber més informació de caràcter general:
Funció molecular:
GO:0004842 ubiquitin-protein ligase activity
Procés biològic:
GO:0006464 protein modification
GO:0006512 ubiquitin cycle
Kua és una proteïna recentment descoberta, es troba al mateix cromosoma que UEV, en una regió més upstream. És una proteïna transmembrana que es troba localitzada es estructures citoplasmàtiques.
No es coneix bé la funció que fa. Tanmateix, segons l'article "Fusion of the human gene for the poliubiquitination coeffector UEV1 with Kua, a newly identified gene", la descoberta d'un ortòleg en un bacteri (CarF) pot ajudar a descobrir quina és la funció de Kua per similaritat. Una possibilitat podria ser que Kua actués com un sensor general de les reaccions redox.
Les conseqüències d'aquesta funció en la proteïna de fusió Kua-UEV encara no se saben.
Kua té la capacitat de localitzar en posició citoplasmàtica a la proteïna de fusió Kua-UEV, tenint en compte que UEV1 es localitza en el nucli cel·lular, de manera que en formar-se la proteïna de fusió és redireccionada al citoplasma enlloc del nucli. Es localitza perinuclearment, en una estructura citoplasmàtica, possiblement al reticle endoplasmàtic.
Les funcions de la proteïna de fusió semblen ser les mateixes que les de la UEV, però, com que es localitza citoplasmàticament, és possible que utilitzi altres substrats en realitzar la polibuquitinació. Per tant, els productes finals seran diferents.
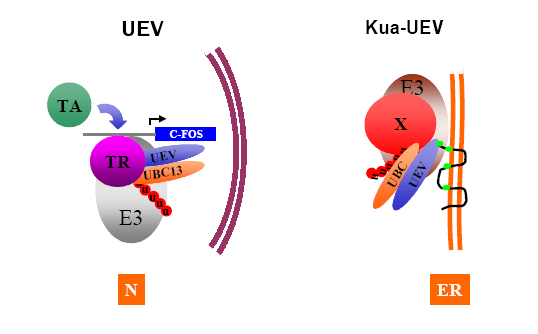
Imatge 5: Comparació de la funció de UEV i de Kua-UEV. La primera es localitza al nucli i la segona al citoplasma
Clicar als links de gene ontology per a saber més informació de caràcter general:
Funció molecular:
GO:0004840 ubiquitin conjugating enzyme activity
GO:0004842 ubiquitin-protein ligase activity
GO:0016563 transcriptional activator activity
Procés biològic:
GO:0000074 regulation of progression through cell cycle
GO:0000209 protein polyubiquitination
GO:0006282 regulation of DNA repair
GO:0006355 regulation of transcription, DNA-dependent
GO:0006464 protein modification
GO:0006512 ubiquitin cycle
GO:0030154 cell differentiation
| -Inici- |
En iniciar l'estudi de la proteïna, tansols tenim la seva seqüència aminoacídica: hem de saber a quina proteïna pertany i en quina espècie es troba. Inicialment realitzem un blastp (blast de proteïna contra proteïna) on aliniem la seqüència d'aminoàcids que tenim amb totes les seqüències existents a la base de dades. El resultat és una llarga llista de possibles proteïnes que alinien correctament amb la nostra seqüència proteica, cada una amb un percentatge d'identitat lleugerament diferent. De totes les que tenen una bona puntuació escollim la més alta (100% d'homologia): és la "ubiquitin-conjugating enzyme E2 Kua-UEV isoforma 1", d'Homo sapiens.
Busquem informació en varis papers per determinar quin tipus de proteïna és. Finalment veiem que és una proteïna de fusió formada per dues proteïnes que venen codificades per dos gens diferents: Kua i UEV.
Iniciem la búsqueda en les bases de dades per tal d'estudiar els dos gens per separat, i ens trobem en un conflicte: la informació obtinguda dels diversos llocs és molt diferent. En la base de dades de l'ensembl no apareixen les dues proteïnes per separat, simplement apareix la de fusió. Busquem noms sinònims per les dues proteïnes i tot i així no trobem res: l'ensembl mostra la proteïna de fusió (on hi podem arribar des del nom de Kua-UEV, Kua, UBE21V...) amb molts transcrits, dels quals algun d'ells són una de les dues proteïnes que volem estudiar. En veure que aquesta base de dades no ens dóna la informació necessaria per realitzar l'estudi, busquem al UCSC.
Inicialment fem un BLAT per tornar a alinear la seqüència proteica inicial amb la base de dades del UCSC i assegurar-nos de quina és la nostra proteïna. Ens apareixen un llistat de possibles resultats i agafem el de l'score més alt (1097), ens diu que la proteïna té 370 aminoàcids (igual que la que hem d'estudiar) i un 99,8% d'identitat amb la seqüència proteica que li hem donat. Triem aquesta informació i en entrar al genome browser del UCSC, veiem que és la Kua-UEV. En aquesta base de dades ens mostra la proteïna de fusió, i, per altra banda, les dues proteïnes per separat (es troben seguides al mateix cromosoma, per tant, ho podem veure tot des d'una mateixa finestra).
Iniciem l'estudi de la proteïna des del genome browser, utilitzant la base de dades del UCSC. Cal dir que sempre hem partit des d'aquesta base de dades, però que hem acabat utilitzant la informació cedida per RefSeq per una sèrie d'arguments que s'explicaran a continuació.
Per iniciar l'estudi dels dos gens, hem volgut aconseguir la seva seqüència. Per al gen de Kua ha estat fàcil perquè tan sols ens apareixia un transcrit i per tant, és el que hem utilitzat, però per l'estudi de UEV ha sigut més complicat, ja que ens apareixien 4 transcrits, tan a l'UCSC com a RefSeq. Per tant, inicialment hem mirat quin transcrit és el que concorda amb la part 3' de la proteïna de fusió.
S'ha realitzat un clustalw per cada transcrit contra la seqüència proteica de la proteïna de fusió per veure quin alineava millor. Els transcrits utilitzats els hem obtingut des del genome browser i han estat: el primer que apareix al UCSC, el primer que apareix a RefSeq, (eren els dos que se'ns mostraven d'entrada com a candidats immediats) i el transcrit més llarg (el quart de RefSeq: l'hem utilitzat per si de cas era el correcte, ja que en ser més llarg, potser contenia més part de la proteïna de fusió). També s'ha realitzat un clustalw de la proteïna de Kua contra la proteïna de fusió. Després de comparar els resultats s'ha vist que el millor alineament en el qual la proteïna de fusió queda coberta completament, és utilitzant el primer transcrit de UEV de la base de dades del RefSeq (NM_001032288.1). Per tant, a partir d'aquest punt del treball, ens hem basat principalment amb la informació obtinguda des d'aquesta base de dades, tan per UEV com per Kua.
Des de RefSeq hem pogut obtenir el cDNA i el DNA genòmic de cada proteïna, per tal de poder observar els diferents exons que contenien, quina regió era codificant, possibles regions UTR, etc.
Per fer l'estudi dels transcrits i les isoformes de UEV hem utilitzat, per una banda, les seves seqüències proteiques per tal d'alinear-les amb la seqüència de UEV amb el clustalw i veure la seva homologia. Per altre banda, hem fet un table browser de cada transcrit, demanant que el format de sortida fos en GTF (gene transfer format), d'aquesta manera hem aconseguit la posició de cada exó, la regió exacta on es troba el CDS (inici i final), la posició de l'start codon, la posició dins el codó on es produeix l'inici de lectura per realitzar la traducció... S'ha realitzat una taula comparativa pels quatre transcrits de UEV i així s'ha pogut veure la diferència entre ells: el nombre d'exons codificants i no codificants, el canvi de pauta de lectura i el número d'isoformes finals.
Per estudiar la proteïna de fusió hem utilitzat bàsicament la base de dades del RefSeq i en ocasions la del UCSC. L'Ensembl també proporcionava informació vàlida per Kua-UEV, però l'hem descartat perquè no ens permetia estudiar els dos gens Kua i UEV per separat, i hem considerat que és més lògic treballar sempre des de la mateixa base de dades per les 3 proteïnes. Per a l'estudi de les isoformes de Kua-UEV també s'ha extret informació de UniPROT però inicialment aquesta no coincidia amb la informació que ja teniem de RefSeq, ja que apareixien 7 isoformes. Tanmateix, en fer els alineaments de cada isoforma amb la seqüència d'aminoàcids de Kua-UEV s'ha comprovat que una d'aquestes isoformes en realitat té la seqüència idèntica a la Kua independent, 4 són idèntiques a les isoformes que havíem estudiat de UEV, i Finalment només han quedat 2 isoformes, que efectivament coincideixen amb les 2 proteïnes Kua-UEV obtingudes previament des de RefSeq.
Per tal de trobar els ortòlegs de cada proteïna estudiada s'han realitzat varis procediments:
- Inicialment hem accedit des de la pàgina del genome browser on teníem el nostre gen localitzat a la pàgina del NCBI Map Viewer: hem anat a parar directament a la regió cromosòmica d'interés i hem clicat a l'enllaç "hm" (homology). Des d'allà hem obtingut les espècies que tenen el gen conservat. Hem analitzat varis alineaments (fets amb el Blast) i hem obtingut una taula de "pairwise scores". Amb aquesta informació hem pogut realitzar taules d'ortòlegs per cada proteïna.
Paral·lelament hem relitzat un blast de les seqüència mRNA de les proteïnes estudiades per comprovar la veracitat del pas anterior.
- Cal dir que la informació de la taula d'espècies ortòlogues que hem realitzat per la proteïna de fusió prové de l'ensembl, ja que la informació obtinguda era més completa. En buscar la informació al NCBI (igual que en els dos casos anteriors), tansols ens apareixien 4 espècies ortològues, per tant, hem decidit utilitzar les dades obtingudes des de l'ensembl.
Per tal de realitzar l'arbre filogenètic entre les espècies hem realitzat els següents passos: obtenir les seqüències en cDNA de totes les espècies ortòlogues, alinera-les amb el clustalw i introduir-les al programa MEGA 3.1. Al MEGA 3.1 hem fet la transformació de fitxers per tal de que es poguessin llegir correctament i hem realitzat un arbre de NJ.
- Per tal de realitzar l'arbre de les taxes evolutives hem seguit els següents passos: hem obtingut el cDNA de les diferents espècies i hem realitzat un alineament de totes elles amb el Revtrans (s'introdueix la seqüència CDS, el programa fa l'alineament basat en aminoàcids, i seguidament et presenta la informació altre vegada en DNA). Finalment, per obtenir les taxes de substitució del CDS, s'ha utilitzat el software PAML (Phylogenetic Analaysis by Maximum Likelihood), específicament el Branch analysis.
- Per tal de realitzar la comparació específica entre Homo sapiens i Pan troglodytes: hem fet un Blast de la seqüència d'un i altre per veure si coincidien, hem analitzat l'apartat del Tree View que apareixia a la pàgina on es veia l'evoluació de totes les variants de proteínes. Hem buscat informació referent a homologia de les proteïnes estudiades al NCBI per l'espècie Pan troglodytes, hem realitzat un alineament amb el clustalw de les seqüències de les dues espècies.
Per tal de saber en quins teixits o cèl·lules s'expresaven els gens, hem utilitzat la informació cedida per la base de dades del USCS on es mostra l'experssió en microarrays. A més a més, hem utilitzat el VisiGene, però els resultats no han sigut satisfactoris ja que gairebé no hi havia cap imatge. Per últim hem buscat informació al Gene Sorter, però tampoc contenia dades de les proteïna Kua i UEV (UBE2V1), tansols n'hem trobat de la proteïna de fusió.
- Per obtenir les regions promotores dels gens estudiats, des del genome browser hem anat a la informació que ofereix RefSeq clicant a sobre del gen, des d'allà hem clicat al link per obtenir la seqüència genòmica. Hem variat les opcions que hi havia: només s'ha seleccionat "Promoter by 1000 bases" i "Downstream by 100 bases". Així hem pogut obtenir la seqüència promotora en format fasta, que ens servirà posteriorment per executar el PROMO i el programa que nosaltres hem fet.
- Per obtenir els possibles factors de transcripció que s'uneixen a la seqüència promotora hem utilitzat el programa PROMO. Hem seleccionat l'espècie Homo sapiens i hem introduït la seqüència promotora. Hem obtingut una llarga llista de resultats, la hem passat a una taula i l'hem ordenada en ordre ascendent d'acord amb els valors de RE equally inicialment, i tot seguit segons els valors de dissimilarity. Hem considerat que el llindar d'acceptació per a ser un factor de transcripció que s'unís a la seqüència promotora i pogués regular l'expressió del gen sigués de 0.09.
- Hem realitzat un programa en Perl per tal de que ens llegís i analitzés la unió de possibles factors de transcripció a la regió promotora i ens calculés el p-value final. Amb les dades obtingudes s'han realitzat una sèrie de taules, per poder analitzar-ne els resultats.
- Finalment, s'han comparat els resultats obtinguts amb el PROMO i amb el programa realitzat per nosaltres.
Per estudiar la funció dels gens hem utilitzat la informació del gene ontology (GO) per saber en quins processos estaben implicats, i hem vist que es tractava de processos generals: ubiquitinació, regulació del cicle cel·lular, regulació de la diferenciació... En ser informació de caràcter general, hem buscat a varis papers la funció específica de les proteïnes que estudiem.
| -Inici- |
A mode de conclusió, a continuació se sintetitza la informació que hem trobat de Kua-UEV i de les dues proteïnes que la constitueixen.
Kua-UEV és una proteïna híbrida que es codifica a partir de la fusió dels gens de les proteïnes que la formen: el gen Kua que genera la proteïna independent Kua i el gen UBE2V1 que genera la proteïna independent UEV1.
El gen Kua s'ubica a la regió cromosòmica 20q13.13, entre les bases 48,173,717 i 48,203,667. La proteïna Kua que es transcriu a partir d'aquest gen té 29951 nt, organitzats en 6 exons que generen una seqüència aminoacídica de 270 aminoàcids.
Per altra banda, el gen UBE2V1 s'ubica a la regió cromosòmica 20q13.12, entre les bases 48,163,117 i 48,132,712. Codifica per la proteïna UEV1, que presenta 4 exons i està formada per 147 aminoàcids.
La proteïna híbrida es crea quan el gen Kua, que es localitza en la regió més 5', es comença a transcriure, però aquesta transcripció no cesa quan acaba el gen Kua sino que es continua passant a mRNA el següent gen (UBE2V1). D'aquesta manera, per splicing alternatiu es codifica UEV en el mateix producte que Kua, generant la proteïna de fusió.
Kua té 1 transcrit que genera una sola isoforma, mentre que UEV té 4 transcrits que poden generar fins a 3 isoformes diferents, tenint en compte que 2 transcrits generen la mateixa isoforma. Kua-UEV té 2 transcrits possibles que generen 2 isoformes diferents.
Pel què fa a conservació d'aquestes proteïnes en altres espècies diferents de l'humà, hem vist que tant UEV com Kua presenten un percentatge d'identitat força elevat en la majoria d'espècies on se'ls ha trobat una proteïna ortòloga. Però hem vist certes excepcions, com per exemple en el cas de UEV en Pan troglodytes. Kua-UEV també presenta gran conservació al llarg de l'evolució tant a nivell intraespecífic com entre diferents espècies, fet que demostra que segurament té importància biològica.
En quant a expressió de Kua i UEV en teixits, hem vist que UEV presenta una expressió més global i generalitzada que Kua, tot i que coincideixen en alguns llocs com per exemple en teixits relacionats amb el sistema immune. Estudiant també l'expressió de Kua-UEV, hem vist que aquesta es localitza en teixits que són més específics de Kua que no pas de UEV. Per tant podriem dir que Kua és capaç de delimitar els teixits d'expressió de la proteïna de fusió.
En l'estudi de la caracterització de regions promotores, realitzat a partir de dos mètodes diferents (mitjançant PROMO i programa en llenguatge Perl) s'han mostrat aquells factors de transcripció capaços d'induïr l'expressió de Kua i UEV com a proteïnes independents. Els factors que activen l'expressió de la proteïna de fusió coincideixen amb els que reconeixen millor el gen Kua, conclusió òbvia si pensem que en la proteïna de fusió la primera regió coincideix amb l'inici de la seqüència de Kua. El factor de transcripció més eficient per induïr l'expressió de Kua (i per tant també de la proteïna de fusió) és el NRSF, i en segon lloc AR i AP1. El factor més eficient per activar la transcripció del gen UBE2V1 és sobretot el NF-AT1.
Finalment, en relació a la funció de la proteïna de fusió, hem cercat informació en diversos articles relacionats i podem concloure que, tot i que Kua sigui una proteïna recentment descoberta i encara poc estudiada, té la capacitat de modificar la localització subcel·lular del híbrid proteic, translocant-lo del nucli (on se situa UEV) a estructures citoplasmàtiques. Actualment, la funció biològica de Kua-UEV no està exactament definida, tot i que es creu que participa en processos de poliubiquitinació.
| -Inici- |
Bases de dades i software:
NCBI: http://www.ncbi.nih.gov/
UCSC: http://genome.ucsc.edu/
RefSeq: http://www.ncbi.nlm.nih.gov/RefSeq/index.html
Clustalw: http://www.ebi.ac.uk/clustalw/
Ensembl: http://www.ensembl.org/
Uniprot: www.ebi.uniprot.org/index.shtml/
Gene ontology: http://www.geneontology.org
Pubmed: http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?db=PubMed
BLAST: http://www.ncbi.nlm.nih.gov/BLAST/
PROMO: http://alggen.lsi.upc.es/cgi-bin/promo_v3/promo/promoinit.cgi?dirDB=TF_8.3
RevTrans: http://www.cbs.dtu.dk/services/RevTrans/
| -Inici- |
Març 2007