Estructura genómica
Como se ha especificado anteriormente, la proteína en la que basamos este trabajo, resulta de la fusión del gen ALK1 y el gen NPM1.
El gen ALK1 se encuentra localizado en el cromosoma 2 brazo p 23.2.

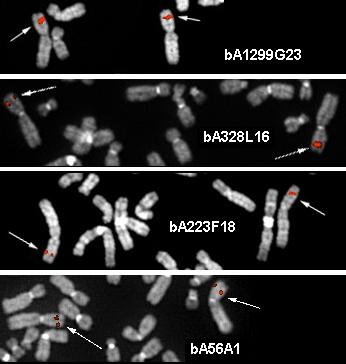
Figuras 1 y 2: Localización cromosómica del gen ALK.1,2
Este gen consta de
30 exones.
No observamos evidencias de exones no codificantes (UTR) en la traducción de esta proteína en la base de datos de Ensembl. Pero en cambio, si analizamos este gen en la base de datos UCSC encontramos que el gen tiene 907 nucleótidos upstream que forman parte de un UTR y 448 nucleótidos downstream que forman parte de un exón no codificante.
Consta de dos tránscritos, uno de ellos con 30 exones y el otro de 29 exones. En las bases de datos encontramos que esta proteína tiene 3 tránscritos pero realizando un alineamiento entre éstos observamos que dos de ellos tienen un 100% de identidad por lo que concluímos que debe tratarse del mismo tránscrito.
La regulación por splicing alternativo del exón 9 comporta su desaparición. Este proceso se debe a que los cinco nucleótidos que forman éste exón son iguales a los 5 primeros nucleótidos del intrón 8-9, de esta forma se produce un splicing que englobaría estos nucleótidos intrónicos en el final del exón 8 y el exón 9 pasaría a formar parte del intrón 8-9. De esta forma se mantiene la pauta de lectura aunque obtenemos una proteína con un exón menos pero con el mismo número de aminoácidos en la proteína traducida final. Existe un cambio motivado por este splicing altenativo que convella a la presencia de una cisteína en la posición 548 en vez de una serina. Esto se muestra de manera gráfica en el siguiente link.
La parte de la proteína que se fusiona con NPM1 consta de 10 exones que se corresponden con la parte final de ALK1. Esta región no se ve afectada por el splicing por lo tanto pensamos que ambos tránscritos podrían formar parte de la proteína de fusión.
En la siguiente figura podemos ver una representación exónica de los diferentes tránscritos.
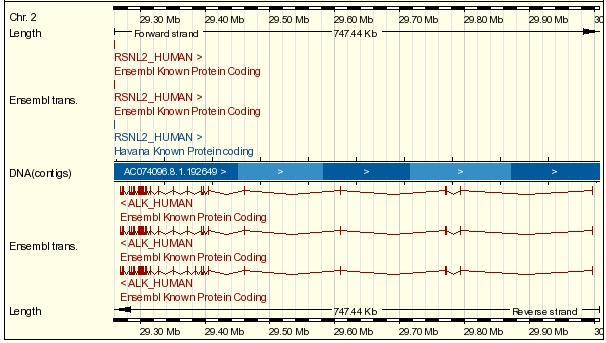
Figura 3: Estructura genómica de los tránscritos del gen ALK1.3
El gen NPM1 se encuentra localizado en el cromosoma 5 en el brazo q35.1.
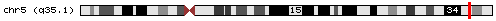
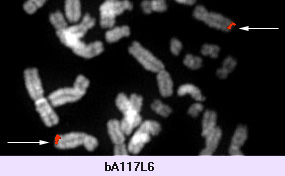
Figuras 4 y 5: Localización cromosómica del gen NPM.1,4
Este gen consta de 12 exones. De estos 12 exones, el primero no es codificante conteniendo 119 nucleótidos. El segundo exón contiene una secuencia no codificante a su inicio de 16 nucleótidos y a partir de éstos comienza la secuencia codificante. También existe una región no codificante en el último exón que abarca los últimos 318 nucleótidos de la secuencia.
Este gen contiene dos tránscritos, uno de 12 exones y el otro de 11 que se dan por la pérdida del exón 9 por splicing alternativo. La diferencia entre las isoformas encontradas en cada tránscrito se dan en una región codificante, hecho que provoca que la proteína sea de menor tamaño. Se mantiene la pauta de lectura, la parte anterior a este exón y la parte posterior son idénticas en ambas proteínas, pero sin embargo en una aparece el exón 9 codificante y en la otra no. Toda esta información proviene de la base de datos de Ensembl pero hemos contrastado estos datos con la información que aparece en UCSC. En esta base nos aparecen tres isoformas para esta proteína. Mirando las correspondencias con RefSeq, comprobamos que dos de estas tres isoformas son exactamente iguales a los tránscritos encontrados en Ensembl. En este trabajo nos basaremos en Ensembl ya que nos parece más fiable la información proporcionada para este gen.
La parte de la proteína que se fusiona con ALK1 alcanza los cinco primeros exones de ésta, siendo el primero UTR (no codificante).
En la siguiente figura (Figura 2) podemos ver una representación exónica de los diferentes tránscritos.
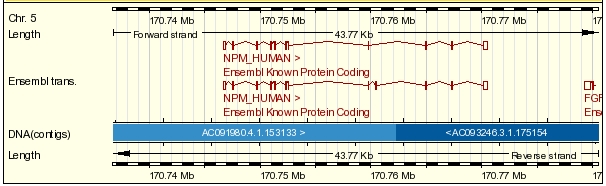
Figura 6: Estructura genómica de los tránscritos del gen NPM1.3
Conservación en otras especies
Para estudiar la conservación de las proteínas ALK1 y NPM1 en las diferentes especies nos hemos basado en los datos proporcionados por Ensembl. Hemos contrastado el porcentaje de identidad mediante BIOMART obteniendo los mismo restultado que los observados en Ensembl.
El gen ALK1 se encuentra en 25 especies además de en humano. En la siguiente tabla podemos ver un resumen de las diferentes especies donde se ecuentra, el identificador de Ensembl correspondiente para cada ortólogo y el porcentaje de identidad de cada especie con Homo sapiens. De las 25 especies encontradas hemos hecho una selección de aquellas que tienen un porcentaje de homología superior a 60%.
| Especie ortóloga | Identificador Ensembl | Porcentaje de identidad | Imagen |
| Macaca Mulatta | ENSMMUG00000010265 | 97 |  |
| Rattus norvegicus | ENSRNOG00000008683 | 88 |  |
| Pan troglodytes | ENSPTRG00000011796 | 87 |  |
| Mus musculus | ENSMUSG00000055471 | 87 |  |
| Felis catus | ENSFCAG00000001792 | 83 |  |
| Bos taurus | ENSBTAG00000007379 | 77 |  |
| Tupaia belangeri | ENSTBEG00000016366 | 74 |  |
| Canis familiaris | ENSCAFG00000005297 | 70 | |
| Dasypus novemcinctus | ENSDNOG00000007522 | 68 |  |
| Loxodonta africana | ENSLAFG00000014602 | 66 |  |
| Echinops telfairi | ENSETEG00000007715 | 65 |  |
| Oryctolagus cuniculus | ENSOCUG00000001085 | 63 |  |
| Monodelphis domestica | ENSMODG00000015531 | 62 |  |
Tabla 1: Especies ortólogas para ALK 1.3
Esta tabla está ordenada de mayor a menor porcentaje de homologia, siendo la primera especie en aparecer Macaca mulatta seguido de Rattus norvegicus.
También hemos realizado un árbol filogenético del gen ALK1 en las diferentes especies en las que se encuentra. En el árbol aparece Pan troglodytes como especie más cercana a Homo sapiens, siendo Macaca mulatta la segunda formando el grupo de primates y el grupo formado por los roedores es el más cercano a ellos. Dentro de esta rama debería estar incluido Monodelphis domestica ya que se trata de un roedor pero creemos que no está bien secuenciado ya que aparece muy alejado del grupo de roedores.
Para el gen NPM 1 no hemos encontrado especies ortólogas en Ensembl. Para verificar estos resultados, intentamos buscar en BIOMART secuencias ortólogas en las diferentes especies mediante el identificador de Ensembl sin obtener resultado alguno. Realizamos un BlastP en NCBI utilizando la secuencia proteica de NPM y obtuvimos secuencias homólogas con un e-value siempre mayor de 0. Las que tenían un e-value menor correspondían a proteínas humanas y la mayoría de las que pertenecían a otras especie resultaban ser hipotéticas por lo que decidimos descartarlas.
Caracterización de la expresión
El gen ALK1 se expresa principalmente en el cerebro y el sistema nervioso central. También lo podemos encontrar en el intestino delgado y testículos. Normalmente no se expresa en células linfoides. En los siguientes links podéis encontrar la expresión tisular de este gen, en rojo aparecen los tejidos donde se presenta mayor expresión de este gen y en verde aquellos donde hay menos expresión. En negro encontramos los tejido donde no hay expresión de este gen:
- GNF Expression Atlas 2 Human Data
- GNF Expression Atlas 1 Human Data
- Normal Human Tissue cDNA Microarrays
El gen NPM1 se encuentra expresado en gran parte de los tejidos. Esta proteína se asocia con ribonucleoproteínas y une ssDNA, hecho que podría justificar su gran extensión tisular. En los siguientes links podéis encontrar la expresión tisular de este gen.
- GNF Expression Atlas 2 Human Data
- GNF Expression Atlas 1 Human Data
- Normal Human Tissue cDNA Microarrays
Caracterización de las regiones promotoras
Para poder determinar los factores de transcripción capaces de unirse a las regiones promotoras de nuestra proteína de fusión hemos desarrollado un programa en Perl capaz de leer las diferentes secuencias promotoras y determinar posibles lugares de unión a diferentes factores de transcripción que nos han sido proporcionados en forma de matriz. Mediante este programa obtenemos la región donde cada factor es capaz de unirse así como el score máximo obtenido. También obtenemos el valor de p-value para esta posición que nos permitirá determinar, junto con el score, cuales son los factores que se unen de manera específica a la secuencia.
Por otro lado, hemos analizado las secuencias promotoras de cada una de las dos proteínas mediante el programa PROMO obteniendo una lista de los posibles factores que pueden unirse a las secuencias promotoras. Para la obtención de los factores hemos aplicado un 5% de disimilaridad como filtro para obtener únicamente aquellos que presentan una similitud del 95%. De todos los obtenidos, escogimos únicamente aquellos que presentaban un E-value inferior al 0.09 ya que son los que seguramente se unirán a la secuencia promotora.
- Regiones promotoras de ALK 1
- Regiones promotoras de NPM 1
Esta tabla contiene los resultados obtenidos del análisis de la región promotora del gen ALK mediante el programa en lenguaje Perl que hemos creado. Las mejores predicciones son aquellas que tienen un score muy alto y un p-value bajo. Si miramos la tabla podemos ver como el único factor que cumple estos requisitos es AR. Observamos que el factor YY1 tiene un score alto que nos podría indicar que sería un buen candidato a unirse a la secuencia pero observamos que su p-value es también muy alto. Esto es debido a que la secuencia reconocida por este factor tan solo está formada por 4 nucleótidos, al ser tan corta es más fácil que reconozca secuencias dentro de las aleatorias que hemos creado.
Estos valores de p-value no siempre serán los mismos ya que este valor se calcula en función de las secuencias aleatorias creadas cada vez que ejecutamos el programa.
| Factor de transcripción | Posición | Score | P-value |
| AP-1 | 111 | -995,4928 | 0.75 |
| AR | 733 | 3,648668182 | 0.11 |
| C-Myc | 107 | -995,934 | 0.84 |
| NF-AT1 | 78 | 3,094534 | 0.56 |
| NK-kappaB | 477 | 3,094534 | 0.93 |
| SRF | 38 | 3,094534 | 0.53 |
| YY1 | 429 | 3,094534 | 1 |
| RXR-alpha | 15 | 3,094534 | 0.26 |
| HIF-1 | 956 | -995,5313 | 0.45 |
| AhR | 864 | 3,0457 | 0.33 |
| PU.1 | 407 | 2,6779 | 0.56 |
| HNF-4 | 205 | -995,6188 | 0.46 |
| NRSF | 874 | -995,86419 | 0.41 |
Tabla 2: Factores de transcripción capaces de unirse a la región promotora de ALK 1 encontrados mediante programa Perl.
En la siguiente tabla se presentan los factores de transcripción obtenidos mediante el programa PROMO. Podemos ver que los que tienen más probabilidad de unirse a la secuencia promotora son PU.1 y Sp3. Estos dos factores presentan un score que se consideraría alto y un p-value muy bajo. Ninguno de los dos coincide con los predichos mediante el programa realizado aunque PU.1 sí era uno de los escogidos en el análisis hecho por el programa.
| Factor name | Start position | End position | Dissimilarity | String | RE equally | RE query |
| c-Ets-2 [T00113] | 513 | 521 | 1.644150 | TTCCTCCTC | 0.02518 | 0.02489 |
| c-Ets-2 [T00113] | 73 | 81 | 4.017001 | CGAAAGGAA | 0.08812 | 0.06443 |
| Elk-1 [T00250] | 512 | 520 | 0.134348 | CTTCCTCCT | 0.03357 | 0.03974 |
| GCF [T00320] | 927 | 935 | 1.269230 | TCCCTGCGC | 0.01678 | 0.03057 |
| HOXD10 [T01425] | 555 | 564 | 0.954221 | CAGTTTTATT | 0.00839 | 0.00355 |
| HOXD9 [T01424] | 555 | 564 | 0.954221 | CAGTTTTATT | 0.00839 | 0.00355 |
| IRF-1 [T00423] | 74 | 82 | 3.692688 | GAAAGGAAA | 0.03777 | 0.03058 |
| Pax-5 [T00070] | 849 | 855 | 3.075094 | GGGCGAG | 0.06714 | 0.11046 |
| POU2F1 [T00641] | 794 | 804 | 0.871129 | ATTTGCATAGG | 0.00761 | 0.00473 |
| PU.1 [T02068] | 509 | 521 | 3.723988 | CCACTTCCTCCTC | 0.00095 | 0.00106 |
| RAR-beta [T00721] | 132 | 141 | 3.226064 | TCTGAACCCT | 0.06714 | 0.07315 |
| Sp1 [T00759] | 1077 | 1086 | 3.984471 | CGGCCGCCCT | 0.04930 | 0.08898 |
| Sp1 [T00759] | 748 | 757 | 1.253855 | GGGGCGGGCT | 0.01783 | 0.03826 |
| Sp1 [T00759] | 916 | 925 | 0.000000 | GCCCCGCCCC | 0.00105 | 0.00291 |
| Sp1 [T00759] | 900 | 909 | 0.000000 | GCCCCGCCCC | 0.00105 | 0.00291 |
| Sp1 [T00759] | 843 | 852 | 0.000000 | GGGGCGGGGC | 0.00105 | 0.00291 |
| Sp1 [T00759] | 1001 | 1010 | 3.623596 | GGGGCGGCAG | 0.04091 | 0.07961 |
| Sp3 [T02338] | 912 | 927 | 1.662071 | CCGGGCCCCGCCCCCT | 0.00001 | 0.00003 |
| TBP [T00794] | 23 | 32 | 2.807313 | TTGTTATAAA | 0.06714 | 0.03398 |
Tabla 3: Factores de transcripción capaces de unirse a la región promotora de ALK 1 encontrados mediante PROMO.
Si aumentamos la disimilitud a un 15% en PROMO vemos la aparición de varios factores de transcripción que sí se encuentran en las matrices utilizadas para el análisis de los promotores en nuestro programa, como son NF-AT1, RxR-alpha, YY1 y PU.1. De todos ellos hemos visto únicamente una coincidencia entre ambos análisis. La primera secuencia encontrada por PROMO para el factor NF-AT1 es la misma que la predicha por el programa en Perl.
Esta tabla contiene los resultados obtenidos del análisis de la región promotora del gen NPM mediante el programa. Si miramos la tabla podemos ver como los factores que tienen un score alto y un p-value bajo son AhR y PU.1. Podemos ver que el factor YY1 tiene un score alto que nos podría indicar que sería un buen candidato a unirse a la secuencia pero observamos que su p-value es también muy alto.Esto es debido a que la secuencia reconocida por este factor tan solo está formada por 4 nucleótidos, al ser tan corta es más fácil que reconozca secuencias dentro de las aleatorias que hemos creado.
Estos valores de p-value no siempre serán los mismos ya que este valor se calcula en función de las secuencias aleatorias creadas cada vez que ejecutamos el programa.
| Factor de transcripción | Posición | Score | P-value |
| AP-1 | 287 | 2,65075 | 0.45 |
| AR | 513 | 2,1729 | 0.78 |
| C-Myc | 760 | -995,934 | 0.87 |
| NF-AT1 | 617 | 3,163 | 0.55 |
| NK-kappaB | 562 | -996,1161 | 0.78 |
| SRF | 336 | -994,62484 | 0.25 |
| YY1 | 613 | 2,46511 | 0.93 |
| RXR-alpha | 512 | -996,62484 | 0.31 |
| HIF-1 | 551 | 3,3225622 | 0.8 |
| AhR | 809 | 3,32256 | 0.17 |
| PU.1 | 259 | 3,33859122 | 0.08 |
| HNF-4 | 512 | -995,3981 | 0.46 |
| NRSF | 823 | -996,37728199 | 0.44 |
Tabla 4: Factores de transcripción capaces de unirse a la región promotora de NPM encontrados mediante programa Perl.
En la siguiente tabla se presentan los factores de transcripción obtenidos mediante el programa PROMO. Podemos ver que el que tiene más probabilidad de unirse a la secuencia promotora es FOXO4. Este factor presenta un score que se consideraría alto y un p-value muy bajo. Dentro de los obtenidos por PROMO encontramos algunos de los que analizamos mediante el programa pero no dan resultados relevantes.
| Factor name | Start position | End position | Dissimilarity | String | RE equally | RE query |
| AhR:Arnt [T05394] | 805 | 814 | 3,888628 | GCACGCGTGC | 0,01259 | 0,01889 |
| AhR:Arnt [T05394] | 977 | 986 | 2,810335 | GCACGCGCGC | 0,00944 | 0,01787 |
| AR [T00040] | 416 | 424 | 4,241082 | GGACAGAGC | 0,03777 | 0,0376 |
| c-Ets-2 [T00113] | 256 | 264 | 1,64415 | CAGGAGGAA | 0,02518 | 0,0283 |
| c-Ets-2 [T00113] | 466 | 474 | 4,589988 | TTCCTTCCT | 0,03777 | 0,03698 |
| c-Ets-2 [T00113] | 32 | 40 | 4,017001 | TTCCTTTCT | 0,08812 | 0,0681 |
| c-Myb [T00137] | 531 | 538 | 4,270092 | TAACTGCG | 0,03357 | 0,03325 |
| E2F-1 [T01542] | 991 | 998 | 4,545253 | GCGGGACT | 0,08392 | 0,11009 |
| E2F-1 [T01542] | 751 | 758 | 1,490375 | GCGGGAGA | 0,03357 | 0,04543 |
| Elk-1 [T00250] | 469 | 477 | 4,892803 | CTTCCTAAC | 0,0042 | 0,00436 |
| Elk-1 [T00250] | 31 | 39 | 2,987643 | CTTCCTTTC | 0,04196 | 0,03988 |
| Elk-1 [T00250] | 408 | 416 | 3,381796 | CTCAGGAAG | 0,02518 | 0,0245 |
| FOXO4 [T03403] | 69 | 82 | 3,616253 | TAAATTTGTTTGAT | 0,00054 | 0,00029 |
| GATA-2 [T00308] | 384 | 392 | 1,111111 | AGATAAGGA | 0,05035 | 0,04597 |
| GCF [T00320] | 1037 | 1045 | 0 | GCGCGGGGA | 0,05035 | 0,08469 |
| GCF [T00320] | 736 | 744 | 0 | GGCCGGCGC | 0,05035 | 0,08469 |
| GCF [T00320] | 1074 | 1082 | 2,339499 | GTCCTGCGC | 0,03357 | 0,0478 |
| GCF [T00320] | 937 | 945 | 2,339499 | GCGCAGGAC | 0,03357 | 0,0478 |
| HNF-1C [T01951] | 266 | 274 | 1,940349 | GTTAAAGAT | 0,01259 | 0,00755 |
| IRF-1 [T00423] | 1056 | 1064 | 3,689552 | TTTCCCTGG | 0,03777 | 0,03155 |
| IRF-1 [T00423] | 613 | 621 | 4,968836 | ATGGGGAAA | 0,04196 | 0,03896 |
| NF-AT1 [T01948] | 799 | 808 | 4,823485 | TGGAAAGCAC | 0,04196 | 0,0361 |
| p53 [T00671] | 601 | 607 | 4,786849 | GGGCACT | 0,06714 | 0,08469 |
| Pax-5 [T00070] | 901 | 907 | 3,075094 | GGGCGAG | 0,06714 | 0,11263 |
| PXR-1:RXR-alpha [T05671] | 512 | 519 | 0,11263 | TGAACTTT | 0,03357 | 0,02256 |
| RXR-alpha [T01345] | 921 | 927 | 1,87833 | GGGTTGA | 0,06714 | 0,0864 |
| SRY [T00997] | 473 | 481 | 0,999172 | CTAACAAAG | 0,03357 | 0,02469 |
| SRY [T00997] | 428 | 436 | 0,999172 | AAAACAAAG | 0,03357 | 0,02469 |
| TFIID [T00820] | 701 | 707 | 3,075094 | TTTTGCA | 0,06714 | 0,04943 |
Tabla 5: Factores de transcripción capaces de unirse a la región promotora de NPM encontrados mediante PROMO.
Si aumentamos la disimilitud a un 15% en PROMO vemos la aparición de los factores de transcripción NF-AT1, RxR-alpha, YY1, AR y AP-1. De todos ellos hemos visto únicamente una coincidencia entre ambos análisis. La segunda secuencia encontrada por PROMO para el factor YY1 es la misma que la predicha por el programa en Perl.
Tenemos que destacar que las matrices que nos proporcionaron para desarrollar el programa contienen datos diferentes a las utilizadas por PROMO. Ésto podría explicar las diferencias entre ambas predicciones.
Función del gen
 El producto del gen ALK es un receptor transmembrana de señalización con actividad tirosina kinasa. Está implicado en el desarrollo del sistema nervioso, desde su formación hasta la maduración de su estructura. También se ha visto que está implicado en el desarrollo del mesodermo intestinal. Actualmente no se conoce nada sobre el ligando de ALK pero se cree que podría ser un factor soluble o una proteína anclada a membrana, incluso el receptor ALK podría actuar como una molécula de reconocimiento celular permitiendo la interacción entre células. Es posible que ALK esté involucrada en la interacción neurona-neurona o neurona-glía. Ésto junto al hecho de que la expresión de ALK es más alta en estados neonatales indica que esta interacción puede estar implicada en la formación de la red neuronal o en el crecimiento del cono axonal.5
El producto del gen ALK es un receptor transmembrana de señalización con actividad tirosina kinasa. Está implicado en el desarrollo del sistema nervioso, desde su formación hasta la maduración de su estructura. También se ha visto que está implicado en el desarrollo del mesodermo intestinal. Actualmente no se conoce nada sobre el ligando de ALK pero se cree que podría ser un factor soluble o una proteína anclada a membrana, incluso el receptor ALK podría actuar como una molécula de reconocimiento celular permitiendo la interacción entre células. Es posible que ALK esté involucrada en la interacción neurona-neurona o neurona-glía. Ésto junto al hecho de que la expresión de ALK es más alta en estados neonatales indica que esta interacción puede estar implicada en la formación de la red neuronal o en el crecimiento del cono axonal.5
 El producto del gen NPM es una fosfoproteína nucleolar que se encuentra continuamente viajando entre el núcleo y el citoplasma. Está involucrado en la activación del factor de transcripción NF-KappaB6, ensamblaje y el transporte ribosomal7, estabilidad y la transcripción de p537, involucrado en el transporte de proteínas7, down-regulation de la proliferación celular7, transporte nucleocitoplasmático7, respuesta al estrés7, se encuentra regulada por CDK2/Cyclin E en la duplicación del centrosoma8, transducción de señales9 y también actuaría como chaperona molecular previniendo la agregación de proteínas en los alrededores del núcleo.
El producto del gen NPM es una fosfoproteína nucleolar que se encuentra continuamente viajando entre el núcleo y el citoplasma. Está involucrado en la activación del factor de transcripción NF-KappaB6, ensamblaje y el transporte ribosomal7, estabilidad y la transcripción de p537, involucrado en el transporte de proteínas7, down-regulation de la proliferación celular7, transporte nucleocitoplasmático7, respuesta al estrés7, se encuentra regulada por CDK2/Cyclin E en la duplicación del centrosoma8, transducción de señales9 y también actuaría como chaperona molecular previniendo la agregación de proteínas en los alrededores del núcleo.
El producto de la translocación cromosómica t(2;5)(p23;q35) genera un híbrido que contiene un dominio intracelular perteneciente a ALK1 con actividad tirosina kinasa yuxtapuesta a NPM. Esta proteína de fusión tiene constitutivamente activada la actividad tirosin-kinasa y es capaz de transformar células hematopoyéticas en tumorales produciendo entre un 50 y 60% de los casos de ALCL (anaplastic large-cell lymphomas). Éstos son un tipo de linfomas no-Hodgkin10. La vía por la que esta proteína quimérica genera transformación de las células en tumorales solo se entiendo parcialmente. Se ha visto que esta activación constitutiva produce una activación de la vía de señalización MEK/ERK implicada en procesos de proliferación, diferenciación, supervivencia, migración y división celular11.